Viaggio nel Cuore delle Cellule Staminali: Svelati i Segreti di ESC e ffEPSC
Ciao a tutti! Oggi voglio portarvi con me in un viaggio affascinante nel mondo microscopico delle cellule staminali, un universo pieno di potenziale e, come abbiamo scoperto, di sorprendente diversità. Parleremo di cellule staminali embrionali umane (le famose ESC) e di un tipo più “giovane” e versatile, le cellule staminali pluripotenti estese feeder-free (ffEPSC). Preparatevi, perché stiamo per usare una tecnologia pazzesca per spiare cosa succede dentro queste cellule, una per una!
Le Protagoniste: ESC vs ffEPSC
Immaginate le cellule staminali come dei veri e propri jolly biologici. Le ESC sono note da tempo: possono trasformarsi in quasi tutti i tipi di cellule del nostro corpo, ma rappresentano uno stadio di sviluppo che assomiglia a quello dell’embrione dopo l’impianto nell’utero. Gli scienziati lo chiamano stato “primed“.
Poi ci siamo noi (e altri gruppi di ricerca, ovviamente!) che abbiamo lavorato per ottenere cellule ancora più “indietro” nel tempo dello sviluppo, più vicine ai primissimi stadi embrionali. Le ffEPSC sono proprio questo: cellule in uno stato di “pluripotenza estesa“, capaci non solo di formare l’embrione stesso, ma potenzialmente anche i tessuti extra-embrionali (come parte della placenta). Capire come si passa da uno stato “primed” (ESC) a uno “esteso” (ffEPSC) è fondamentale, sia per la ricerca di base sullo sviluppo, sia per future applicazioni in medicina rigenerativa. Ma c’è un “ma”: queste popolazioni di cellule non sono tutte uguali al loro interno. C’è un sacco di eterogeneità, una sorta di “personalità” diversa da cellula a cellula, che complica un po’ le cose.
La Lente d’Ingrandimento: Smart-seq2 Single-Cell RNA-seq
Come fare a capire questa eterogeneità? Abbiamo usato una tecnica potentissima chiamata single-cell RNA sequencing (scRNA-seq), in particolare una versione molto sensibile e dettagliata chiamata Smart-seq2. In pratica, è come se avessimo preso singole cellule ESC e ffEPSC e avessimo letto l’intero “libretto di istruzioni” attivo in quel momento (l’RNA messaggero) per ognuna di esse. Questo ci dà una fotografia ad altissima risoluzione di cosa sta facendo ogni cellula individualmente.
Abbiamo seguito un protocollo preciso: prima abbiamo coltivato le nostre ESC (la linea H9, per i più tecnici) e le abbiamo “riprogrammate” per diventare ffEPSC usando un cocktail chimico specifico (chiamato LCDM-IY). Poi, abbiamo isolato le singole cellule, estratto il loro RNA e preparato le “librerie” per il sequenziamento con Smart-seq2. Infine, abbiamo analizzato questa montagna di dati usando bioinformatica avanzata.
Cosa Abbiamo Visto: Gene Expression e Sorprese
La prima cosa che abbiamo fatto è stata confrontare i profili di espressione genica generali. Come ci aspettavamo, le ESC esprimevano alti livelli di geni associati alla pluripotenza “primed” e anche alcuni geni legati alle fasi iniziali della differenziazione. Le ffEPSC, invece, mostravano un’espressione più alta di geni legati all’attivazione del genoma zigotico (ZGA) – un evento chiave nelle primissime fasi dello sviluppo – e geni della pluripotenza “classica” o “naive“. Fin qui, tutto bene, i dati confermavano le identità delle cellule.
Ma l’analisi a singola cellula ha rivelato di più! Usando algoritmi di clustering (che raggruppano cellule simili), abbiamo visto che la popolazione di ESC non era omogenea. Anzi, sembrava più eterogenea delle ffEPSC, almeno guardando l’espressione dei geni “classici”. Abbiamo identificato due sottogruppi principali di ESC:
- ESC_C1: La maggioranza delle cellule, chiaramente nello stato “primed”, con geni come DNMT3B e THY1 molto attivi. Assomigliavano di più allo stadio di blastocisti tardiva dell’embrione.
- ESC_C2: Un gruppo più piccolo che sembrava trovarsi in uno stato leggermente precedente, con geni della pluripotenza naive (come TFAP2C) e legati alla ZGA (come YAP1) più espressi.
Questa scoperta sottolinea come anche una popolazione “standard” di ESC contenga cellule in stati leggermente diversi.
Il Ruolo Nascosto delle Sequenze Ripetute (e del Genoma T2T!)
Qui le cose si fanno ancora più interessanti. Oltre ai geni “normali”, il nostro genoma è pieno di sequenze di DNA ripetute, spesso considerate “DNA spazzatura”. Ma ricerche recenti (e anche la nostra!) stanno dimostrando che queste sequenze, specialmente quelle derivate da antichi retrovirus (chiamate elementi trasponibili o TE), giocano ruoli cruciali nello sviluppo embrionale.
Per analizzarle al meglio, abbiamo usato il nuovissimo genoma umano di riferimento completo, chiamato T2T (Telomere-to-Telomere), che mappa anche queste regioni ripetute molto meglio dei genomi precedenti. E cosa abbiamo trovato analizzando l’espressione di queste sequenze ripetute in ogni singola cellula?
Sorpresa! Mentre l’analisi genica mostrava più eterogeneità nelle ESC, l’analisi delle sequenze ripetute ha rivelato una maggiore eterogeneità nelle ffEPSC! Queste cellule si sono divise nettamente in due gruppi basandosi sull’espressione dei TE:
- ffEPSC_C1: Esprimeva TE tipici degli stadi di sviluppo un po’ più avanzati (morula e blastocisti tardiva) e più geni legati alla pluripotenza.
- ffEPSC_C2: Esprimeva TE caratteristici degli stadi precocissimi (zigote fino a 8 cellule) e più geni associati alla totipotenza (la capacità di formare *tutto*, anche i tessuti extra-embrionali).
Questo ci dice che le sequenze ripetute sono come delle “firme” molecolari che possono distinguere stati cellulari molto vicini tra loro, specialmente negli stadi più precoci rappresentati dalle ffEPSC. È affascinante vedere come queste sequenze, un tempo ignorate, siano così informative!
Ricostruire il Percorso: l’Analisi Pseudotemporale
Avendo dati da ESC e ffEPSC, che rappresentano un punto di partenza e uno di arrivo (o quasi), potevamo provare a ricostruire il “percorso” di transizione da uno stato all’altro. Abbiamo usato un’analisi chiamata pseudotempo, che ordina le cellule lungo una traiettoria virtuale basata sulla somiglianza della loro espressione genica.
Il risultato? Abbiamo ottenuto una traiettoria che andava dalle ESC più “primed” (ESC_C1), passava per le ESC leggermente precedenti (ESC_C2), arrivava alle ffEPSC più “pluripotenti” (ffEPSC_C1) e infine alle ffEPSC più “totipotenti” (ffEPSC_C2). Questo percorso virtuale assomigliava moltissimo a quello che succede realmente nello sviluppo embrionale umano dai primissimi stadi!
Abbiamo anche identificato geni chiave che cambiano lungo questa traiettoria. Ad esempio, il gene SUMO3 (coinvolto in una modifica proteica chiamata SUMOilazione) diminuiva progressivamente. È interessante perché si sa che inibire la SUMOilazione nei topi aiuta le cellule a tornare a uno stato simile a quello di 2 cellule! Altri geni, legati alla segnalazione BMP o alla mitosi (come E2F1, EXT1, EML1), invece aumentavano.
Mettere Tutto Insieme: Integrazione con Dati Embrionali
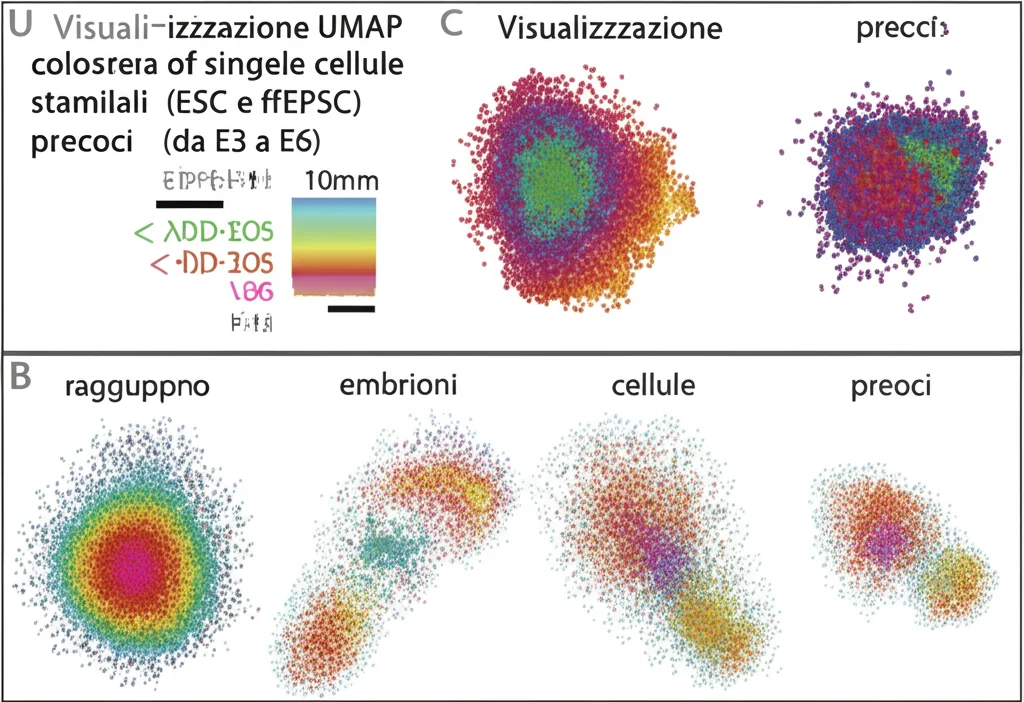
Per dare un contesto ancora più solido ai nostri risultati, abbiamo integrato i nostri dati con dataset pubblici di scRNA-seq da embrioni umani reali, coprendo lo sviluppo dallo zigote fino a 14 giorni. Quando abbiamo visualizzato tutte queste cellule insieme (le nostre ESC/ffEPSC e quelle degli embrioni), abbiamo visto che le nostre popolazioni si posizionavano esattamente dove ci aspettavamo:
- Le ffEPSC_C2 (quelle più “totipotenti” secondo i TE) si raggruppavano con le cellule embrionali degli stadi E3-E4 (morula).
- Le ffEPSC_C1 e le ESC si posizionavano intorno allo stadio E6 (blastocisti tardiva/inizio impianto).
- Le due sottopopolazioni di ESC si allineavano con gli stadi E5-E6 (blastocisti).
Questa integrazione conferma la rilevanza biologica delle nostre ffEPSC e delle sottopopolazioni che abbiamo identificato, collocandole precisamente nel panorama dello sviluppo embrionale precoce.
Conclusioni (Per Ora!)
Cosa ci portiamo a casa da questo studio? Beh, prima di tutto, abbiamo generato un dataset incredibilmente ricco e dettagliato (grazie a Smart-seq2!) che mappa le differenze tra ESC e ffEPSC a livello di singola cellula. Abbiamo confermato che le ffEPSC rappresentano uno stato di pluripotenza più precoce e abbiamo svelato un’eterogeneità inaspettata in entrambe le popolazioni.
Forse la cosa più eccitante è aver dimostrato l’importanza delle sequenze ripetute nel definire questi stati cellulari e la loro eterogeneità, specialmente usando il genoma T2T. Queste sequenze sono molto più che “spazzatura”! L’analisi pseudotemporale ci ha dato un’idea del percorso dinamico tra gli stati pluripotenti, collegandolo allo sviluppo reale.
Questo lavoro non solo approfondisce la nostra comprensione della pluripotenza e delle primissime fasi della vita umana, ma fornisce anche una risorsa preziosa per tutta la comunità scientifica che lavora sulle cellule staminali, sullo sviluppo e sulla medicina rigenerativa. C’è ancora tantissimo da scoprire, ma ogni passo come questo ci avvicina a comprendere (e magari un giorno a sfruttare appieno) l’incredibile potenziale di queste cellule.
Fonte: Springer