Cuore “Rigido”? Tutta Colpa di un Gene? Viaggio nella Variante TNNI3 c.406 C>T
Ciao a tutti! Oggi voglio portarvi con me in un viaggio affascinante nel mondo della genetica e delle malattie cardiache, un campo dove ogni scoperta può davvero fare la differenza. Parleremo di una condizione rara ma seria, la cardiomiopatia restrittiva (RCM), e di come una specifica “lettera” fuori posto nel nostro DNA possa scatenare tutto. Pronti a fare i detective genetici insieme a me?
Cos’è questa Cardiomiopatia Restrittiva (RCM)?
Immaginate il cuore come una pompa instancabile. Nella RCM, le pareti dei ventricoli (le camere inferiori del cuore) diventano rigide, quasi “testarde”. Questa rigidità impedisce al cuore di riempirsi correttamente di sangue tra un battito e l’altro (la famosa disfunzione diastolica). Meno sangue entra, meno sangue viene pompato in circolo. Il risultato? Sintomi tipici dello scompenso cardiaco come mancanza di respiro (dispnea), stanchezza cronica ed edemi (gonfiori).
Le cause possono essere diverse: malattie infiltrative, fibrosi, ma sempre più spesso scopriamo che dietro c’è una questione genetica. Varianti in geni specifici possono alterare le proteine che compongono il muscolo cardiaco, rendendolo meno elastico e funzionale. Ed è qui che entra in gioco il nostro protagonista di oggi: il gene TNNI3.
Il Gene TNNI3: Un Componente Chiave del Motore Cardiaco
Il gene TNNI3 (che fa parte della grande famiglia della Titina, una proteina gigantesca e fondamentale per l’elasticità e la contrazione muscolare) codifica per una proteina cruciale per il corretto funzionamento delle cellule del muscolo cardiaco. Pensatela come un ingranaggio essenziale nel complesso meccanismo che permette al nostro cuore di battere ritmicamente. Se questo gene subisce delle modifiche (varianti), la proteina prodotta potrebbe non funzionare a dovere, compromettendo l’intera struttura e funzione del cuore. E indovinate un po’? Varianti in TNNI3 sono state già associate a diverse forme di cardiomiopatia.
Il Caso Clinico: Una Lente sulla RCM Genetica
Ora vi racconto la storia che ha dato il via a questa specifica ricerca. Parliamo di una bambina, la primogenita di una famiglia sana, senza genitori consanguinei e con un fratellino senza problemi. Già a 4 anni, mostrava un certo ritardo nella crescita (alta 92 cm, pesava 12 kg). Nonostante l’intervento di un nutrizionista, la situazione non migliorava. A 6 anni, ha iniziato a manifestare una dispnea progressiva da sforzo, peggiorata negli ultimi 12 mesi.
Portata in ospedale, le è stato diagnosticato uno scompenso cardiaco. Indagini più approfondite in centri specializzati erano necessarie. Alla fine, presso il Rajaie Cardiovascular Institute di Teheran, un ecocardiogramma ha rivelato segni compatibili proprio con la RCM. Ed è qui che siamo entrati in gioco noi, con la consulenza genetica.

La Caccia alla Variante: Il Potere del WES
Per capire la base genetica del disturbo, abbiamo utilizzato una tecnica potentissima: il Whole Exome Sequencing (WES). Immaginatela come una sorta di “Google Maps” del DNA, che ci permette di leggere tutte le regioni codificanti dei geni (l’esoma) per scovare eventuali “errori di battitura” o varianti significative. Abbiamo analizzato il DNA della bambina estratto da un campione di sangue.
E bingo! Abbiamo trovato una variante “nuova”, mai descritta prima in relazione alla RCM, nel gene TNNI3: la c.406 C>T. La cosa interessante è che la bambina la possedeva in omozigosi, cioè aveva ereditato la stessa variante da entrambi i genitori. Per confermare, abbiamo usato il sequenziamento Sanger (una tecnica più “classica” ma precisa) sia sulla paziente che sui genitori. Risultato: i genitori erano portatori eterozigoti (avevano una copia normale e una copia con la variante), confermando una modalità di trasmissione autosomica recessiva. Questo significa che solo chi eredita entrambe le copie alterate del gene manifesta la malattia.
Decifrare il Messaggio: Cosa Fa Questa Variante?
Ok, abbiamo trovato la variante. Ma è davvero lei la colpevole? Qui entrano in campo gli strumenti bioinformatici e l’analisi strutturale.
- Analisi In Silico: Abbiamo usato diversi software predittivi (come MutationTaster, PolyPhen-2, SIFT, FATHMM, CADD). MutationTaster l’ha classificata come “disease-causing” (causante malattia). Il punteggio CADD era 14.02, indicando un impatto potenzialmente significativo.
- Linee Guida ACMG: Seguendo i criteri rigorosi dell’American College of Medical Genetics and Genomics, abbiamo classificato la variante come “likely pathogenic” (probabilmente patogenica).
- Impatto sulla Proteina: La variante c.406 C>T causa uno “stop” prematuro nella lettura del gene (tecnicamente, p.R136*). L’amminoacido Arginina (R) alla posizione 136 viene sostituito da un segnale di stop. Questo significa che la proteina TNNI3 prodotta è tronca, incompleta. Abbiamo verificato che questa posizione (136) è altamente conservata tra diverse specie, suggerendo la sua importanza funzionale.
- Modellazione Strutturale: Utilizzando software come SWISS-MODEL, abbiamo costruito un modello 3D della proteina. Questo ci ha mostrato come la versione troncata sia probabilmente incapace di ripiegarsi correttamente e di svolgere le sue funzioni essenziali, perdendo domini critici per la contrazione muscolare.
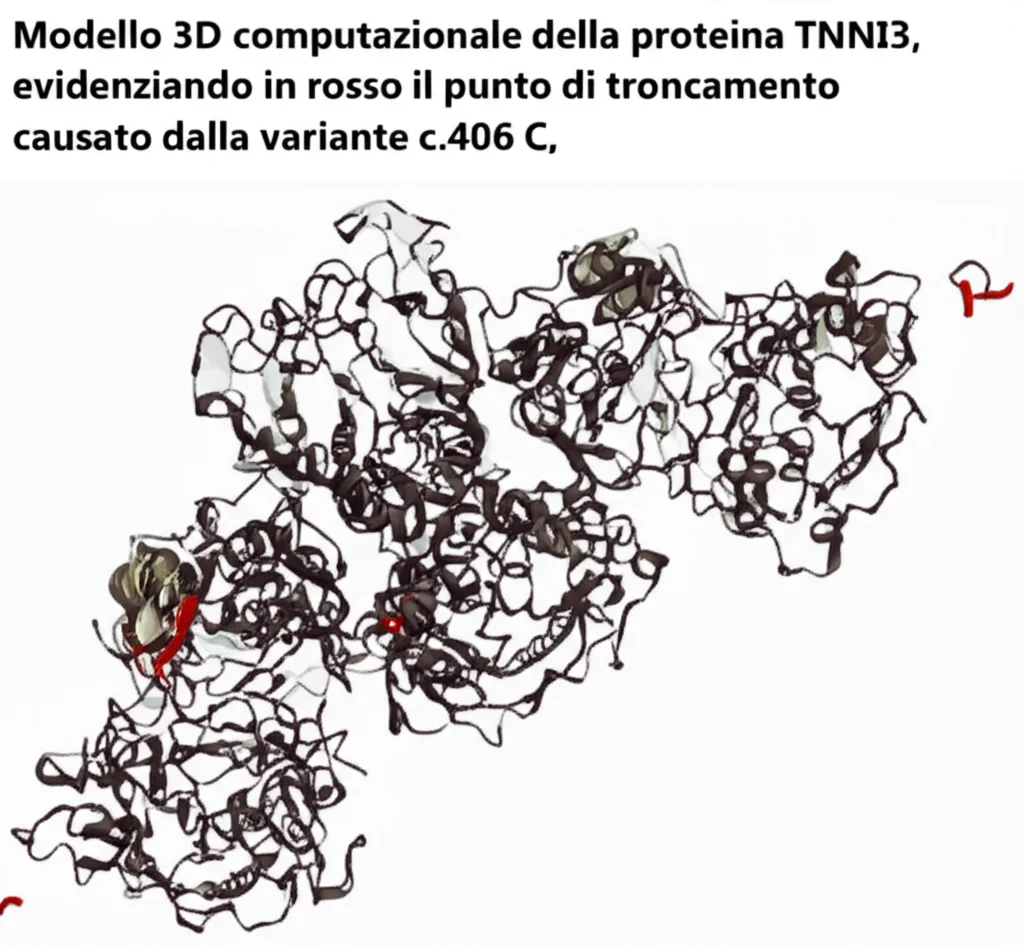 T (p.R136*), accanto a un modello della proteina completa in blu per confronto, sfondo scientifico astratto.” />
T (p.R136*), accanto a un modello della proteina completa in blu per confronto, sfondo scientifico astratto.” />
Collegare i Punti: Genetica, Clinica e Ricerca Futura
I risultati dell’ecocardiogramma della paziente erano perfettamente in linea con la RCM: funzione sistolica (la “spremitura” del cuore) preservata (FE 40%, un po’ ridotta ma non drammatica), ma funzione diastolica (il riempimento) gravemente compromessa (rapporto E/A elevato di 2.1). Entrambi gli atri (le camere superiori) erano dilatati, segno che facevano fatica a svuotarsi nei ventricoli rigidi. C’erano anche rigurgiti valvolari (mitralico moderato, tricuspidale lieve).
La scoperta di questa variante omozigote c.406 C>T nel gene TNNI3 in una paziente con RCM, ritardo della crescita e dispnea progressiva è un tassello importante. Rafforza il legame tra TNNI3 e le cardiomiopatie, in particolare quelle con disfunzione diastolica severa. È interessante notare come casi simili in letteratura, legati ad altre varianti di TNNI3, mostrino spesso esordi precoci e gravi, talvolta con implicazioni sistemiche come il ritardo di crescita, proprio come nel nostro caso.
Questo studio, sebbene basato su un singolo caso (e questo è un limite, lo ammetto!), sottolinea quanto sia cruciale la valutazione genetica nella RCM. Identificare la causa genetica non solo aiuta nella diagnosi e nella comprensione dei meccanismi della malattia, ma apre anche la porta alla consulenza genetica per le famiglie e, speriamo in futuro, a terapie mirate. Al momento, purtroppo, la gestione della RCM associata a TNNI3 è principalmente di supporto (diuretici, a volte beta-bloccanti o ACE-inibitori), ma non risolutiva.
La nostra scoperta della variante c.406 C>T, probabilmente patogenica e responsabile di una proteina TNNI3 troncata, aggiunge un pezzo al complesso puzzle delle cardiomiopatie ereditarie. Ci ricorda che dobbiamo continuare a indagare le basi genetiche di queste malattie per poter offrire, un giorno, soluzioni concrete ai pazienti e alle loro famiglie. La strada è ancora lunga, ma ogni passo avanti nella conoscenza è una vittoria!
Fonte: Springer