TACIT: Decodificare Tipi e Stati Cellulari nella Multiomica Spaziale
Ciao a tutti! Oggi voglio parlarvi di qualcosa di veramente affascinante che sta cambiando il modo in cui guardiamo dentro i tessuti biologici: la biologia spaziale. Immaginate di poter vedere non solo quali cellule compongono un tessuto, ma esattamente dove si trovano e come interagiscono tra loro, quasi come avere una mappa super dettagliata di una città vivente a livello microscopico. Questo campo è esploso grazie a tecnologie incredibili che ci permettono di analizzare molecole come RNA (trascrittomica) e proteine (proteomica) mantenendo intatta la loro posizione nello spazio. Addirittura, ora possiamo combinare queste analisi sullo stesso pezzetto di tessuto! Fantastico, vero?
La Sfida: Identificare le Cellule nel Caos Organizzato
Ma c’è un “ma”. Con tutti questi dati incredibili, una delle sfide più grandi e, diciamocelo, a volte frustranti, è capire esattamente che tipo di cellula stiamo guardando e in che stato si trova (ad esempio, se è attiva, a riposo, infiammata…). Questo processo, chiamato annotazione cellulare, è spesso lento, pieno di potenziali errori e complicato da diversi fattori:
- Rumore tecnico dovuto alla segmentazione (il processo di delineare i confini di ogni cellula).
- Segnali che “sbavano” da una cellula all’altra (bleed-through).
- Pannelli di marcatori (le molecole che usiamo per identificare le cellule) a volte limitati.
- La complessità di gestire dati multimodali (cioè, da diverse tecnologie come trascrittomica e proteomica insieme).
I metodi tradizionali, come il clustering non supervisionato (che raggruppa cellule simili basandosi sul loro profilo molecolare generale), spesso faticano quando i tipi cellulari sono definiti da pochi marcatori specifici, o quando dobbiamo trovare popolazioni cellulari rare ma magari importantissime. Anche gli algoritmi di deep learning, pur potenti, hanno bisogno di enormi quantità di dati di addestramento e faticano a generalizzare tra tessuti, specie o condizioni diverse. Insomma, avevamo bisogno di uno strumento più agile, robusto e universale.
La Nostra Soluzione: Ecco a Voi TACIT!
Ed è qui che entra in gioco TACIT (Threshold-based Assignment of Cell Types from Multiplexed Imaging DaTa). Ho avuto il piacere di contribuire allo sviluppo di questo algoritmo, ed è pensato proprio per superare queste difficoltà. La bellezza di TACIT è che è non supervisionato, il che significa che non ha bisogno di dati di addestramento pre-esistenti. Funziona utilizzando delle “firme” molecolari predefinite per ogni tipo cellulare che ci aspettiamo di trovare.
Come fa? In pratica, TACIT usa un approccio intelligente basato su soglie (thresholding) per distinguere i segnali “veri” (cellule positive per un marcatore) dal rumore di fondo. Si concentra sui marcatori davvero rilevanti per ogni tipo cellulare e ha un meccanismo specifico per risolvere i casi ambigui, cioè quelle cellule che sembrano esprimere marcatori di più tipi cellulari contemporaneamente (le chiamiamo “mixed cells”).
Il processo, in breve:
- Si parte dalla matrice di dati CELLULA x MARCATORE ottenuta dopo la segmentazione.
- Si definisce una matrice TIPO CELLULARE x MARCATORE (la nostra “firma”).
- Le cellule vengono raggruppate in piccoli “microcluster” (MC) molto omogenei.
- Calcoliamo un punteggio di “Rilevanza del Tipo Cellulare” (CTR) per ogni cellula rispetto a ogni tipo cellulare definito.
- TACIT impara automaticamente una soglia ottimale per ogni CTR, separando i segnali forti dal rumore, analizzando la distribuzione dei punteggi CTR mediani nei microcluster tramite regressione segmentale.
- Le cellule che superano una sola soglia sono classificate come “pulite” (clean). Quelle che ne superano più d’una sono “miste” (mixed).
- Le cellule miste vengono “deconvolute” analizzando le cellule vicine (usando l’algoritmo k-NN) in uno spazio definito solo dai marcatori rilevanti per quell’ambiguità.
Questo approccio mirato ci permette di essere molto più precisi, specialmente con dati sparsi o complessi.

TACIT alla Prova dei Fatti: Accuratezza e Scalabilità
Ovviamente, non basta dire che funziona, bisogna dimostrarlo! Abbiamo messo alla prova TACIT su ben cinque dataset diversi, provenienti da cervello, intestino e ghiandole salivari, sia umani che murini, per un totale di quasi 5 milioni di cellule e 51 tipi cellulari diversi. Abbiamo confrontato le performance di TACIT con altri metodi non supervisionati molto usati (come CELESTA, SCINA e il classico clustering Louvain).
I risultati? Sono stati davvero incoraggianti! TACIT ha costantemente superato gli altri metodi in termini di accuratezza (misurata con F1 score, precision e recall) e si è dimostrato incredibilmente scalabile, capace di gestire dataset enormi anche su un normale laptop. È riuscito a identificare correttamente sia le popolazioni cellulari dominanti che quelle rare, cosa che altri metodi faticavano a fare. Abbiamo anche confrontato TACIT con metodi più recenti, inclusi quelli basati su deep learning, su dataset di benchmark pubblici (PCF-CRC per la proteomica e MERFISH per la trascrittomica), e anche qui TACIT si è distinto per le sue performance superiori.
Applicazioni Reali: Dalle Malattie Autoimmuni alla Multiomica
Ma dove TACIT brilla davvero è nelle applicazioni concrete. Lo abbiamo usato per studiare due malattie infiammatorie delle ghiandole esocrine (simili alla sindrome di Sjögren), riuscendo a identificare nuovi fenotipi cellulari e associazioni tra cellule immunitarie e strutturali che prima ci sfuggivano.
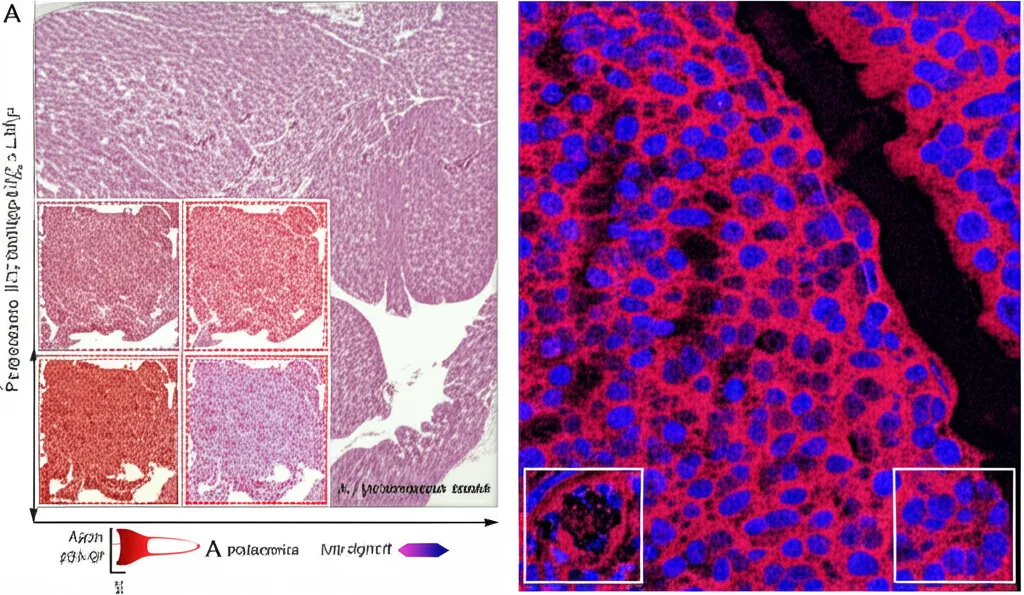
Poi abbiamo fatto qualcosa di ancora più avanzato: abbiamo applicato la trascrittomica spaziale (Xenium) e la proteomica spaziale (PhenoCycler-Fusion) sulla stessa identica sezione di tessuto di ghiandole salivari minori affette da malattia del trapianto contro l’ospite (GvHD). Questo ci ha permesso di avere dati di RNA e proteine per ogni singola cellula! Utilizzando TACIT, siamo riusciti ad annotare le cellule in questo complesso scenario multimodale.
Qui abbiamo scoperto cose interessantissime:
- La correlazione tra i livelli di un marcatore misurato come proteina (PCF) e come RNA (Xenium) è spesso bassa, specialmente per le cellule immunitarie. Questo ci dice che dobbiamo essere cauti quando interpretiamo dati da una sola modalità!
- Usando solo i marcatori comuni ad entrambe le tecnologie, l’accordo nell’identificazione dei tipi cellulari tra proteomica e trascrittomica è salito notevolmente (dall’34% all’81%). Questo suggerisce l’importanza di pannelli multimodali ben progettati.
- Siamo riusciti a studiare anche lo stato cellulare, ad esempio l’espressione di marcatori di checkpoint immunitario come PD-1/PDCD1 o di proliferazione come Ki-67/MKI67, vedendo differenze significative tra i livelli proteici e di RNA nella stessa cellula. Questo ha implicazioni cliniche enormi, per esempio per predire la risposta alle immunoterapie.

Abbiamo anche analizzato delle aree particolarmente complesse chiamate strutture linfoidi terziarie (TLS), zone ad alta densità di cellule immunitarie dove la segmentazione è una vera sfida. Anche qui, TACIT si è dimostrato capace di “ripulire” i segnali misti dovuti a errori di segmentazione, fornendo un quadro molto più dettagliato e biologicamente sensato della composizione cellulare e delle interazioni all’interno di queste nicchie rispetto ad altri metodi come Louvain. Ad esempio, TACIT ha rivelato la presenza di piccole navi vascolari e cellule dendritiche vicine alle cellule T, interazioni chiave per la funzione immunitaria che Louvain non riusciva a cogliere.
Perché TACIT Fa la Differenza
Quindi, perché credo che TACIT sia un passo avanti importante? Perché affronta direttamente i limiti dei metodi attuali:
- Automatizza un processo altrimenti lungo e soggettivo.
- È preciso nell’identificare tipi cellulari basandosi su firme specifiche, anche quelle rare.
- È scalabile a milioni di cellule.
- È robusto e non richiede tuning infinito di parametri.
- È intrinsecamente multimodale, pronto per i dati del futuro.
- È agnostico rispetto alla tecnologia, alla specie, all’organo o alla malattia.
Il suo successo deriva dal focus iniziale sulle caratteristiche specifiche del tipo cellulare e dalla capacità di risolvere le ambiguità guardando solo ai marcatori pertinenti.
Uno Sguardo al Futuro
La biologia spaziale è solo all’inizio del suo viaggio. Il futuro vedrà l’integrazione di ancora più strati di informazioni: epigenomica spaziale (come le modifiche al DNA influenzano l’espressione genica nello spazio), metabolomica spaziale (le firme metaboliche delle cellule), sequenziamento dei recettori delle cellule B e T, e persino la combinazione con le colorazioni istologiche tradizionali (come l’Ematossilina-Eosina) che i patologi usano da sempre. Immaginate atlanti tissutali completi che combinano tutti questi dati! TACIT è uno strumento progettato per essere pronto a questa rivoluzione multi-omica, aiutandoci a costruire mappe sempre più dettagliate della vita a livello cellulare e, speriamo, a tradurre queste scoperte in diagnosi più precise e terapie personalizzate. È un momento incredibilmente eccitante per essere in questo campo!

Fonte: Springer