L’Abbraccio Molecolare che Organizza il Cervello: Svelata la Struttura del Complesso C1ql3-BAI3!
Ciao a tutti, appassionati di scienza e misteri del cervello! Oggi voglio portarvi con me in un viaggio affascinante nel cuore delle nostre connessioni neuronali, le sinapsi. Immaginatele come i punti di contatto, le “strette di mano” tra neuroni che permettono al nostro cervello di funzionare. Al centro di questa danza complessa ci sono proteine specifiche, e una di queste, chiamata BAI3 (Brain-specific Angiogenesis Inhibitor-3), gioca un ruolo da protagonista nell’organizzare queste sinapsi.
Per anni, però, un pezzo importante del puzzle è rimasto oscuro: come fa esattamente BAI3 a interagire con uno dei suoi partner molecolari, una proteina chiamata C1ql3 (appartenente alla famiglia delle C1q-like)? Sembra una domanda tecnica, ma capire questo “abbraccio” molecolare è fondamentale per comprendere come si formano e si mantengono le connessioni nel nostro cervello.
Beh, tenetevi forte, perché oggi vi racconto come siamo riusciti a “fotografare” questo momento cruciale!
Una Foto ad Altissima Risoluzione: La Crio-Microscopia Elettronica
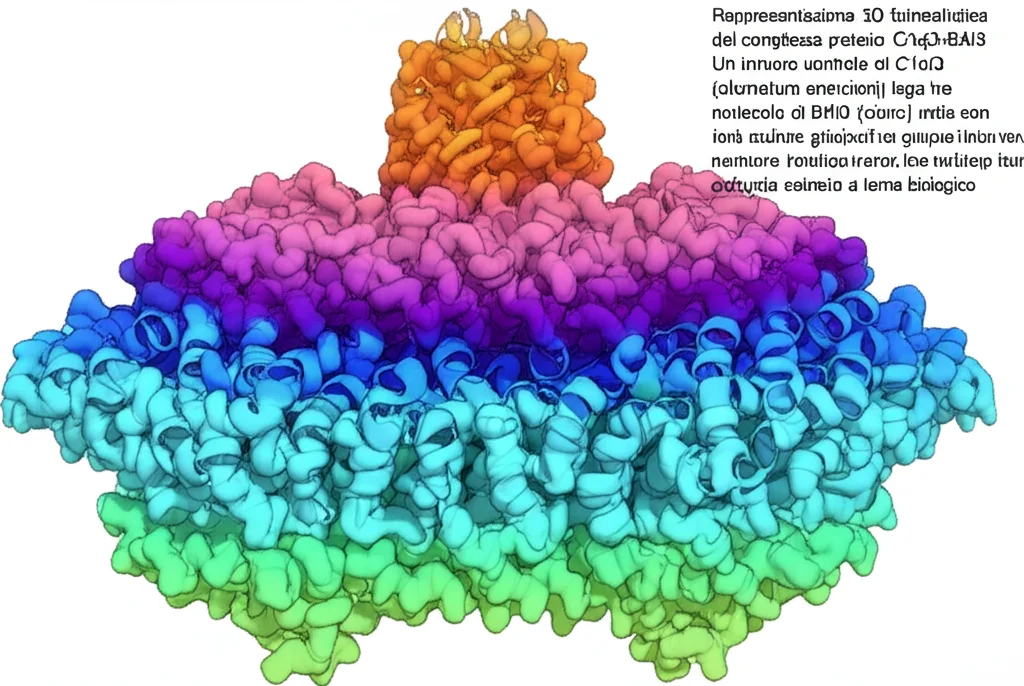
Grazie a una tecnica potentissima chiamata crio-microscopia elettronica (cryo-EM) a particella singola, siamo riusciti a ottenere un’immagine incredibilmente dettagliata, con una risoluzione di 2.8 Ångström (pensate, è una misura piccolissima, a livello atomico!), del complesso formato da C1ql3 e BAI3. È come avere una mappa 3D precisissima di come queste due molecole si legano.
E cosa abbiamo scoperto? La struttura ha rivelato una configurazione sorprendente: una sorta de “fiore molecolare” esamerico. Al centro, tre molecole di C1ql3 si uniscono per formare un trimero (un trio). Questo trimero centrale agisce come una sorta di “calamita” che cattura efficacemente tre molecole di BAI3. Immaginate queste molecole di BAI3 che si incastrano perfettamente nelle scanalature presenti tra i domini C1q trimerici delle C1ql3. È un incastro quasi perfetto, un design molecolare elegante!
Un Legame Speciale Mediato dal Calcio
Ma la vera sorpresa è stata scoprire *come* avviene questo legame. Abbiamo visto che gli ioni calcio (Ca2+) giocano un ruolo cruciale, agendo come dei piccoli “ponti” che aiutano a tenere insieme le due proteine. La cosa affascinante è che questo meccanismo di interazione mediato dal calcio è diverso da quello osservato in altri complessi simili precedentemente studiati, come quello tra Cerebellina-1 (Cbln1) e GluD2, un altro importante sistema di organizzazione sinaptica.
Nel nostro complesso C1ql3-BAI3, lo ione calcio è coordinato (cioè “tenuto in posizione”) da residui amminoacidici specifici sia di C1ql3 (Asp180 e Asp232) sia di BAI3 (Lys66 e Asp63), anche se attraverso molecole d’acqua che non siamo riusciti a visualizzare direttamente ma la cui presenza è implicata. Inoltre, c’è un’interazione chiamata “π-stacking” (una sorta di attrazione tra anelli aromatici) che coinvolge residui specifici (Trp188 di C1ql3, Lys66 e Tyr67 di BAI3) e che stabilizza ulteriormente il complesso. Non solo, anche un secondo dominio C1q contribuisce all’interfaccia, con legami idrogeno tra residui come Tyr244, Asn242, Asn240 di C1ql3 e Asp63, Glu60 di BAI3. Insomma, un’interazione robusta e ben definita su un’area totale di circa 564 Ångström quadrati!

Identikit di BAI3: Un Dominio CUB con Sorprese
La struttura ci ha anche permesso di capire meglio la natura di una parte specifica di BAI3, il suo dominio N-terminale (NTD). Facendo ricerche in database strutturali (usando uno strumento chiamato DALI), abbiamo scoperto che assomiglia molto ai cosiddetti domini CUB, trovati in altre proteine come la C1r del sistema del complemento. Nonostante una bassa somiglianza nella sequenza di amminoacidi (solo 8-19%), la forma tridimensionale del “cuore” della struttura (i foglietti beta) è molto conservata. La differenza principale? Il dominio NTD di BAI3 ha delle eliche alfa aggiuntive all’esterno di questo nucleo, che potrebbero essere coinvolte in altre interazioni ancora sconosciute. Questo suggerisce che il dominio CUB di BAI3 potrebbe avere funzioni regolatorie o di segnalazione, anche se diverse da altri domini CUB noti come quello di GPR126.
Perché BAI1 è Diverso? Il Segreto nella Sequenza
Un’altra domanda che ci incuriosiva era: perché BAI3 e la sua “sorella” BAI2 legano C1ql, mentre un’altra proteina molto simile, BAI1, non lo fa? Analizzando le sequenze e usando modelli predittivi come AlphaFold, abbiamo trovato la risposta. Nonostante la somiglianza generale, alcuni amminoacidi cruciali per il legame con C1ql sono conservati in BAI2 ma non in BAI1.
In particolare, due sostituzioni in BAI1 rispetto a BAI3 sono determinanti:
- Una Treonina (Thr65 in BAI3) diventa Arginina (Arg) in BAI1. Questa sostituzione crea un “ingombro” fisico (sterico) che impedisce il legame con C1ql3.
- Un Acido Glutammico (Glu60 in BAI3) diventa Arginina (Arg) in BAI1, rompendo un legame idrogeno importante con C1ql3 (Asn240).
Queste differenze spiegano perché solo BAI2 e BAI3, ma non BAI1, possono “stringere la mano” a C1ql. Abbiamo anche visto che questa differenza è conservata nell’evoluzione: guardando le proteine BAI in diverse specie (dal pesce zebra all’uomo), BAI1 manca costantemente di questi residui chiave per il legame con C1ql. Al contrario, i residui di C1ql3 che legano BAI sono conservatissimi nelle altre isoforme di C1ql (C1ql1, C1ql2, C1ql4), spiegando perché tutte loro possono legare BAI2/3.
La Prova del Nove: Esperimenti in Cellula
Per confermare le nostre scoperte strutturali, abbiamo fatto esperimenti direttamente su cellule (HEK293T) in laboratorio. Abbiamo “decorato” la superficie di queste cellule con BAI3 o BAI1 e poi abbiamo aggiunto C1ql3 marcata. I risultati sono stati chiari: C1ql3 si legava brillantemente alle cellule con BAI3, ma ignorava quelle con BAI1.
Abbiamo anche testato delle mutazioni: cambiando anche un solo amminoacido chiave in BAI3 (come Thr65 in Arg) si perdeva il legame con C1ql3. Allo stesso modo, mutando residui specifici nell’interfaccia di C1ql3 (come un triplo mutante Asn240Ala, Asn242Ala e Tyr244Ala) si impediva l’interazione. Questo ha confermato l’importanza dei contatti che avevamo identificato nella nostra struttura 3D.

Un Modo Diverso di “Tenersi per Mano”: C1ql3 vs Cbln1
Come accennavo prima, il modo in cui C1ql3 lega BAI3 è diverso da altri complessi noti che coinvolgono domini C1q-like. Il caso più studiato è quello di Cbln1 che lega GluD2. Lì, un trimero di Cbln1 lega una sola copia di GluD2 (stechiometria 3:1). Nel nostro caso, invece, il trimero di C1ql3 lega tre copie di BAI3 (stechiometria 3:3). È una differenza fondamentale!
Inoltre, il ruolo del calcio è diverso, e le superfici molecolari utilizzate per il legame sono distinte. Questo ci dice che i domini C1q-like sono incredibilmente versatili e possono adottare diverse strategie per interagire con i loro partner.
Un’Ipotesi Affascinante: Reticoli Esagonali alle Sinapsi?
Considerando la struttura che abbiamo osservato (3 C1ql3 + 3 BAI3) e il fatto che C1ql3 potrebbe formare strutture più grandi (forse 18-meri), abbiamo formulato un’ipotesi intrigante. Forse C1ql3 e BAI3 possono organizzarsi in un reticolo esagonale sulla superficie della sinapsi, un po’ come avviene per altre famiglie di recettori e ligandi (come TNF).
Cosa significherebbe? Che C1ql potrebbe agire raggruppando, “clusterizzando”, le molecole di BAI3 in punti specifici della sinapsi. Questa oligomerizzazione indotta dal ligando potrebbe essere un meccanismo importante per regolare la segnalazione di BAI3 (che è un recettore accoppiato a proteine G, GPCR) e contribuire direttamente al modo in cui C1ql3 aiuta a formare e stabilizzare le sinapsi. È un’idea eccitante che apre nuove prospettive sul ruolo multifunzionale delle proteine C1ql nello sviluppo neuronale. Ovviamente, sottolineamo che questa del reticolo esagonale è ancora un’ipotesi che richiederà ulteriori indagini per essere confermata.
Conclusioni e Prospettive
In sintesi, il nostro studio ha svelato la struttura atomica del complesso C1ql3-BAI3, mostrando una geometria e un’interfaccia di legame mai osservate prima per un complesso recettore/dominio C1q-like. Abbiamo capito perché BAI3 e BAI2 legano C1ql, ma BAI1 no, e abbiamo evidenziato il ruolo cruciale e unico del calcio in questa interazione.
Questi risultati non solo ci aiutano a capire meglio come si organizzano le sinapsi, ma suggeriscono anche un nuovo meccanismo potenziale attraverso cui le proteine C1ql potrebbero agire come organizzatori sinaptici, ovvero clusterizzando i recettori BAI. È un passo avanti importante che ci ricorda quanto sia complessa ed elegante la biologia molecolare alla base del funzionamento del nostro cervello!
Fonte: Springer