snCED-seq: Il Freddo che Rivela i Segreti dell’RNA nei Tessuti d’Archivio
Un Tesoro Nascosto nei Tessuti d’Archivio
Immaginate un tesoro di informazioni mediche, accumulato per decenni negli archivi ospedalieri: milioni di campioni di tessuto fissati in formalina e inclusi in paraffina (i cosiddetti FFPE). Questi campioni sono una miniera d’oro potenziale per capire le malattie, scoprire nuovi biomarcatori e sviluppare terapie mirate. C’è solo un piccolo problema: estrarre informazioni molecolari di alta qualità, specialmente l’RNA, da questi tessuti è sempre stata una sfida enorme. La formalina, usata per preservare la struttura del tessuto, crea dei legami chimici (cross-linking) che “intrappolano” e danneggiano l’RNA, rendendo difficile analizzarlo con tecniche moderne come il sequenziamento dell’RNA a singola cellula (o meglio, a singolo nucleo, snRNA-seq).
La Sfida dell’Estrazione dai Tessuti FFPE
Per anni, noi ricercatori ci siamo scontrati con questo muro. I metodi tradizionali per isolare i nuclei (il “cuore” della cellula che contiene il materiale genetico) dai tessuti FFPE spesso richiedono trattamenti aggressivi, come alte temperature ed enzimi potenti (un approccio che potremmo chiamare HED – High-temperature Enzymatic Dissociation). Questi metodi, purtroppo, possono danneggiare ulteriormente l’RNA già fragile, rompere la delicata membrana nucleare e portare alla perdita di materiale genetico prezioso. Inoltre, spesso si ottiene una bassa resa di nuclei e molta “spazzatura” (detriti cellulari), che complica le analisi successive. Altri approcci, basati sull’omogeneizzazione meccanica (MHD), funzionano meglio sui campioni freschi o congelati, ma sui tessuti FFPE, resi più resistenti dalla fissazione, faticano a rompere il tessuto in modo efficiente, portando ancora a bassa resa, perdita di nuclei più piccoli e contaminazione da detriti e RNA ribosomiale (rRNA). Insomma, ottenere nuclei intatti e di alta qualità da campioni FFPE era un po’ come cercare un ago in un pagliaio… un pagliaio molto appiccicoso e danneggiato.
La Nostra Soluzione: Il Freddo è la Chiave (snCED-seq)
E se la soluzione fosse… il freddo? Ci siamo chiesti: invece di combattere la “rigidità” del tessuto FFPE con il calore, perché non sfruttare le basse temperature? Da qui è nata la nostra idea: la Dissociazione Enzimatica Criogenica (CED). Abbiamo sviluppato un protocollo che utilizza enzimi specifici (come la proteinasi K per digerire le proteine in eccesso) e detergenti delicati (come il sarcosyl, più “gentile” sulla membrana nucleare rispetto ad altri) a basse temperature (4°C). Questo approccio “freddo” ha diversi vantaggi cruciali:
- Rallenta le reazioni enzimatiche indesiderate che potrebbero degradare l’RNA.
- Preserva meglio l’integrità della membrana nucleare, evitando la perdita di molecole di RNA.
- Elimina la necessità di passaggi complessi e potenzialmente dannosi come l’ultracentrifugazione su gradiente di saccarosio o filtrazioni multiple.
Il risultato? Un metodo più rapido, semplice e, soprattutto, capace di estrarre un numero molto maggiore di nuclei di alta qualità, pronti per l’analisi snRNA-seq. Abbiamo chiamato questa combinazione snCED-seq.
La Prova del Nove: snCED-seq vs Metodi Convenzionali
Naturalmente, non ci siamo fidati solo delle nostre intuizioni. Abbiamo messo alla prova snCED-seq confrontandolo fianco a fianco con i metodi convenzionali (HED e MHD) su campioni di cervello di topo, sia fissati (PFA, FFPE) che freschi/congelati (FF). I risultati sono stati sbalorditivi e hanno confermato la superiorità del nostro approccio criogenico:
- Resa Nucleare Decuplicata: Abbiamo ottenuto fino a 10 volte più nuclei rispetto ai metodi tradizionali partendo dalla stessa quantità di tessuto. Questo è fondamentale, perché molte piattaforme snRNA-seq richiedono un alto numero di nuclei in ingresso.
- RNA di Qualità Superiore: L’RNA estratto dai nuclei isolati con CED mostrava una minore degradazione (valori RIN più alti e frammenti di cDNA più lunghi), molto più simile a quello ottenuto da campioni freschi/congelati rispetto ai nuclei ottenuti con metodi ad alta temperatura.
- Maggiore Sensibilità e Ricchezza Genica: Le analisi snCED-seq hanno permesso di rilevare un numero significativamente maggiore di geni e trascritti (UMIs) per singolo nucleo rispetto ai metodi HED.
- Minore Contaminazione: Abbiamo osservato una percentuale drasticamente ridotta di geni mitocondriali e ribosomiali, indicando una maggiore purezza dei nuclei e minore contaminazione citoplasmatica.
- Migliore Copertura Intronica: snCED-seq ha mostrato una maggiore copertura delle regioni introniche, suggerendo una migliore preservazione dell’RNA nucleare (che contiene molti trascritti non ancora processati) e nuclei con minore contaminazione citoplasmatica.
- Robustezza e Riproducibilità: I profili di espressione genica ottenuti con snCED-seq erano altamente correlati tra repliche tecniche e mostravano una maggiore somiglianza con i campioni freschi/congelati rispetto ai metodi HED. Sorprendentemente, i nuclei isolati con CED potevano essere crioconservati senza danni evidenti, rompendo il dogma che i nuclei siano estremamente sensibili al congelamento/scongelamento.
In pratica, snCED-seq ci ha permesso di ottenere dati di sequenziamento più profondi, puliti e affidabili dai campioni FFPE, avvicinandoci alla qualità ottenibile dai campioni freschi.
Applicazioni nel Mondo Reale: Alzheimer e Tessuto Polmonare Umano
Ma a cosa serve una nuova tecnica se non può essere applicata a problemi reali? Abbiamo testato snCED-seq su due fronti importanti:
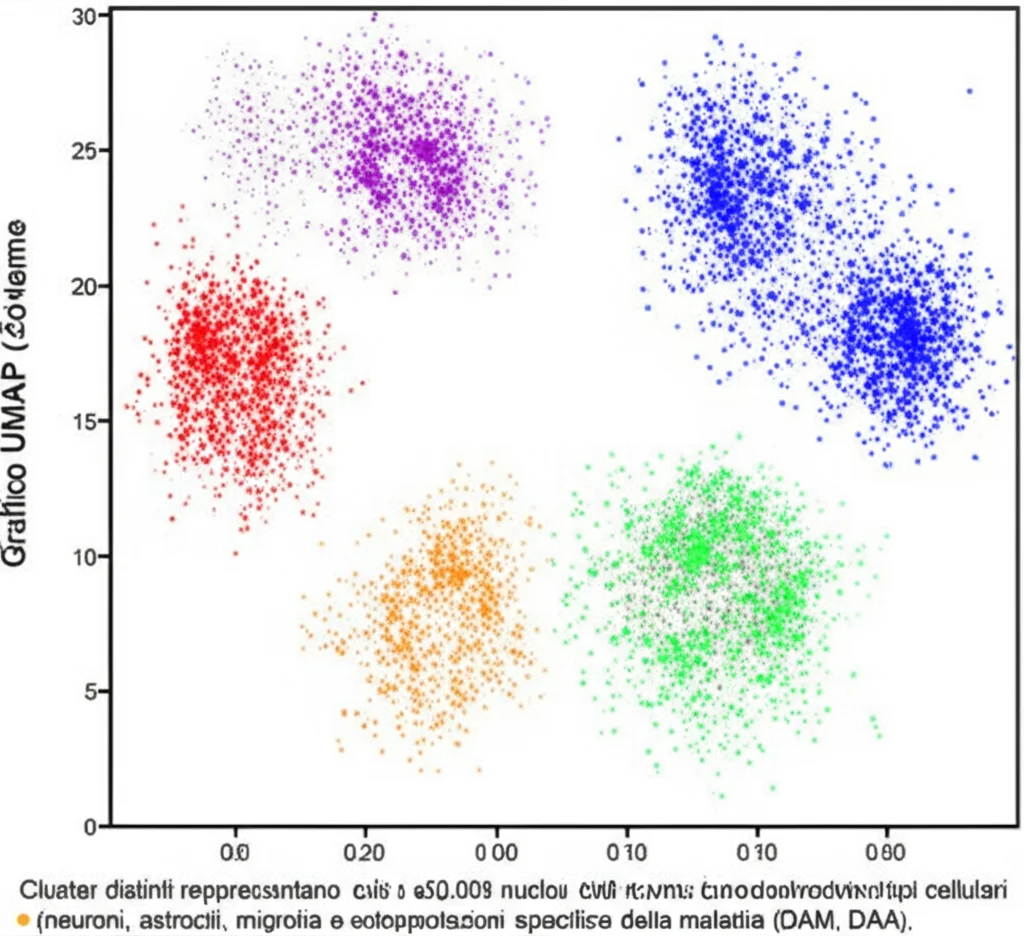
1. Studio sulla Malattia di Alzheimer (AD): Abbiamo analizzato campioni di ippocampo FFPE provenienti da topi modello per l’Alzheimer (5xFAD) e topi di controllo sani. Utilizzando snCED-seq, siamo riusciti a identificare oltre 60.000 nuclei di alta qualità e a caratterizzare la complessa eterogeneità cellulare in questa regione cerebrale cruciale per la memoria. Non solo abbiamo distinto i principali tipi cellulari (neuroni eccitatori e inibitori, astrociti, oligodendrociti, microglia, ecc.), ma siamo anche riusciti a identificare sottopopolazioni specifiche associate alla malattia, come:
- Due distinti stati di microglia associata alla malattia (DAM), uno dei quali proliferante.
- Due tipi di astrociti associati alla malattia (DAA), con profili trascrizionali specifici e arricchiti nei topi AD.
- Oligodendrociti associati alla malattia (DAO), che mostravano sorprendenti somiglianze trascrizionali con alcuni DAA, suggerendo possibili percorsi comuni attivati dalla patologia.
Abbiamo anche rilevato cambiamenti trascrizionali significativi in cellule vascolari (endoteliali ed ependimali), spesso meno studiate nell’AD. Questi risultati dimostrano che snCED-seq può svelare dettagli cellulari fini legati alla malattia anche da campioni FFPE.
2. Analisi di Tessuto Polmonare Umano FFPE: Per dimostrare la versatilità del metodo su campioni clinici umani, abbiamo applicato snCED-seq a una singola sezione sottile (50 µm) di tessuto polmonare umano FFPE (paracanceroso). Anche da questo piccolo campione, siamo riusciti a isolare circa 150.000 nuclei e, dopo il sequenziamento, a identificare con successo tutti i principali tipi cellulari attesi nel polmone: cellule epiteliali alveolari (AT1, AT2), cellule immunitarie (linfociti B/T, mastociti, macrofagi), cellule endoteliali e fibroblasti. Questo risultato è importantissimo perché dimostra che snCED-seq può essere utilizzato anche su biopsie o campioni clinici di piccole dimensioni, aprendo la strada all’analisi trascrittomica di routine su materiale d’archivio umano.
Perché snCED-seq Cambia le Regole del Gioco
La capacità di estrarre nuclei di alta fedeltà da campioni FFPE con snCED-seq è, a mio avviso, un vero e proprio cambio di paradigma. Significa poter finalmente attingere a quell’immenso patrimonio di campioni clinici archiviati, corredati da preziosi dati clinici e di follow-up dei pazienti. Questo apre possibilità enormi:
- Studi retrospettivi su larga scala: Analizzare coorti di pazienti del passato per identificare nuovi biomarcatori o capire perché alcuni pazienti rispondono alle terapie e altri no.
- Comprensione più profonda delle malattie: Studiare l’eterogeneità cellulare e le interazioni tra cellule in campioni umani reali, non solo in modelli animali o colture cellulari.
- Medicina di precisione: Potenzialmente, utilizzare l’analisi trascrittomica su campioni FFPE di routine per guidare diagnosi e scelte terapeutiche personalizzate.
- Oltre il trascrittoma: La qualità dei nuclei ottenuti con CED pone le basi anche per future applicazioni multi-omiche (analisi simultanea di RNA, DNA, proteine, accessibilità cromatinica) sullo stesso campione FFPE.
Il nostro metodo è relativamente semplice, non richiede attrezzature particolari oltre a quelle standard di un laboratorio di biologia molecolare e, come abbiamo visto, è robusto e applicabile a diversi tipi di tessuto. Crediamo fermamente che snCED-seq possa diventare uno strumento fondamentale per sbloccare il potenziale nascosto negli archivi FFPE, accelerando la ricerca biomedica e avvicinandoci a una comprensione più completa delle malattie umane. Il freddo, in questo caso, non conserva soltanto, ma rivela!
Fonte: Springer