SlideCNA: La Mappa Genetica dei Tumori Diventa Spaziale (e Super Dettagliata!)
Ciao a tutti! Oggi voglio parlarvi di qualcosa di veramente affascinante che sta cambiando il modo in cui guardiamo i tumori. Immaginate un tumore non come una massa uniforme, ma come una città complessa, con quartieri diversi, ognuno con le sue caratteristiche, i suoi abitanti (le cellule) e persino i suoi “codici genetici” specifici. Capire questa mappa spaziale è fondamentale per comprendere come cresce un tumore, come interagisce con il suo ambiente e come risponde alle terapie.
La Sfida: Vedere i Dettagli Genetici nello Spazio
Negli ultimi anni, sono nate tecnologie pazzesche chiamate “trascrittomica spaziale”. Queste ci permettono di “fotografare” quali geni sono attivi (l’RNA) in diverse zone di un tessuto, mantenendo la loro posizione originale. Pensate a tecniche come Slide-seq: ci danno una risoluzione incredibile, quasi a livello di singola cellula! Il problema? Più andiamo nel dettaglio, più i dati diventano “sparsi”, cioè abbiamo poche informazioni per ogni singolo punto di misurazione (chiamato “bead” o “spot”).
Ora, una cosa super importante da capire nei tumori sono le alterazioni del numero di copie (CNA). In pratica, pezzi interi di DNA possono essere duplicati o persi nelle cellule tumorali, e questo influenza tantissimo il loro comportamento. Esistono già metodi computazionali, come InferCNV o CopyKAT, che provano a dedurre queste CNA dai dati di RNA di singole cellule (scRNA-seq). Hanno fatto un ottimo lavoro, ma quando proviamo ad applicarli ai dati spaziali super dettagliati ma sparsi come quelli di Slide-seq… beh, fanno un po’ fatica. Catturano RNA da cellule vicine che potrebbero essere diverse, i segnali sono deboli e, soprattutto, non sfruttano appieno l’informazione più preziosa: la posizione.
La Nostra Idea: Nasce SlideCNA!
Ed è qui che entriamo in gioco noi! Ci siamo detti: “E se potessimo creare uno strumento che non solo gestisca la sparsità dei dati di Slide-seq, ma usi anche la mappa spaziale per migliorare il rilevamento delle CNA?”. Così è nato SlideCNA.
Come funziona? L’idea di base è semplice ma potente. Invece di guardare ogni “bead” isolato, SlideCNA fa una cosa intelligente: raggruppa i bead vicini che hanno profili di espressione genica simili. Lo chiamiamo “binning spazio-molecolare”. In pratica, calcoliamo quanto due bead sono vicini sia fisicamente sul vetrino, sia in termini di geni attivi. Poi combiniamo queste due “distanze” per creare dei “super-bead” o “bin”. Questo ci permette di sommare i segnali deboli di più bead vicini e simili, ottenendo un quadro molto più chiaro delle CNA, senza però perdere la preziosa informazione spaziale. È un po’ come ascoltare meglio dei sussurri in una folla raggruppando le persone vicine che stanno dicendo cose simili.
Una volta creati questi bin, usiamo un approccio simile a InferCNV per “lisciare” l’espressione genica lungo i cromosomi e confrontarla con quella di cellule di riferimento (che sappiamo essere non tumorali) per calcolare i punteggi di CNA (amplificazioni o delezioni).
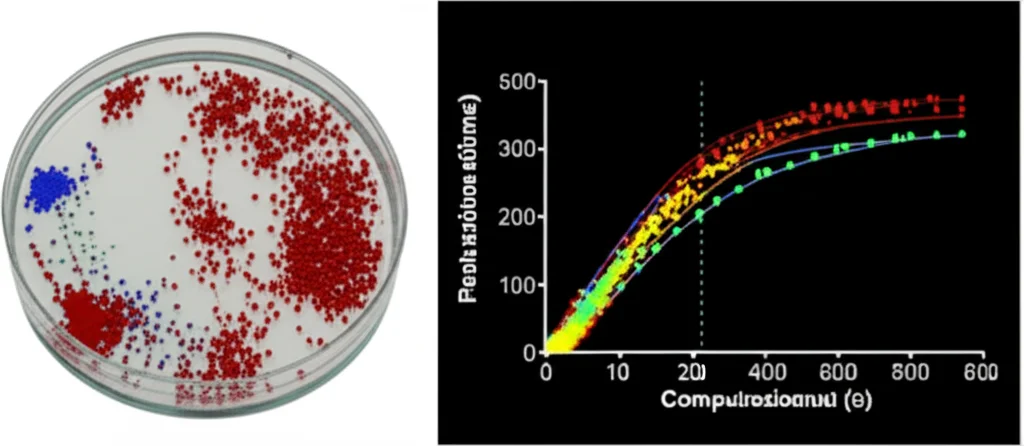
Alla Prova dei Fatti: Simulazioni e Cancro al Seno Metastatico
Ovviamente, non ci siamo fidati solo dell’idea. Abbiamo messo alla prova SlideCNA. Prima, abbiamo creato dei dati “finti” (in silico) che imitassero quelli di Slide-seq, partendo da dati reali di singole cellule di cancro al seno metastatico (MBC). Abbiamo costruito delle mappe spaziali artificiali dove sapevamo esattamente dove si trovavano cellule con diverse CNA e cellule normali, mescolandole in modi diversi. Poi abbiamo reso questi dati “sparsi”, proprio come quelli reali di Slide-seq. Risultato? SlideCNA è riuscito a ricostruire le mappe di CNA e a distinguere i diversi “cloni” tumorali in modo impressionante, molto meglio di quanto ci aspettassimo, anche con dati molto rarefatti! I profili CNA che abbiamo trovato erano super coerenti con quelli ottenuti dai dati originali di singole cellule usando metodi standard come InferCNV.
Poi siamo passati all’azione reale. Abbiamo applicato SlideCNA a campioni reali di Slide-seq di cancro al seno metastatico (gli stessi da cui avevamo preso i dati per le simulazioni). E qui le cose si sono fatte davvero interessanti! Abbiamo visto chiaramente come le CNA variavano nello spazio all’interno del tumore. In un campione, abbiamo identificato due gruppi distinti di cellule tumorali (probabili subcloni) con profili CNA diversi, che occupavano regioni spaziali differenti, corrispondenti a quanto osservato da un patologo esperto su una sezione di tessuto adiacente! In un altro campione, le differenze erano più sottili, ma SlideCNA è stato comunque in grado di distinguerle, mostrandoci la sua sensibilità.
Abbiamo anche verificato la coerenza su repliche tecniche (sezioni seriali dello stesso tumore) e i risultati erano solidissimi. Confrontando i risultati di SlideCNA sui dati Slide-seq con quelli ottenuti da dati “gemelli” di snRNA-seq (usando InferCNV e CopyKAT) e persino da dati di sequenziamento dell’esoma intero (WES), abbiamo visto una coerenza notevole. Anzi, SlideCNA applicato ai dati Slide-seq spesso dava risultati più in linea con i dati snRNA-seq/WES rispetto a quando applicavamo InferCNV o CopyKAT direttamente ai dati Slide-seq. Questo ci dice che il nostro approccio di binning spaziale funziona davvero per recuperare segnali CNA affidabili dalla sparsità di Slide-seq.
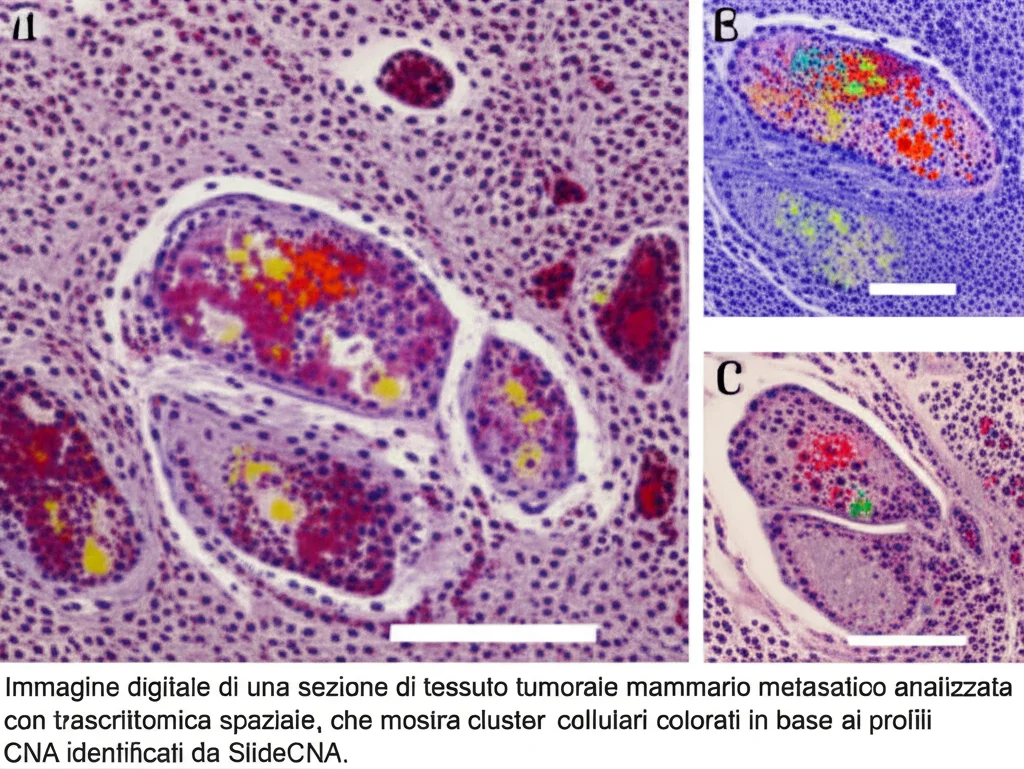
Perché SlideCNA è Speciale (e Cosa Riserva il Futuro)
Quindi, cosa rende SlideCNA un passo avanti?
- Gestisce la sparsità dei dati ad alta risoluzione come Slide-seq.
- Sfrutta attivamente l’informazione spaziale per migliorare il segnale.
- Permette di identificare subcloni tumorali con profili CNA distinti e la loro localizzazione.
- Mostra una maggiore coerenza con altri tipi di dati genomici rispetto ai metodi precedenti applicati a Slide-seq.
Certo, ci sono delle limitazioni. SlideCNA funziona al meglio con dati di trascrittoma intero e con tecnologie spazialmente dense. Non è adatto per pannelli di geni limitati o per tecnologie che non misurano direttamente il conteggio delle molecole di RNA. Abbiamo anche esplorato l’uso della “decomposizione dei bead” (un metodo per stimare i tipi cellulari dentro ogni bead) prima di applicare SlideCNA: sembra promettente per migliorare il segnale quando cellule tumorali e normali sono molto mescolate, ma bisogna stare attenti a possibili artefatti.
Ma il potenziale è enorme! SlideCNA apre le porte a studi su larga scala che collegano direttamente le alterazioni genetiche (CNA) alla loro posizione spaziale, all’espressione genica e all’ecosistema tumorale circostante. Questo ci aiuterà a capire meglio come i tumori evolvono, come resistono alle terapie e come metastatizzano. Insomma, stiamo aggiungendo un livello di dettaglio cruciale alla nostra mappa del cancro, e speriamo che questo porti a nuove strategie per combatterlo. È un campo in rapidissima evoluzione, e non vediamo l’ora di vedere cosa scopriremo dopo!
Fonte: Springer