Fibrosi Epatica: Ho Scoperto Come SIRT7 Accende l’Infiammazione nel Fegato dei Pesci!
Ragazzi, lasciate che vi racconti una storia affascinante che arriva direttamente dal mio laboratorio, una storia che parla di fegato, infiammazione e di come piccole modifiche a livello molecolare possano scatenare grandi problemi. Parliamo di fibrosi epatica, una condizione brutta in cui il fegato si riempie di cicatrici, perdendo la sua funzionalità. È un problema serio, non solo per noi umani ma anche per altre specie, come il nostro protagonista inaspettato: il persico trota (largemouth bass).
Perché proprio il persico trota?
Vi chiederete: cosa c’entra un pesce d’acqua dolce con la fibrosi epatica umana? Beh, questo pesce è particolarmente interessante perché, un po’ come noi, soffre se mangia troppi carboidrati e grassi (la cosiddetta dieta HCHFD). Sviluppa una sorta di diabete di tipo 2 e, guarda caso, anche la fibrosi epatica. Questo lo rende un modello animale quasi perfetto per studiare come la dieta influenzi la salute del fegato. Il problema è che capire *esattamente* cosa succede a livello molecolare è un bel rompicapo. Ed è qui che entriamo in gioco noi.
Una strana scoperta: meno “etichette” nel fegato malato
Abbiamo iniziato osservando i fegati dei persici trota nutriti con la dieta HCHFD o trattati con una sostanza tossica (CCl4) che causa fibrosi. Abbiamo notato una cosa strana: nei fegati malati, pieni di cicatrici (lo vedevamo bene con colorazioni specifiche come il Sirius Red e il Masson, e controllando i livelli della proteina α-SMA, un marcatore classico di fibrosi), c’era una riduzione generale di un tipo specifico di “etichetta” molecolare sulle proteine. Queste etichette si chiamano gruppi acetili, e il processo di aggiungerle o toglierle si chiama acetilazione e deacetilazione. È una delle tante “modifiche post-traduzionali” (PTM) che le cellule usano per regolare finemente la funzione delle proteine. Vedere una diminuzione generale dell’acetilazione nei fegati fibrotici ci ha fatto drizzare le antenne: e se fosse proprio questo il nodo del problema?

L’indagine high-tech: caccia alla proteina chiave
Per capirci di più, abbiamo usato tecniche avanzate (proteomica basata su TMT e spettrometria di massa LC-MS/MS) per mappare tutte le proteine acetilate nei fegati sani e in quelli fibrotici. Immaginate una sorta di censimento molecolare super dettagliato. Abbiamo trovato centinaia di siti di acetilazione diversi, ma la stragrande maggioranza (475 su 497!) era *meno* acetilata nei fegati malati. Tra tutte le proteine coinvolte, una in particolare ha catturato la nostra attenzione: RELA.
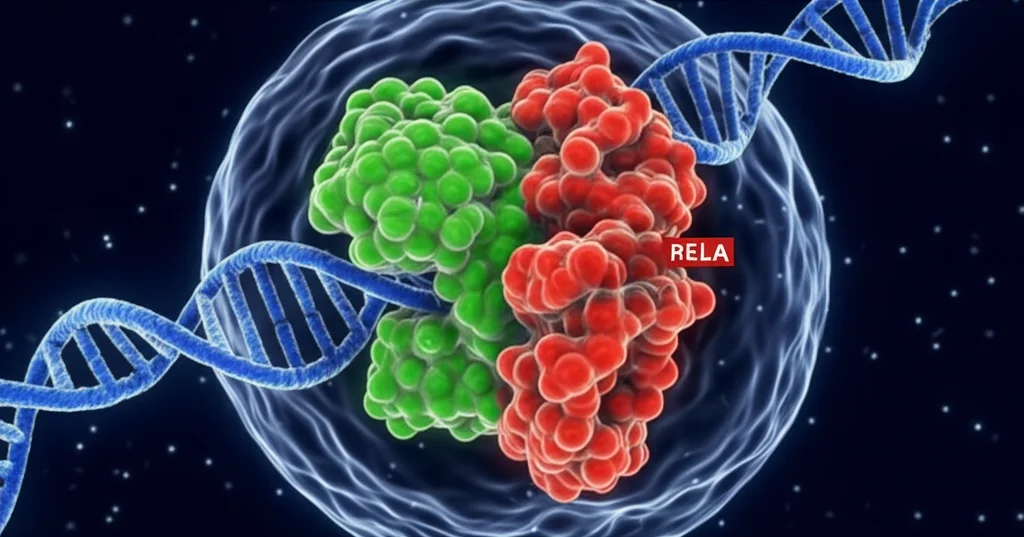
RELA è una vecchia conoscenza nel campo dell’infiammazione. È una subunità chiave del complesso NF-κB, un interruttore molecolare fondamentale che accende la risposta infiammatoria. Il nostro “censimento” ha rivelato che un sito specifico di RELA, la lisina in posizione 119 (che abbiamo chiamato K119), era significativamente deacetilato nei fegati fibrotici. La cosa interessante è che questo sito K119 si trova proprio nella regione della proteina che lega il DNA, ed è conservato in tantissime specie, persino negli esseri umani (dove corrisponde alla lisina K105)!
Abbiamo confermato questa scoperta anche in pesci con infiammazione acuta indotta da LPS (un componente batterico che scatena l’infiammazione) e in cellule epatiche isolate (epatociti primari) trattate con LPS. In tutti i casi: infiammazione e/o fibrosi aumentate, acetilazione di RELA diminuita. Sembrava proprio che la deacetilazione di RELA K119 fosse un evento cruciale.
Il colpevole smascherato: entra in scena SIRT7
Ok, RELA viene deacetilata. Ma *chi* è il responsabile? Chi toglie quel gruppo acetile? Esistono diverse famiglie di enzimi “deacetilasi”. Abbiamo fatto un po’ di esperimenti usando inibitori specifici. Un inibitore generico delle HDAC (un tipo di deacetilasi) chiamato TSA non sembrava avere un grande effetto sull’acetilazione di RELA. Invece, un inibitore delle sirtuine (un’altra famiglia di deacetilasi) chiamato NAM aumentava significativamente l’acetilazione di RELA. Bingo! Il colpevole doveva essere una sirtuina.
Le sirtuine sono una famiglia di 7 proteine (SIRT1-7). Quale di queste era la nostra “forbice” molecolare? Con esperimenti di co-immunoprecipitazione (una tecnica per vedere quali proteine si “legano” tra loro), abbiamo scoperto che RELA interagiva specificamente con SIRT7, e non con le altre sirtuine (nemmeno SIRT1 e SIRT2, che erano state implicate in studi sui mammiferi). Non solo interagivano, ma quando mettevamo insieme RELA e SIRT7, l’acetilazione di RELA diminuiva! E, cosa fondamentale, se usavamo una versione mutata di RELA che mimava l’acetilazione costante a K119 (K119Q), SIRT7 non riusciva più a diminuirne l’acetilazione. Questo ci ha dato la prova definitiva: SIRT7 deacetila RELA proprio sul sito K119.
Un altro dettaglio importante: SIRT7 si trova principalmente nel nucleo della cellula. RELA, invece, normalmente sta nel citoplasma, ma quando la cellula è sotto stress (come con la dieta HCHFD o l’LPS), si sposta nel nucleo per attivare i geni dell’infiammazione. Abbiamo visto che l’interazione tra RELA e SIRT7 avveniva proprio nel nucleo, ed era più forte quando c’era infiammazione e l’acetilazione di RELA era bassa. Tutto tornava.
Come la deacetilazione scatena il caos
Ma perché questa deacetilazione di K119 è così problematica? Abbiamo fatto altri esperimenti per capirlo. Usando versioni mutate di RELA (una che mimava la deacetilazione, K119R, e una che mimava l’iper-acetilazione, K119Q), abbiamo scoperto che:
- La forma deacetilata (K119R) si legava molto più forte al DNA rispetto alla forma normale (WT) o a quella acetilata (K119Q).
- La forma deacetilata (K119R) era molto più brava ad “accendere” i geni bersaglio di RELA, come quelli per le citochine infiammatorie TNFα e IL-6 (lo abbiamo visto con saggi di luciferasi, che misurano l’attività genica).
- La forma deacetilata (K119R) tendeva a localizzarsi preferenzialmente nel nucleo, mentre quella acetilata (K119Q) preferiva rimanere nel citoplasma.
In pratica, la deacetilazione di K119 da parte di SIRT7 agisce come un turbo per RELA: la fa entrare più facilmente nel nucleo, la fa legare più saldamente al DNA e la rende super-attiva nell’accendere i geni dell’infiammazione. Questo porta a un rilascio massiccio di fattori pro-infiammatori, che a loro volta peggiorano l’infiammazione e accelerano la fibrosi epatica. Un circolo vizioso devastante.
Spegnere l’interruttore: l’inibitore di SIRT7 come possibile soluzione
Se SIRT7 è il problema, possiamo bloccarlo? Abbiamo testato un inibitore farmacologico specifico per SIRT7. I risultati sono stati incredibilmente promettenti!
Nei pesci con infiammazione acuta indotta da LPS, trattare con l’inibitore di SIRT7:
- Ha ridotto i livelli dei geni e delle proteine infiammatorie (TNFα, IL-8, IL-1β, Mmp9).
- Ha ripristinato i livelli normali di acetilazione di RELA (e delle proteine totali).
- Ha impedito a RELA di accumularsi nel nucleo.
E nei pesci con fibrosi cronica indotta dalla dieta HCHFD? Abbiamo nutrito i pesci con la dieta “cattiva” per 8 settimane fino a sviluppare la fibrosi, e poi li abbiamo trattati con l’inibitore di SIRT7 per altre 2 settimane. Risultato?
- Miglioramento dei parametri epatici nel sangue (ALT, AST).
- Riduzione significativa della fibrosi nel fegato (meno collagene, meno α-SMA).
- Diminuzione dei livelli di geni infiammatori e fibrotici.
- Anche qui, ripristino dell’acetilazione di RELA e riduzione della sua presenza nel nucleo.
Importante: l’inibitore di SIRT7 ha ridotto l’infiammazione e la fibrosi senza causare un aumento della morte cellulare nel fegato (apoptosi), suggerendo che questo approccio potrebbe essere relativamente sicuro, modulando finemente l’attività di NF-κB senza spegnerla completamente (cosa che potrebbe essere dannosa).

Cosa significa tutto questo?
Questa scoperta è entusiasmante! Abbiamo identificato un meccanismo completamente nuovo – l’asse SIRT7-RELA K119 – che gioca un ruolo chiave nell’aggravare l’infiammazione e la fibrosi epatica nel persico trota. Abbiamo dimostrato che SIRT7, deacetilando RELA K119 nel nucleo, ne potenzia l’attività pro-infiammatoria. E, cosa più importante, abbiamo visto che bloccare SIRT7 con un inibitore specifico può invertire questi effetti dannosi.
Questo apre nuove strade:
- Potremmo sviluppare nuove terapie per la fibrosi epatica (magari anche umana, data la conservazione del meccanismo?) mirando specificamente a SIRT7 o a ripristinare l’acetilazione di RELA K119.
- SIRT7 o lo stato di acetilazione di RELA K119 potrebbero diventare biomarcatori utili per diagnosticare o monitorare la progressione della malattia.
- Potrebbe aiutarci a sviluppare mangimi migliori o strategie di allevamento per pesci come il persico trota, limitando i danni epatici.
È un esempio perfetto di come lo studio di meccanismi molecolari di base, anche in modelli animali inaspettati, possa portare a intuizioni fondamentali con potenziali applicazioni pratiche enormi. La ricerca continua, ma abbiamo aggiunto un tassello cruciale alla comprensione di questa complessa malattia.
Fonte: Springer