Grano Antico, Difesa Moderna: Svelato il Segreto Anti-Oidio dell’Emmer Lxd-682!
Ciao a tutti, appassionati di scienza e agricoltura! Oggi vi porto con me in un viaggio affascinante nel cuore della genetica del frumento, alla scoperta di come un’antica varietà possa custodire la chiave per difendere una delle colture più importanti al mondo. Parliamo del frumento tenero (Triticum aestivum L.), la base della nostra alimentazione, ma costantemente minacciato da malattie insidiose. Una di queste è l’oidio, o mal bianco, causato dal fungo Blumeria graminis f. sp. tritici (che chiameremo Bgt per semplicità). Questo fungo è un vero trasformista, capace di evolversi rapidamente e mettere a dura prova le nostre coltivazioni.
Come possiamo difendere il nostro grano in modo efficace e sostenibile? La risposta, amici miei, sta nel DNA stesso della pianta: nei geni di resistenza. Identificare questi geni e capire come funzionano è la strategia vincente a lungo termine. Ed è qui che entra in gioco il protagonista della nostra storia: un’accessione di frumento emmer coltivato (Triticum dicoccum), chiamata Lxd-682.
La Star della Resistenza: Lxd-682
Immaginate questa scena: abbiamo testato Lxd-682 contro ben 13 diversi isolati del fungo Bgt. Il risultato? Ha mostrato una resistenza incredibile a 12 di essi, quasi come se avesse uno scudo impenetrabile! Solo un isolato è riuscito a scalfirlo leggermente. Quando lo abbiamo infettato con l’isolato E09, Lxd-682 non ha mostrato alcun sintomo (un punteggio di infezione, IT, pari a 0), mentre il suo “parente” suscettibile, chiamato Langdon (LDN), era completamente ricoperto di muffa (IT 4).
Ma come viene ereditata questa super-resistenza? Incrociando Lxd-682 con Langdon, abbiamo osservato le piante figlie (la generazione F1). Tutte resistenti, proprio come il genitore Lxd-682! Questo ci ha subito suggerito che la resistenza fosse dominante. Analizzando poi la generazione successiva (F2), abbiamo contato 177 piante resistenti e 62 suscettibili. Questo rapporto (circa 3:1) segue perfettamente le leggi di Mendel, indicando che la resistenza è controllata da un singolo gene dominante. Per essere sicuri, abbiamo seguito anche la generazione F2:3, confermando ancora il rapporto atteso (1:2:1 tra famiglie resistenti, segreganti e suscettibili). Abbiamo quindi battezzato provvisoriamente questo gene PmLxd-682.
Abbiamo anche voluto vedere da vicino cosa succede quando il fungo attacca. Osservando al microscopio le foglie infettate, abbiamo notato che fino a 12 ore dopo l’inoculo (hpi), il fungo prova a penetrare sia in Lxd-682 che in Langdon. Ma dopo questo momento critico, le strade si dividono: su Langdon, il fungo prospera, forma strutture per nutrirsi (austori) e crea colonie visibili. Su Lxd-682, invece, la sua avanzata viene bloccata efficacemente. È chiaro che PmLxd-682 entra in azione proprio in questa fase cruciale.

A Caccia del Gene con la Tecnologia BSR-Seq
Ora viene il bello: come trovare l’esatta posizione di PmLxd-682 nel vasto genoma del frumento? Qui entra in gioco una tecnica potentissima chiamata Bulked Segregant RNA Sequencing (BSR-Seq). In pratica, abbiamo preso un gruppo di piante F2:3 sicuramente resistenti e un gruppo di piante sicuramente suscettibili. Abbiamo estratto l’RNA (il messaggero che porta le istruzioni dei geni) da entrambi i gruppi e li abbiamo “mescolati” per creare due “pool”: uno resistente e uno suscettibile.
Sequenziando l’RNA di questi due pool, abbiamo ottenuto un’enorme quantità di dati (miliardi di basi!). Confrontando le sequenze dei due pool, abbiamo cercato le differenze, in particolare piccole variazioni chiamate SNP (Single Nucleotide Polymorphisms). L’idea è che gli SNP vicini al gene di resistenza saranno molto diversi tra il pool resistente e quello suscettibile. Analizzando la distribuzione di queste differenze lungo tutti i cromosomi, abbiamo individuato una singola regione candidata: la parte terminale del braccio lungo del cromosoma 2A (2AL), in un intervallo specifico (tra 748 e 776 Mb). Bingo! Avevamo ristretto il campo di ricerca.
Mappatura e Identificazione: È Proprio Lui, Pm4a!
Avendo individuato la zona “calda”, abbiamo usato dei marcatori molecolari noti per essere presenti in quella regione. I marcatori sono come delle bandierine nel genoma che ci aiutano a orientarci. Abbiamo testato otto marcatori sulla nostra popolazione F2:3 e abbiamo visto che erano tutti strettamente associati alla resistenza. Costruendo una mappa genetica più dettagliata, abbiamo “incastonato” PmLxd-682 tra due marcatori, WGRE77413 e WGRC1096.
La cosa ancora più eccitante è che un altro marcatore, chiamato JS717/JS718, co-segregava perfettamente con PmLxd-682. E sapete cosa? Questo marcatore è noto per essere diagnostico per un famoso gene di resistenza all’oidio chiamato Pm4! Questo ci ha dato un indizio fortissimo: PmLxd-682 doveva essere un allele (una versione) del gene Pm4.
Per la prova definitiva, abbiamo clonato e sequenziato l’intera sequenza del gene omologo a Pm4 presente in Lxd-682. Confrontando questa sequenza con quelle degli alleli di Pm4 già conosciuti (Pm4a, Pm4b, ecc.), abbiamo avuto la conferma: la sequenza in Lxd-682 era identica a Pm4a! Quindi, il nostro gene PmLxd-682 è in realtà l’allele Pm4a. Mistero risolto!
Come Funziona Pm4a? Uno Sguardo all’Espressione Genica
Ma non ci siamo fermati qui. Volevamo capire meglio come Pm4a (o PmLxd-682, se preferite) svolge il suo lavoro. Sappiamo che il gene Pm4 può produrre due versioni leggermente diverse della sua proteina attraverso un processo chiamato “splicing alternativo”. Abbiamo chiamato queste versioni PmLxd-682-V1 e PmLxd-682-V2.
Usando la tecnica qRT-PCR (che misura quanto un gene è “acceso”), abbiamo monitorato l’espressione di V1 e V2 nelle foglie di Lxd-682 dopo l’infezione con Bgt. Entrambe le versioni venivano attivate dall’infezione, ma con dinamiche diverse. La cosa più interessante è che l’espressione di V2 era significativamente più alta di quella di V1 in quasi tutti i momenti analizzati (da 0 a 72 ore dopo l’infezione). Questo suggerisce che PmLxd-682-V2 potrebbe giocare il ruolo più importante nel conferire la resistenza all’oidio in Lxd-682.
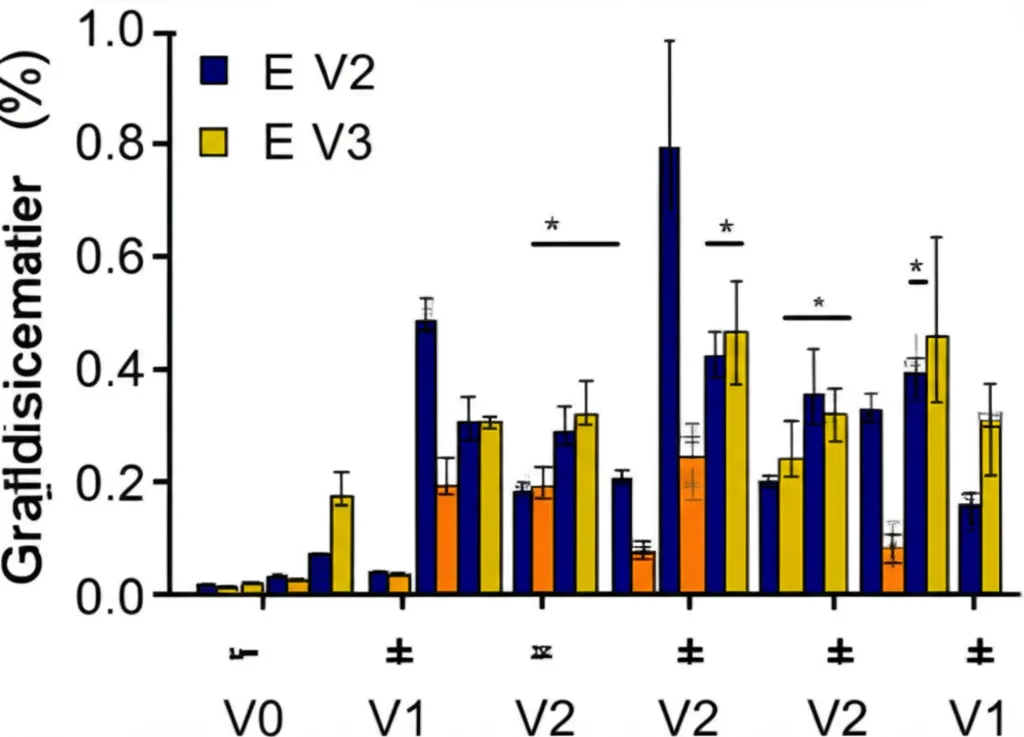
Abbiamo anche dato un’occhiata ad altri geni noti per essere coinvolti nella difesa delle piante, i geni legati alla patogenesi (PR). Abbiamo analizzato l’espressione di quattro di essi (PR1, PR2, PR3, PR10) sia in Lxd-682 che nel suscettibile Langdon. Sorprendentemente, anche se alcuni PR (come PR1 e PR3) erano più attivi in Lxd-682 dopo l’infezione, l’aumento significativo dell’espressione in entrambi i genitori suggerisce che potrebbero non essere i protagonisti principali nella via di resistenza specifica mediata da Pm4a, ma piuttosto parte di una risposta di difesa più generale.
Oltre Pm4a: Alla Ricerca dei Regolatori Nascosti
Grazie alla BSR-Seq, non solo abbiamo trovato Pm4a, ma abbiamo anche identificato ben 1.567 geni che mostravano un’espressione diversa tra il pool resistente e quello suscettibile (li chiamiamo Geni Espressi Differenzialmente, o DEG). Di questi, 1.302 erano meno espressi nel pool resistente e 265 erano più espressi. La cosa interessante è che 490 di questi DEG si trovavano proprio nella regione candidata sul cromosoma 2AL dove risiede Pm4a. Questi 490 geni sono candidati promettenti per essere coinvolti nella risposta all’oidio orchestrata da Pm4a.
Per capire cosa fanno tutti questi geni, abbiamo usato analisi bioinformatiche come GO (Gene Ontology) e KEGG (Kyoto Encyclopedia of Genes and Genomes). Queste analisi ci aiutano a raggruppare i geni in base alle loro funzioni biologiche o alle vie metaboliche in cui sono coinvolti.
- L’analisi GO ha evidenziato l’importanza di processi come il ‘trasporto transmembrana’ (il movimento di sostanze attraverso le membrane cellulari) e la ‘fotosintesi, raccolta della luce nel fotosistema I’. Questo suggerisce che la resistenza potrebbe implicare cambiamenti nel trasporto cellulare e nel metabolismo energetico. È emerso anche un arricchimento per componenti cellulari come la ‘membrana tilacoidale del cloroplasto’, il sito della fotosintesi, che gioca un ruolo anche nelle risposte di difesa.
- L’analisi KEGG ha indicato diverse vie metaboliche e di segnalazione. Una delle più importanti era la “via di segnalazione MAPK”, una cascata di segnali cruciale per molte risposte cellulari, inclusa la difesa dalle malattie.
Per avere una conferma sperimentale, abbiamo selezionato sei DEG promettenti localizzati nella regione di Pm4a e legati alla resistenza alle malattie. Abbiamo misurato la loro espressione dopo l’infezione con Bgt. Tre di questi geni, che codificano per proteine di resistenza note (appartenenti alle classi NBS-LRR e TIR-NBS-LRR), hanno mostrato un aumento dell’espressione in Lxd-682 in alcuni momenti chiave dopo l’infezione. Questi geni (TRITD2Av1G294940, TRITD2Av1G036490 e TRITD2Av1G295220) potrebbero essere attori importanti che collaborano con Pm4a nella difesa.

Implicazioni Pratiche: Marcatori per il Miglioramento Genetico
Tutta questa ricerca non è fine a se stessa! Identificare Pm4a in Lxd-682 e capire un po’ del suo funzionamento ha implicazioni enormi per il miglioramento genetico del frumento. Lxd-682, con la sua resistenza ad ampio spettro, è una risorsa genetica preziosissima.
Inoltre, abbiamo confermato che i marcatori molecolari JS717/JS718 (quello diagnostico per Pm4) e WGRC1096 (uno dei fiancheggianti) funzionano bene per distinguere le piante che portano Pm4a da quelle che non lo portano, anche in diverse varietà di frumento. Questi marcatori possono essere usati nella Selezione Assistita da Marcatori (MAS). Immaginate i breeder che possono analizzare rapidamente il DNA delle piantine e selezionare solo quelle che hanno ereditato il gene Pm4a, senza dover aspettare che le piante crescano e vengano infettate. Questo accelera enormemente la creazione di nuove varietà di frumento resistenti all’oidio.
Certo, rimangono domande aperte. Qual è l’esatto meccanismo molecolare di Pm4a? Come interagisce con altri geni di resistenza? L’introduzione di questo gene dall’emmer al frumento tenero potrebbe avere effetti indesiderati? Sono tutte domande affascinanti per ricerche future.
In conclusione, il nostro viaggio ci ha portato a identificare il gene Pm4a come responsabile della robusta resistenza all’oidio nell’emmer Lxd-682. Abbiamo visto che la sua variante V2 sembra essere particolarmente importante e abbiamo identificato altri geni potenzialmente coinvolti nella sua rete di difesa. Soprattutto, abbiamo fornito strumenti concreti (i marcatori molecolari) per trasferire questa preziosa resistenza nelle future varietà di frumento, contribuendo a un’agricoltura più sostenibile e a garantire la sicurezza alimentare. Non è incredibile come studiando il passato (un grano antico) possiamo costruire un futuro più sicuro?
Fonte: Springer







