Sindrome X Fragile: Il Mistero dei Portatori Sani e il Ruolo Chiave di TET3
Ciao a tutti! Oggi voglio portarvi nel cuore di una ricerca affascinante che tocca un tema complesso ma incredibilmente importante: la Sindrome dell’X Fragile (FXS). È una condizione neuroevolutiva che affligge molte famiglie, e la sua causa è legata a un piccolo “intoppo” nel nostro codice genetico, specificamente nel gene FMR1.
Cos’è la Sindrome dell’X Fragile e il Mistero delle Ripetizioni CGG
Immaginate il gene FMR1 come un’istruzione fondamentale per produrre una proteina chiamata FMRP, essenziale per lo sviluppo del cervello. Nelle persone senza la sindrome, una piccola sezione di questo gene, chiamata ripetizione CGG, è presente in un numero limitato di copie (tra 30 e 55). Il problema sorge quando queste ripetizioni diventano troppe. Se superano le 200 copie (una condizione chiamata “mutazione completa”), scatta un meccanismo biologico chiamato metilazione. Pensate alla metilazione come a un interruttore che spegne il gene FMR1. Quando FMR1 è spento, la proteina FMRP non viene prodotta, e la sua assenza causa i sintomi della FXS, come ritardi nello sviluppo e disturbi dello spettro autistico.
Fin qui, sembra tutto lineare. Ma la biologia ama sorprenderci! Esistono casi rarissimi di persone che hanno più di 200 ripetizioni CGG nel gene FMR1, ma, incredibilmente, il loro gene non viene metilato. Rimane acceso, funzionante. Questi individui sono chiamati portatori di mutazione completa non metilata (UFM – Unmethylated Full Mutation). Sono un vero e proprio enigma scientifico: perché loro sfuggono al “silenziamento” del gene che colpisce quasi tutti gli altri con lo stesso numero di ripetizioni? Cosa li rende diversi?
Questa domanda è cruciale, perché capire perché i portatori UFM non metilano il gene potrebbe svelare i meccanismi esatti che portano alla FXS e, chissà, aprire nuove strade terapeutiche. La cosa si fa ancora più interessante se pensiamo che nei modelli animali, come i topi geneticamente modificati per avere l’espansione CGG, la metilazione spesso non avviene come negli umani. Questo suggerisce che non basta solo l’espansione della ripetizione; deve esserci qualcos’altro, un fattore specifico, forse presente negli umani ma non nei topi, o forse mancante proprio nei portatori UFM.
Alla Ricerca del Fattore Mancante: Lo Studio sui Portatori UFM
Ed è qui che entra in gioco lo studio di cui vi parlo oggi. Un gruppo di ricercatori ha avuto la rara opportunità di studiare le cellule (fibroblasti, per la precisione) di due portatori UFM non imparentati tra loro, confrontandole con quelle di un paziente con FXS classica (con gene metilato) e di un controllo sano. L’obiettivo? Scovare le differenze a livello molecolare che potessero spiegare perché i portatori UFM sono “protetti” dalla metilazione.
Per farlo, hanno usato tecniche potentissime:
- Analisi Trascrittomica (RNA-seq): Hanno “letto” quali geni erano attivi (trascritti in RNA) e in che quantità in ciascun tipo di cellula. È come guardare quali “programmi” stanno girando nel computer cellulare.
- Sequenziamento dell’Intero Genoma (WGS): Hanno analizzato l’intero DNA dei due portatori UFM per cercare varianti genetiche particolari che potessero influenzare l’espressione di geni chiave.
Si sono concentrati in particolare sui geni coinvolti nella modifica epigenetica, cioè quei processi, come la metilazione, che regolano l’attività dei geni senza cambiarne la sequenza di DNA.

Risultati Sorprendenti: FMR1 Attivo e un Nuovo Attore, TET3
Prima di tutto, l’analisi ha confermato quello che già si sospettava: nei portatori UFM, il gene FMR1 è effettivamente trascritto, a livelli persino superiori rispetto ai controlli sani (anche se, paradossalmente, i livelli della proteina FMRP possono essere più bassi, un altro aspetto complesso di questa condizione). Nelle cellule del paziente FXS, invece, come previsto, il gene FMR1 era completamente silente.
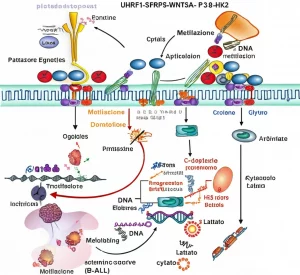
Ma la vera sorpresa è arrivata analizzando i geni “regolatori”. Tra i 44 geni candidati legati all’epigenetica, uno in particolare ha catturato l’attenzione: il gene TET3 (Tet methylcytosine dioxygenase 3). Cosa fa TET3? È un enzima coinvolto proprio nei processi di metilazione e, più specificamente, nella sua rimozione o modifica (tramite un processo chiamato idrossimetilazione). Ebbene, nei fibroblasti di entrambi i portatori UFM, i livelli di espressione di TET3 erano significativamente più bassi – circa l’80% in meno – rispetto sia al controllo sano sia al paziente FXS!
Questa è una scoperta potenzialmente enorme. TET3 ha la capacità di legarsi a sequenze di DNA ricche di CG (proprio come le ripetizioni CGG!) e gioca un ruolo nel definire lo stato di metilazione. Che la sua espressione sia così ridotta proprio negli individui che non metilano l’espansione FMR1 suggerisce fortemente un suo coinvolgimento diretto nel meccanismo.
TET3 è la Chiave? Indizi Genetici e Differenze tra Uomo e Topo
Ovviamente, la domanda successiva è: perché TET3 è meno espresso nei portatori UFM? I ricercatori hanno cercato nel loro genoma delle varianti specifiche nel gene TET3 o nelle sue vicinanze che potessero spiegarlo. Hanno trovato alcune varianti rare condivise dai due portatori UFM. Nessuna sembrava essere una mutazione “distruttiva”, ma una di queste (rs72818011) si trova in una regione del gene (la 3’UTR) che può influenzare la stabilità dell’RNA messaggero di TET3. È possibile che questa variante renda l’RNA di TET3 meno stabile, portando a una minore produzione della proteina? È un’ipotesi intrigante, ma servono ulteriori studi per confermarla. Non si può escludere che la vera causa sia altrove nel genoma.

Un altro pezzo del puzzle viene dal confronto tra uomo e topo. Abbiamo detto che i topi con l’espansione CGG spesso non metilano il gene FMR1. I ricercatori hanno confrontato l’espressione di TET3 durante lo sviluppo cerebrale nelle due specie. Mentre la proteina TET3 è molto simile, i suoi livelli di espressione seguono pattern temporali diversi. Nell’uomo, c’è un picco di espressione di TET3 proprio nel periodo dello sviluppo embrionale (tra 80 e 120 giorni post-concepimento) in cui si pensa avvenga la metilazione del FMR1 nella FXS. Nei topi, questo picco specifico manca. Questa differenza nei tempi di “accensione” di TET3 potrebbe essere un’altra ragione per cui i modelli murini non replicano perfettamente la condizione umana e rafforza l’idea che TET3 sia cruciale nel processo di metilazione dell’FMR1 umano.
Un Possibile Modello: Come Funziona (o Non Funziona) TET3?
Ma come potrebbe agire TET3? Il suo ruolo nella metilazione è complesso e, a tratti, ambiguo. Tradizionalmente, gli enzimi TET sono visti come “cancellatori” di metilazione, convertendo la 5-metilcitosina (5mC, il segno della metilazione) in 5-idrossimetilcitosina (5hmC), un primo passo verso la demetilazione. Tuttavia, studi recenti suggeriscono che TET3 potrebbe avere anche un ruolo nel promuovere la metilazione in specifiche regioni del genoma, forse regolando altri enzimi chiave come DNMT1, la macchina principale della metilazione. Inoltre, la 5hmC stessa non è solo uno stato transitorio, ma un marchio epigenetico stabile, particolarmente abbondante nel cervello e trovato anche vicino alle ripetizioni CGG nei neuroni di pazienti FXS.
Quindi, ci sono due ipotesi principali (che non si escludono a vicenda) su come bassi livelli di TET3 nei portatori UFM potrebbero prevenire la metilazione dell’FMR1:
- Meno 5hmC “iniziale”: Forse TET3 è necessario per stabilire un certo livello di 5hmC sull’espansione CGG, che poi in qualche modo innesca la metilazione completa (5mC). Con poco TET3, questo processo non parte.
- Meno “benzina” per la metilazione: Forse TET3, anche indipendentemente dalla sua attività catalitica, è necessario per regolare l’enzima DNMT1. Bassi livelli di TET3 porterebbero a bassi livelli di DNMT1 attivo nel posto giusto al momento giusto, impedendo la metilazione dell’espansione CGG.
In entrambi i casi, la ridotta espressione di TET3 osservata nei portatori UFM si adatterebbe perfettamente a questo quadro.

Cosa Significa Tutto Questo? Prospettive Future
Questo studio, pur basandosi su un numero limitato di casi (data la rarità dei portatori UFM), è estremamente importante. Identifica TET3 come un candidato fortissimo per essere quel “fattore” elusivo che determina se un’espansione CGG nel gene FMR1 verrà metilata o meno.
Certo, siamo ancora nel campo delle associazioni e delle ipotesi. Serviranno molte altre ricerche per confermare il ruolo causale di TET3, per capire esattamente come agisce e per verificare se la variante genetica identificata nella 3’UTR è davvero responsabile della sua ridotta espressione. Studiare questi meccanismi è complicato anche dalla natura dinamica della metilazione e dall’instabilità delle ripetizioni CGG nelle colture cellulari.
Tuttavia, aver puntato i riflettori su TET3 apre scenari entusiasmanti. Capire a fondo come viene regolata la metilazione dell’FMR1 non solo ci avvicina alla comprensione della patogenesi della Sindrome dell’X Fragile, ma potrebbe, in futuro, ispirare nuove strategie terapeutiche mirate a prevenire o persino a invertire il silenziamento del gene FMR1.
È un esempio perfetto di come lo studio di casi umani “eccezionali”, come i portatori UFM, possa illuminare meccanismi biologici fondamentali validi per molti. La scienza procede a piccoli passi, ma scoperte come questa ci ricordano quanto sia affascinante e pieno di potenzialità il viaggio nell’esplorazione del nostro genoma. Continueremo a seguire gli sviluppi!
Fonte: Springer