Reti Geniche Su Misura: Decifriamo i Segreti del Tumore al Polmone, Paziente per Paziente!
Ciao a tutti! Oggi voglio portarvi nel cuore di una sfida enorme ma super affascinante: quella contro il cancro al polmone, in particolare l’adenocarcinoma polmonare (LUAD). Sapete, questa forma di tumore è un vero e proprio camaleonte, diversissimo da paziente a paziente. E questa sua eterogeneità lo rende particolarmente aggressivo e difficile da trattare. Per anni, noi ricercatori abbiamo cercato di capire i meccanismi che lo governano, e una delle chiavi sembra risiedere nel modo in cui i nostri geni “parlano” tra loro.
Le Reti di Co-espressione Genica: Una Mappa del Dialogo tra Geni
Immaginate i geni come persone in una stanza: alcuni parlano spesso tra loro, altri meno. Le reti di co-espressione genica (GCN) sono un po’ come una mappa di queste conversazioni. Ci mostrano quali geni tendono ad attivarsi (o disattivarsi) insieme, suggerendo che collaborano in qualche processo biologico. Queste mappe sono state fondamentali per capire le differenze tra tessuti sani e malati nel LUAD. Pensate, studi recenti hanno addirittura suggerito che molti tipi di cancro condividono una sorta di “architettura di rete” distintiva. Questo ci fa pensare che la topologia, cioè la forma e la struttura di queste reti, possa svelarci molto sulla patologia.
Però, c’è un “ma”. I metodi tradizionali per costruire queste reti tendono a fare una media tra tanti pazienti. È come se, per capire le conversazioni in diverse stanze, ascoltassimo tutte insieme e cercassimo di trarne un unico discorso. Così facendo, però, perdiamo le sfumature, le specificità di ogni singola “stanza”, ovvero di ogni singolo paziente. E nel cancro, questa eterogeneità è cruciale!
La Svolta: Le Reti Paziente-Specifiche (SSN)
Ed è qui che entra in gioco la nostra idea: e se potessimo creare una mappa genica unica per ogni paziente? È il concetto delle reti a singolo campione (SSN). Noi abbiamo usato un metodo chiamato LIONESS (che sta per Linear Interpolation to Obtain Network Estimates for Single Samples, un nome un po’ lungo, lo so!) per ricostruire ben 334 reti di co-espressione genica paziente-specifiche. Come “linguaggio” per capire le connessioni tra geni, abbiamo usato l’informazione mutua, una misura statistica molto potente.
L’equazione LIONESS, in pratica, confronta una rete costruita su tutti i campioni con una rete costruita escludendo un singolo campione. Questo ci permette di stimare il contributo di quel campione specifico alla rete aggregata e, alla fine, di “estrarre” la sua rete individuale. È un po’ come isolare la voce di una singola persona in un coro. Un lavoro certosino, soprattutto considerando la complessità del calcolo dell’informazione mutua su larga scala, ma ne è valsa la pena!
Prima Scoperta: Nuovi Sottotipi di LUAD Nascosti nelle Reti
Una volta ottenute queste reti personalizzate, ci siamo chiesti: pazienti con reti simili avranno anche destini clinici simili? Abbiamo quindi raggruppato i pazienti in base alla somiglianza delle loro reti. E sorpresa! Sono emersi sei nuovi sottotipi di LUAD, ognuno con “motivi di rete” distinti che riflettono programmi biologici unici. È stato come scoprire sei diverse “impronte digitali” molecolari del tumore.
Ad esempio, il Cluster 1 mostrava una forte associazione con processi legati al movimento e alla struttura delle ciglia cellulari. Le cellule ciliate sono predominanti nelle vie aeree e sono considerate molto differenziate. È stato ipotizzato che possano formare lesioni precancerose. Questo ci suggerisce che i tumori di questo cluster potrebbero rappresentare uno stadio più precoce della malattia. Il Cluster 5, invece, era arricchito in pazienti di sesso femminile e con tumori di piccole dimensioni (T1), e le sue reti erano piene di geni coinvolti nel ciclo cellulare. Sappiamo che ci sono differenze di genere nello sviluppo del cancro al polmone, e questo cluster potrebbe riflettere l’impatto di fattori ormonali o genetici sulla proliferazione tumorale nelle donne.
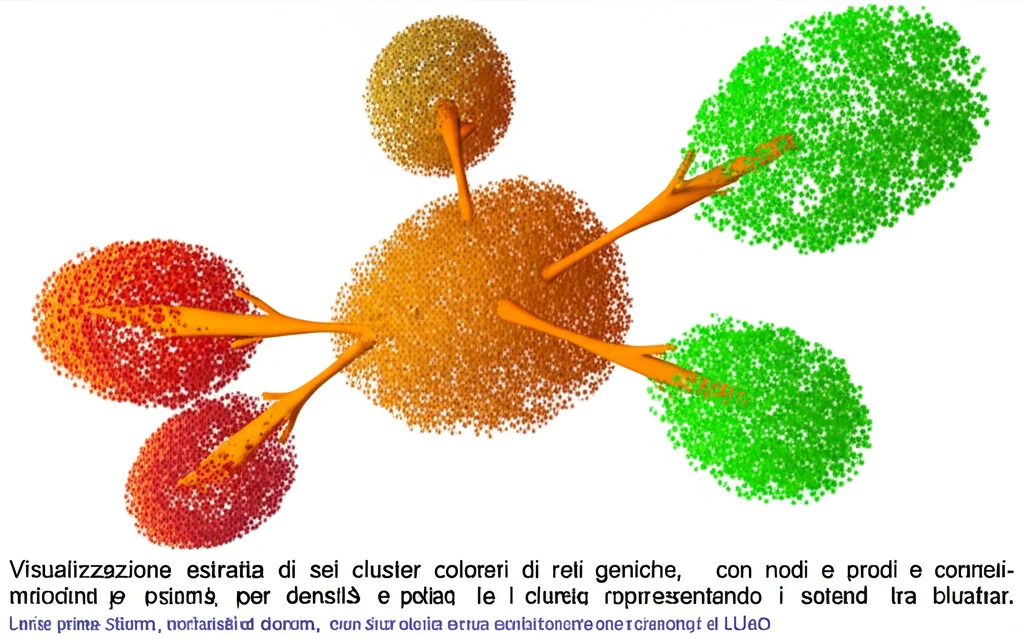
Curiosamente, le caratteristiche cliniche tradizionali (come lo stadio del tumore o il coinvolgimento linfonodale) non erano il motore principale di questa suddivisione, anche se abbiamo trovato alcune associazioni significative, come l’arricchimento di tumori T1 (piccoli e localizzati) nel Cluster 1 e 5, e di pazienti di sesso femminile nel Cluster 5. Questo ci dice che le reti paziente-specifiche colgono informazioni che vanno oltre quello che vediamo con gli esami classici.
Un Focus sull’Ambiente Tumorale: Immunità e Stroma
Due cluster, il Cluster 3 e il Cluster 6, condividevano molti processi biologici legati alla risposta immunitaria, nonostante le loro reti fossero diverse. Questo ci ha incuriosito, e siamo andati a vedere la composizione cellulare dei tumori. Abbiamo scoperto che il Cluster 3 era caratterizzato da una scarsa infiltrazione di cellule immunitarie e stromali (il tessuto di supporto del tumore), mentre il Cluster 6 era l’opposto, con un’alta infiltrazione immunitaria. È come se avessimo identificato tumori “freddi” (poco immuni) e “caldi” (molto immuni) basandoci sulla struttura delle loro reti geniche. Questo è importantissimo, perché l’interazione tra tumore e sistema immunitario è un campo di battaglia cruciale per lo sviluppo di nuove terapie. Identificare questi pattern a livello di rete potrebbe aiutarci a capire meglio come il tumore elude le nostre difese.
È interessante notare che, analizzando i cluster derivati dalla sola espressione genica (il metodo più tradizionale), si osservavano variazioni nell’infiltrazione immunitaria, ma i cluster basati sulle SSN sembravano più sensibili nel catturare gruppi con estremi di infiltrazione (molto bassa vs molto alta). Questo suggerisce che l’analisi delle co-variazioni geniche tramite SSN aggiunge un livello di dettaglio in più.
Seconda Scoperta: Biomarcatori Predittivi per la Sopravvivenza
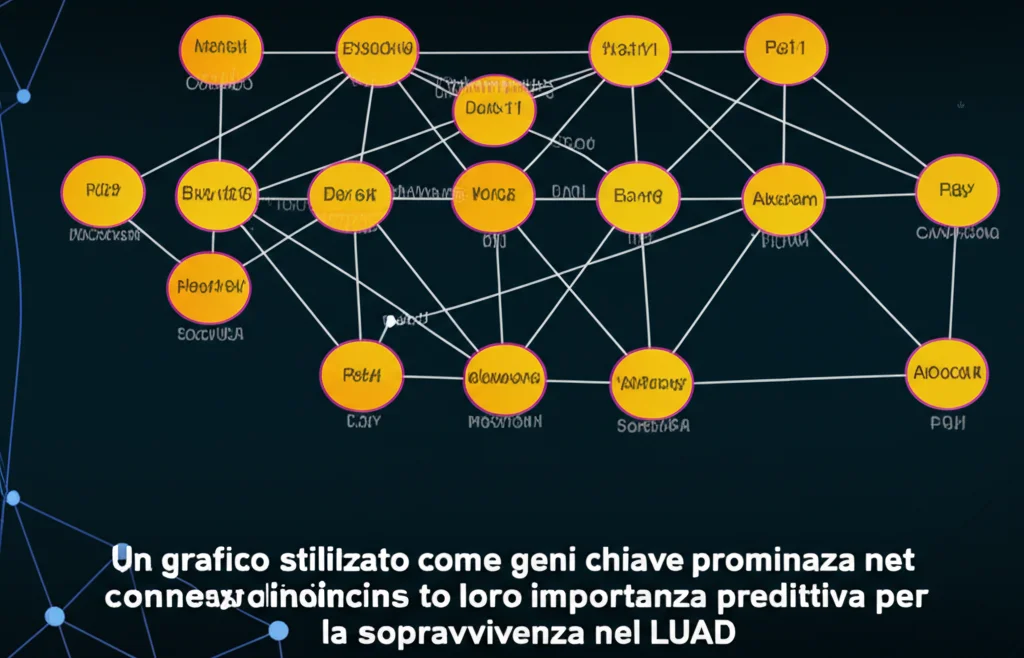
Ma non ci siamo fermati qui. Ci siamo chiesti: la struttura di queste reti individuali può predire quanto a lungo vivrà un paziente? Per rispondere, abbiamo calcolato il “grado pesato” di ogni gene in ogni rete. Immaginatelo come una misura dell’importanza strutturale di un gene all’interno della sua rete specifica: più connessioni pesanti ha, più è “centrale”.
Abbiamo poi usato questi gradi pesati come input per un modello di regressione (una tecnica statistica avanzata chiamata regressione di Cox regolarizzata) per predire la sopravvivenza dei pazienti. E i risultati sono stati entusiasmanti! Non solo i gradi pesati si sono rivelati più predittivi rispetto ai dati di espressione genica da soli, ma abbiamo anche identificato 12 geni la cui importanza strutturale nelle reti individuali è robustamente associata alla sopravvivenza. Questi geni sono: CHRDL2, SPP2, VAC14, IRF5, GUCY1B1, NCS1, RRM2B, EIF5A2, CCDC62, CTCFL, XG e TP53INP2.
- CHRDL2 (Chordin Like 2)
- SPP2 (Secreted Phosphoprotein 2)
- VAC14 (VAC14 Component of PIKFYVE Complex)
- IRF5 (Interferon Regulatory Factor 5)
- GUCY1B1 (Guanylate Cyclase 1 Soluble Subunit Beta 1)
- NCS1 (Neuronal Calcium Sensor 1)
- RRM2B (Ribonucleotide Reductase Regulatory Subunit M2B)
- EIF5A2 (Eukaryotic Translation Initiation Factor 5A2)
- CCDC62 (Coiled-Coil Domain Containing 62)
- CTCFL (CCCTC-Binding Factor Like)
- XG (Xg Blood Group)
- TP53INP2 (Tumor Protein P53 Inducible Nuclear Protein 2)
Molti di questi geni erano già noti per essere coinvolti in vari tipi di cancro, e sette di essi avevano già segnalazioni specifiche per il cancro al polmone. Ad esempio, CHRDL2 e SPP2, i due predittori più stabili, sono antagonisti delle Proteine Morfogenetiche Ossee (BMP), che fanno parte della superfamiglia del TGF-β, un importante regolatore della crescita cellulare. Alterazioni in questa via di segnalazione sono state collegate alla progressione tumorale in molti contesti, incluso il LUAD. La nostra analisi suggerisce che l’importanza di questi geni all’interno della rete di un paziente riflette la complessità con cui queste vie di segnalazione influenzano l’esito clinico nel LUAD.
Cosa Significa Tutto Questo e Dove Andiamo Ora?
Il nostro lavoro dimostra che guardare alle reti geniche a livello del singolo paziente ci apre un mondo di informazioni. Abbiamo identificato nuovi sottotipi di LUAD che non emergono con le analisi tradizionali e abbiamo trovato firme di rete che possono predire la sopravvivenza. Questo è un passo avanti importante perché ci avvicina a una medicina più personalizzata: se capiamo meglio la “rete” specifica del tumore di un paziente, potremmo un giorno scegliere terapie più mirate.
Certo, la strada è ancora lunga. Queste analisi sono state fatte su una coorte specifica (TCGA-LUAD), e sarà fondamentale validare i nostri risultati su altri set di dati. Inoltre, costruire queste reti con l’informazione mutua è computazionalmente intensivo, quindi dovremo pensare a strategie efficienti per studi più ampi. Ma la direzione è chiara: le reti paziente-specifiche sono uno strumento potentissimo per svelare l’eterogeneità del cancro e per guidare la ricerca futura nel campo della medicina di rete e dell’oncologia polmonare.
Crediamo davvero che questo approccio possa contribuire significativamente a quel grande puzzle che è la comprensione del cancro, aiutandoci a trovare nuovi modi per diagnosticare precocemente, trattare più efficacemente e, speriamo, migliorare la vita di tanti pazienti.
Fonte: Springer