Virus al Microscopio? No, con lo Smartphone! L’AI Rivoluziona l’Analisi delle Placche Virali
Ciao a tutti! Oggi voglio parlarvi di qualcosa che potrebbe sembrare uscito da un film di fantascienza, ma che sta diventando realtà nei laboratori di ricerca: usare l’intelligenza artificiale e le foto scattate con un semplice smartphone per studiare i virus. Sì, avete capito bene! Niente più ore infinite passate a contare manualmente minuscole “macchie” al microscopio. Sembra incredibile, vero? Lasciate che vi racconti come ci stiamo riuscendo.
Il “vecchio” modo: il saggio delle placche
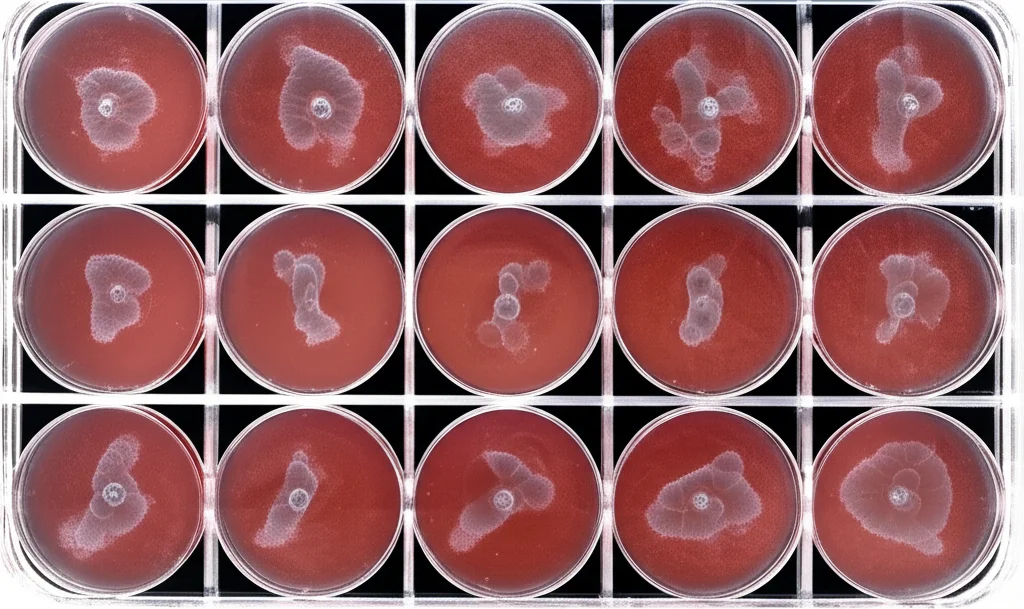
Prima di tuffarci nel futuro, facciamo un passo indietro. In virologia, uno dei metodi fondamentali per capire quanti virus infettivi ci sono in un campione (e come si comportano) è il cosiddetto saggio delle placche virali. Immaginate di avere una piastra con uno strato di cellule ospiti belle sane. Aggiungiamo il nostro campione di virus (opportunamente diluito) e aspettiamo un po’, di solito un paio di giorni. Dove un singolo virus infetta una cellula, inizia a replicarsi e a diffondersi alle cellule vicine, creando una piccola area di distruzione cellulare. Questa area, una volta colorata, appare come una macchiolina visibile, chiamata appunto “placca”. Contando queste placche, possiamo stimare la quantità di virus infettivo nel campione originale.
Questo metodo è un pilastro della ricerca virologica da quasi un secolo! È fondamentale non solo per contare i virus, ma anche per studiarne il ciclo vitale, come si diffondono e persino per testare farmaci antivirali. Pensate al vaiolo: il virus Variola, un tipo di poxvirus, ha devastato l’umanità per secoli prima di essere eradicato proprio grazie a ricerche che si basavano anche su tecniche come questa. E ancora oggi, con nuove minacce come il recente focolaio di monkeypox (vaiolo delle scimmie), la ricerca sui poxvirus, come il Vaccinia Virus (VACV) che abbiamo usato noi, è più importante che mai.
La sfida: tempo, fatica e… occhi stanchi!
Qual è il problema, allora? Beh, tradizionalmente, questo conteggio viene fatto manualmente. Un ricercatore si mette lì, con la sua piastra colorata, e conta una per una le placche. Immaginate di doverlo fare per decine di piastre al giorno… è un lavoro lungo, faticoso e, diciamocelo, anche un po’ noioso, oltre che soggetto a errori umani.
Certo, negli anni sono state proposte modernizzazioni, come l’uso di microscopi avanzati o scanner specifici. Ma queste soluzioni spesso richiedono attrezzature costose e personale specializzato, e possono essere complicate da gestire in ambienti con alti livelli di biosicurezza (necessari quando si lavora con certi virus). Per questo, il metodo classico, con colorazione e conteggio manuale, è ancora diffusissimo per la sua semplicità. Ma se potessimo renderlo più smart?
La rivoluzione: Deep Learning e foto dal cellulare
Ed è qui che entra in gioco la magia del Deep Learning (DL), una branca dell’intelligenza artificiale specializzata nell’analisi di dati complessi come le immagini, e la potenza della fotografia digitale, ormai alla portata di tutti grazie agli smartphone. L’idea è semplice ma potente: e se potessimo “insegnare” a un computer a riconoscere e contare le placche virali direttamente dalle foto delle piastre?
Per fare questo, però, serve una cosa fondamentale: dati. Tanti dati. E non solo le immagini, ma anche le “annotazioni”, cioè indicazioni precise su dove si trovano le placche (e i pozzetti della piastra) in ogni foto. È un lavoro certosino, ma necessario per addestrare gli algoritmi di DL.
Vi presento VACVPlaque: il nostro dataset “open”
Ecco perché abbiamo creato e reso pubblico VACVPlaque, un nuovo dataset di fotografie digitali di saggi di placche del virus Vaccinia (VACV). Abbiamo raccolto 211 foto di piastre a 6 pozzetti, scattate in condizioni realistiche di laboratorio (luce ambientale, diverse angolazioni) usando due comuni smartphone (un iPhone 6 e uno Xiaomi Mi A1). Poi, armati di pazienza e strumenti software, abbiamo annotato manualmente la posizione esatta di ogni pozzetto e di ogni singola placca virale in tutte le immagini.
Perché renderlo “open”, cioè accessibile a tutti? Perché crediamo che la condivisione dei dati sia cruciale per accelerare il progresso scientifico. Mettere a disposizione questo dataset permetterà ad altri ricercatori di sviluppare e testare nuovi algoritmi di DL per l’analisi delle placche, non solo per il Vaccinia ma potenzialmente per molti altri virus. La mancanza di grandi dataset annotati è spesso un ostacolo all’adozione del DL in biologia infettiva, e speriamo che VACVPlaque possa contribuire a superarlo.
Come “vede” l’AI: StarDist e la nostra HydraStarDist
Ok, abbiamo le foto e le annotazioni. Come fa l’AI a trovare le placche? Abbiamo usato un’architettura di DL chiamata StarDist, che è particolarmente brava nella “segmentazione di istanze” (instance segmentation), cioè nell’identificare e delineare i contorni di singoli oggetti in un’immagine. StarDist è ottima per oggetti con forme tendenzialmente rotonde o “a stella” (star-convex), proprio come le nostre placche (e i pozzetti delle piastre).
Normalmente, per identificare sia i pozzetti che le placche al loro interno, avremmo dovuto addestrare due modelli StarDist separati: uno per i pozzetti e uno per le placche. Ma noi abbiamo pensato: perché non fare tutto in un colpo solo? Così abbiamo proposto una modifica all’architettura originale, che abbiamo battezzato HydraStarDist (HSD). Immaginate un’Idra con un corpo comune (l’encoder, che analizza l’immagine) e due teste (i decoder), una specializzata nel trovare i pozzetti e l’altra nel trovare le placche. Questo approccio “single-shot” non solo è più efficiente, ma aiuta anche il modello a capire che le placche devono trovarsi *dentro* i pozzetti, riducendo gli errori.

Funziona davvero? I risultati dei test
Abbiamo messo alla prova sia StarDist (nella versione a due modelli separati) che la nostra HydraStarDist sul dataset VACVPlaque. I risultati? Davvero incoraggianti! Entrambe le architetture sono riuscite a identificare e segmentare pozzetti e placche con buona precisione.
Certo, la versione “specializzata” a due modelli di StarDist è risultata leggermente più performante nel trovare tutte le placche, mentre HSD ne ha mancata qualcuna in più. Tuttavia, le differenze, specialmente per i pozzetti, sono minime. E il vantaggio di HSD è poter fare tutto in un unico passaggio. Questo dimostra non solo che il nostro dataset è ottimo per addestrare modelli di DL, ma anche che l’analisi completa delle placche potrebbe essere fattibile in modo efficiente e integrato.
Abbiamo anche fatto dei test aggiuntivi (chiamati “ablation studies”) per vedere come i modelli si comportano cambiando la luminosità delle foto o simulando uno zoom diverso, per mimare condizioni di scatto variabili. Anche in questi casi, le performance sono rimaste abbastanza stabili, suggerendo che l’approccio è robusto.
Perché tutto questo è importante?
Potreste chiedervi: “Ok, figo, ma a che serve?”. Beh, i vantaggi sono enormi:
- Velocità ed Efficienza: Analizzare una foto richiede pochi secondi per un computer, contro minuti o ore per un umano.
- Oggettività e Riproducibilità: L’AI non si stanca e applica sempre gli stessi criteri, riducendo la variabilità tra diverse analisi o diversi operatori.
- Accessibilità: Usare uno smartphone e algoritmi open-source rende questa tecnologia potenzialmente accessibile a molti più laboratori, anche con risorse limitate.
- Analisi più approfondite: Oltre al conteggio, la segmentazione precisa permette di misurare la dimensione e la forma delle placche, fornendo informazioni aggiuntive sulla biologia del virus.

Certo, siamo ancora all’inizio. Il nostro dataset riguarda specificamente il Vaccinia Virus. Tuttavia, dato che i ricercatori cercano di ottenere placche con caratteristiche simili (ben definite, circolari) anche per altri virus, pensiamo che questi metodi possano essere adattati e applicati più ampiamente, magari con qualche piccolo aggiustamento o riaddestramento del modello.
Il futuro è adesso (o quasi)
Il saggio delle placche, nonostante la sua veneranda età, resta uno strumento essenziale. Ma grazie ai progressi in Computer Vision e Deep Learning, possiamo finalmente dargli una “rinfrescata” tecnologica, rendendolo più veloce, accurato e accessibile. Il nostro dataset VACVPlaque e l’architettura HydraStarDist sono un passo in questa direzione.
È affascinante pensare a come tecnologie che usiamo tutti i giorni, come la fotocamera del nostro telefono, possano combinarsi con algoritmi intelligenti per aiutarci a combattere le malattie e a capire meglio il complesso mondo dei virus. Il futuro della virologia potrebbe essere letteralmente a portata di click!
Fonte: Springer