Staphylococcus Aureus: Ho Progettato un Vaccino al Computer Contro Questo Superbatterio!
Ciao a tutti! Oggi voglio raccontarvi un’avventura scientifica che mi ha tenuto incollato allo schermo del computer, un viaggio nel mondo microscopico dei batteri e delle molecole, alla ricerca di una nuova arma contro un nemico davvero ostico: lo *Staphylococcus aureus*. Sì, proprio lui, quel batterio che spesso sentiamo nominare in relazione a infezioni ospedaliere e resistenze agli antibiotici. Sviluppare strategie per combatterlo è una priorità assoluta, ce lo dice anche l’Organizzazione Mondiale della Sanità!
Il Nemico Invisibile: Staphylococcus Aureus e la Sfida dell’Antibiotico-Resistenza
Immaginate un batterio incredibilmente versatile, capace di causare dalle più banali infezioni della pelle a malattie gravissime come polmoniti, endocarditi o sepsi. Ecco, questo è *S. aureus*. Un problema nel problema è la sua crescente capacità di resistere agli antibiotici. Ricordate il famoso MRSA (Methicillin-Resistant *Staphylococcus aureus*)? Nato negli ospedali, ora si trova anche nella comunità, colpendo persone apparentemente sane. E anche l’antibiotico di ultima linea, la vancomicina, inizia a mostrare qualche crepa nella sua efficacia.
Un campo di battaglia particolarmente critico sono le ulcere del piede diabetico. Qui, *S. aureus* è spesso il principale colpevole delle infezioni, peggiorando la situazione e aumentando il rischio di amputazioni e conseguenze gravi. Gli antibiotici faticano, le terapie tradizionali non sempre bastano. C’è un bisogno disperato di nuove soluzioni. E se una di queste fosse un vaccino?
LukED: L’Arma Segreta di S. aureus
Nella mia ricerca, mi sono concentrato su una delle “armi” più potenti dello *Staphylococcus aureus*, una tossina chiamata Leukotoxin ED, o più semplicemente LukED. Non è una tossina qualsiasi. È composta da due parti (LukE e LukD) che insieme formano dei pori nelle membrane delle nostre cellule immunitarie. Avete capito bene: uccide i soldati del nostro sistema immunitario! Neutrofili, linfociti T, macrofagi, cellule dendritiche… LukED li prende di mira legandosi a specifici recettori (CCR5, CXCR1, CXCR2) presenti sulla loro superficie. In pratica, disarma le nostre difese, sia quelle innate che quelle adattative, spianando la strada all’infezione.
Ho pensato: e se potessimo creare un vaccino che insegni al nostro corpo a produrre anticorpi proprio contro LukED? Questi anticorpi potrebbero neutralizzare la tossina prima che faccia danni, impedendole di legarsi ai suoi recettori e di uccidere le cellule immunitarie. Bloccare questa via di fuga del batterio potrebbe ridare forza al nostro sistema immunitario per combattere l’infezione. Una scommessa affascinante, no?
La Progettazione al Computer: Nasce un Vaccino Multi-Epitopo
Ma come si progetta un vaccino del genere? Sviluppare vaccini usando proteine intere può portare a problemi di sicurezza o scarsa efficacia. Per questo, ho scelto un approccio più “chirurgico”: la progettazione *in silico*, ovvero al computer, di un vaccino multi-epitopo. Cosa significa? Invece di usare tutta la tossina LukED, ho cercato i suoi “punti deboli” più importanti dal punto di vista immunitario: piccoli frammenti chiamati epitopi. Sono queste le sequenze che vengono riconosciute dal nostro sistema immunitario (in particolare dai linfociti B per produrre anticorpi e dai linfociti T helper, o HTL, per coordinare la risposta).
Il primo passo è stato recuperare le sequenze delle proteine LukE e LukD da database pubblici come UniProt. Poi, per assicurarmi che il vaccino potesse funzionare contro tanti ceppi diversi di *S. aureus*, ho analizzato migliaia di sequenze di LukE e LukD provenienti da isolati differenti, cercando le regioni più conservate, quelle che cambiano poco o nulla tra un ceppo e l’altro.
Armato di potenti strumenti bioinformatici (come BepiPred, bcepred, ElliPro per gli epitopi B; IEDB per gli epitopi HTL), ho iniziato la caccia agli epitopi promettenti. Ma non bastava trovarli! Dovevano superare una serie di test virtuali:
- Conservazione: Essere presenti in regioni conservate (>90%) tra i vari ceppi.
- Immunogenicità: Essere capaci di stimolare una risposta immunitaria (legarsi bene alle molecole HLA umane, che presentano gli epitopi ai linfociti T).
- Sicurezza: Non essere allergenici, non essere tossici e, importantissimo, non assomigliare a proteine umane per evitare reazioni autoimmuni (ho usato BLASTp per confrontarli con il proteoma umano).
Alla fine di questa selezione rigorosa, ho identificato cinque epitopi B (quattro da LukD, uno da LukE) e quattro epitopi HTL principali (tutti da LukE, capaci di legarsi a diverse molecole HLA-II comuni nella popolazione mondiale). Curiosamente, per LukD non ho trovato epitopi T che soddisfacessero tutti i criteri.
Dagli Epitopi alle Regioni: Un Cambio di Strategia Cruciale
Inizialmente, ho provato a costruire il vaccino semplicemente “cucendo” insieme questi epitopi selezionati, usando dei “distanziatori” molecolari chiamati linker (come GPGPG, KK, EAAAK). Ho anche aggiunto un adiuvante, una molecola che dà una spinta in più al sistema immunitario. Ho scelto la beta-defensina umana 3 (hBD3), nota per le sue proprietà immunostimolanti e antimicrobiche. Ho creato ben 17 possibili “architetture” del vaccino.
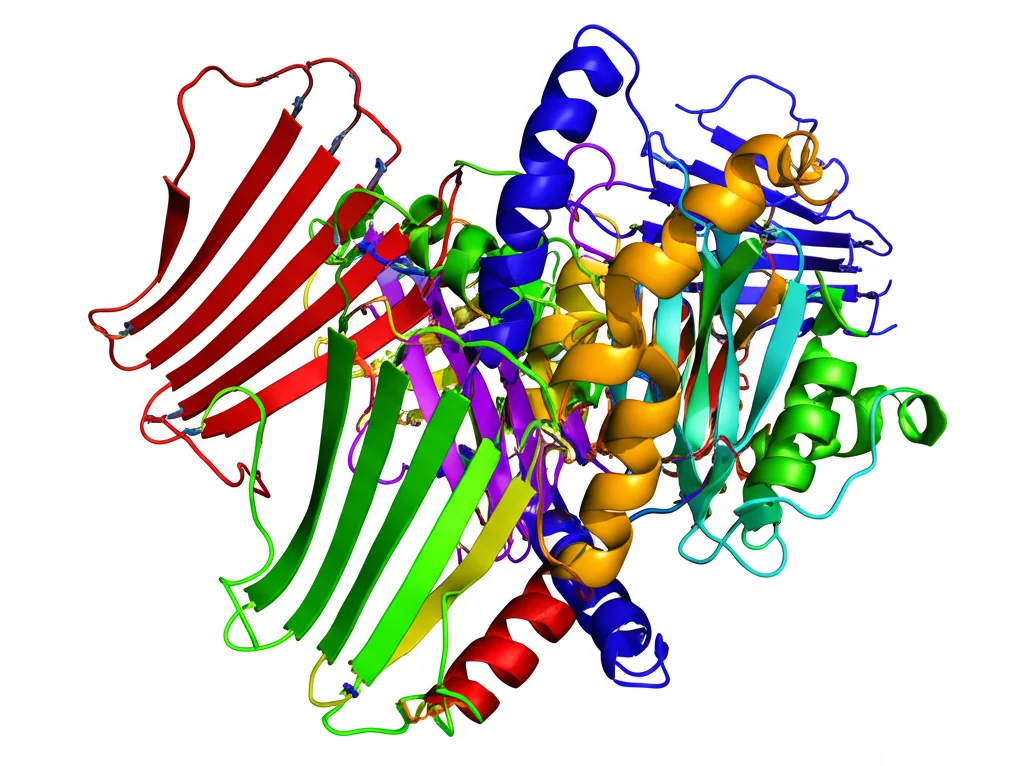
Tuttavia, quando ho simulato il comportamento di queste strutture 3D nel tempo (ne parlo tra poco), ho avuto una sorpresa: tendevano a “srotolarsi”, perdendo la loro forma tridimensionale compatta. Non era l’ideale per un vaccino efficace, che dovrebbe mimare il più possibile la struttura nativa dell’antigene.
Quindi, cambio di strategia! Invece di collegare singoli epitopi sparsi, ho deciso di utilizzare regioni più ampie delle proteine LukE e LukD, regioni naturalmente ricche di epitopi (una di 72 amminoacidi da LukE e una di 169 da LukD). Le ho unite usando linker specifici (KK, che aiutano anche la processazione dell’antigene) e ho collegato l’adiuvante hBD3 usando un linker flessibile (GGGGS)3, che si è rivelato il migliore per ottenere una conformazione stabile. Questa strategia mira a preservare una struttura più simile a quella naturale, potenzialmente migliorando l’efficacia immunologica.
Modellazione 3D e Simulazioni: Vedere per Credere
Avere la sequenza di amminoacidi del vaccino è solo l’inizio. Dovevo capire come questa catena si ripiega nello spazio tridimensionale, perché è la forma 3D che determina la funzione. Qui entra in gioco la modellazione computazionale. Ho usato ColabFold (basato sul potentissimo AlphaFold2) per predire la struttura 3D del mio costrutto vaccinale.
Ma una struttura predetta va validata! Ho usato strumenti come ProSA-web (che dà un punteggio Z, più è negativo meglio è) e il diagramma di Ramachandran (che verifica se gli angoli tra gli amminoacidi sono “permessi” dal punto di vista chimico-fisico). Il mio modello finale aveva un buon punteggio Z (-5.98) e la maggior parte dei residui (82.5%) nelle regioni favorite del diagramma di Ramachandran. Sembrava promettente!
Il test del nove, però, è stata la simulazione di dinamica molecolare (MD). Immaginate di poter osservare la proteina “vivere” e muoversi al computer per un tempo simulato (in questo caso, 100 nanosecondi), in un ambiente acquoso simile a quello biologico. Queste simulazioni, fatte con GROMACS, mi hanno permesso di verificare la stabilità della struttura 3D nel tempo. E sì, il costrutto basato sulle regioni ricche di epitopi si è dimostrato stabile! Ho anche verificato che gli epitopi B fossero esposti sulla superficie della proteina, pronti per essere riconosciuti dagli anticorpi.

L’Interazione Chiave: Il Vaccino Incontra il Sistema Immunitario (Virtualmente)
Un aspetto cruciale era capire se l’adiuvante (hBD3) nel mio vaccino potesse effettivamente interagire con i recettori del sistema immunitario che ne mediano l’effetto, in particolare i Toll-like receptors (TLR) 1 e 2. Questi TLR sono come delle sentinelle sulle cellule immunitarie, e legarli attiva la risposta.
Ho usato tecniche di docking molecolare (con HADDOCK) per predire come il mio vaccino si lega al complesso TLR1/2. I risultati indicavano una buona affinità. Ma non mi sono fermato lì. Ho eseguito un’altra simulazione MD, questa volta sull’intero complesso vaccino-TLR1/2, per 100 nanosecondi.
È stato affascinante vedere come le due molecole interagivano stabilmente nel tempo! Analizzando parametri come RMSD (deviazione quadratica media, misura della stabilità strutturale), RMSF (fluttuazione quadratica media, misura della flessibilità locale) e i legami idrogeno tra le molecole, ho potuto confermare un’interazione solida e stabile, soprattutto tra l’adiuvante e TLR2. Il legame induceva anche piccoli cambiamenti conformazionali nei TLR, suggerendo l’attivazione di segnali intracellulari. Il calcolo dell’energia libera di legame (MM/PBSA) ha confermato un’interazione molto forte, dominata da forze elettrostatiche. Era la prova virtuale che il mio vaccino aveva il potenziale per “accendere” il sistema immunitario nel modo giusto!
Pronti per il Laboratorio? Ottimizzazione e Clonazione In Silico
Un vaccino progettato al computer è fantastico, ma alla fine deve poter essere prodotto realmente in laboratorio. Il modo più comune per produrre proteine ricombinanti come questa è usare batteri come *Escherichia coli*. Per massimizzare le possibilità di successo, ho ottimizzato la sequenza di DNA che codifica per il mio vaccino, adattandola ai “codoni” preferiti da *E. coli* K12 (usando JCat). Ho ottenuto ottimi parametri (indice CAI di 0.94 e contenuto GC del 47.70%), che suggeriscono un’alta probabilità di espressione.
Infine, ho simulato l’ultimo passo: l’inserimento di questa sequenza di DNA ottimizzata in un vettore di espressione standard, il plasmide pET-28a(+), usando specifici siti di restrizione (NcoI e XhoI). Ho anche verificato che la proteina risultante avesse un’alta probabilità di essere solubile quando prodotta in *E. coli*. Tutto pronto, almeno virtualmente, per passare alla fase sperimentale!
Conclusioni e Prospettive Future: La Strada Verso un Vaccino Reale
Quindi, cosa abbiamo ottenuto? Abbiamo progettato *in silico* un candidato vaccino multi-epitopo contro la tossina LukED di *Staphylococcus aureus*. Utilizzando regioni ricche di epitopi invece di singoli epitopi, abbiamo ottenuto una struttura 3D stabile che mima meglio la conformazione nativa. Le simulazioni suggeriscono che è immunogenico, stabile, con buona copertura della popolazione mondiale (stimata al 65.15%), e capace di interagire efficacemente con i recettori TLR1/2 grazie all’adiuvante hBD3. È anche ottimizzato per la produzione in *E. coli*.
Questo lavoro pone le basi, ma la strada è ancora lunga. Ora servono esperimenti reali, *in vitro* e *in vivo*, per validare questi risultati computazionali. Bisognerà produrre la proteina, testare la sua capacità di indurre anticorpi neutralizzanti in modelli animali e valutarne sicurezza ed efficacia contro le infezioni da *S. aureus*.
È un percorso complesso, ma la possibilità di avere un giorno un vaccino efficace contro questo superbatterio, magari proprio grazie a strategie innovative come questa basata sulla bioinformatica, è una motivazione potentissima. Incrociamo le dita!
Fonte: Springer