Polθ Svelata: Viaggio al Cuore della Riparazione del DNA!
Ciao a tutti! Oggi voglio portarvi con me in un viaggio affascinante nel mondo microscopico delle nostre cellule, un luogo dove si combattono battaglie invisibili ma cruciali per la nostra sopravvivenza. Parleremo di DNA, di danni e, soprattutto, di un eroe molecolare un po’ misterioso: la proteina Polθ (Polimerasi Theta).
Immaginate il nostro DNA come un manuale di istruzioni incredibilmente lungo e complesso per costruire e far funzionare il nostro corpo. A volte, però, questo manuale subisce dei danni, come delle pagine strappate. Uno dei danni più gravi è la rottura del doppio filamento (DSB – Double-Strand Break). È come se il libro venisse tagliato netto in due punti! Se non riparate correttamente, queste rotture possono portare a caos genomico, instabilità e, nel peggiore dei casi, al cancro.
I Guardiani del Genoma: Meccanismi di Riparazione
Per fortuna, le nostre cellule hanno dei sistemi di riparazione super efficienti. I principali sono la Ricombinazione Omologa (HR) e l’Unione delle Estremità Non Omologhe (NHEJ). La HR è molto precisa, una sorta di “restauratore esperto”, mentre la NHEJ è più rapida ma a volte un po’ “approssimativa”.
Ma cosa succede quando la HR, il nostro restauratore di fiducia, non funziona bene? Questo accade, ad esempio, in alcune cellule tumorali che hanno mutazioni nei geni BRCA1 o BRCA2 (sì, quelli famosi legati ai tumori al seno e all’ovaio). In queste cellule, definite HR-deficient (HRD), entra in gioco un percorso alternativo, quasi un piano B: la Microhomology-Mediated End Joining (MMEJ), nota anche come Theta-Mediated End Joining (TMEJ). E chi è il protagonista indiscusso di questo piano B? Proprio lei, la nostra Polθ!
Polθ diventa fondamentale per la sopravvivenza di queste cellule tumorali HRD. È un po’ come dire che se manca il restauratore esperto (HR), ci si affida a un altro tipo di artigiano (Polθ con MMEJ) per tenere insieme i pezzi. Questa dipendenza rende Polθ un bersaglio incredibilmente promettente per nuove terapie antitumorali: se blocchiamo Polθ nelle cellule tumorali HRD, queste non riescono più a riparare i danni al DNA e muoiono. È un concetto chiamato letalità sintetica. Fantastico, vero?
Il Mistero di Polθ: Come Funziona Davvero?
Nonostante la sua importanza, i dettagli molecolari di come Polθ riesca a orchestrare la MMEJ sono rimasti a lungo poco chiari. Sappiamo che Polθ è una proteina grande, composta da tre parti principali: un dominio elicasi (Polθ-hel), che svolge il DNA; un dominio centrale flessibile; e un dominio polimerasi (Polθ-pol), che sintetizza nuovo DNA. Il nostro interesse si è concentrato sul dominio elicasico, Polθ-hel, perché sembra giocare un ruolo chiave nelle prime fasi della MMEJ.
Si pensava che Polθ-hel fosse coinvolta nel legare il DNA danneggiato, rimuovere altre proteine (come RPA) che si legano ai filamenti singoli, svolgere piccole porzioni di DNA e, soprattutto, cercare e appaiare brevi sequenze identiche (le microomologie) sulle estremità rotte del DNA per poi “incollarle”. Ma come faceva esattamente tutto questo?
Per scoprirlo, abbiamo deciso di “vedere” Polθ-hel in azione. Abbiamo utilizzato una tecnica potentissima chiamata crio-microscopia elettronica (cryo-EM), che permette di ottenere immagini tridimensionali ad altissima risoluzione delle molecole biologiche nel loro stato quasi naturale.

Abbiamo preparato dei “pezzi” di DNA che mimano le estremità rotte che Polθ incontra nella cellula: un doppio filamento con delle code sporgenti a singolo filamento (chiamate 3′-overhangs), alcune delle quali contenevano piccole sequenze di microomologia. Poi abbiamo mescolato questi DNA con la nostra Polθ-hel purificata e abbiamo congelato rapidamente i campioni per osservarli al cryo-EM.
La Sorpresa: Polθ Lavora in Coppia!
Le immagini ottenute sono state spettacolari! Ci hanno rivelato una serie di strutture di Polθ-hel in diversi momenti chiave: mentre lega il DNA, mentre cerca le microomologie e mentre le appaia (annealing).
Una delle prime scoperte importanti è stata che Polθ-hel funziona come un dimero, cioè due copie della proteina lavorano insieme, testa a testa. Questo era già stato ipotizzato, ma le nostre strutture lo hanno confermato in modo chiaro nel contesto della sua funzione con il DNA. Abbiamo visto che quando Polθ-hel lega il DNA, preferisce questa forma dimerica rispetto a una forma tetramerica (quattro copie unite) che a volte si osserva quando la proteina è libera. Sembra proprio che il legame al DNA spinga Polθ-hel a organizzarsi nella sua forma attiva: il dimero.
Il Viaggio del DNA nel Cuore dell’Elicasi
Le nostre strutture ci hanno mostrato in dettaglio come il DNA a singolo filamento (ssDNA) delle code sporgenti si infili attraverso un canale centrale presente in ciascuna delle due unità di Polθ-hel nel dimero. È un percorso preciso, guidato da interazioni specifiche tra il DNA e parti della proteina.
Abbiamo identificato i “punti di contatto” chiave:
- All’ingresso del canale, residui carichi positivamente (come le lisine K347 e K348) “afferrano” il DNA.
- All’interno, il DNA scivola lungo un’elica proteica (la “ratchet helix”) che probabilmente aiuta a spingerlo in una sola direzione (3′-5′), come un cricchetto. Residui idrofobici (V757 e M761) si intercalano tra le basi del DNA, agendo come un cuneo per impedire che torni indietro.
- All’uscita del canale, altre cariche positive (arginine R193 e R200) tengono fermo il DNA prima che questo emerga nello spazio tra le due unità del dimero.
È affascinante vedere come la struttura sia perfettamente progettata per gestire e orientare il filamento di DNA!
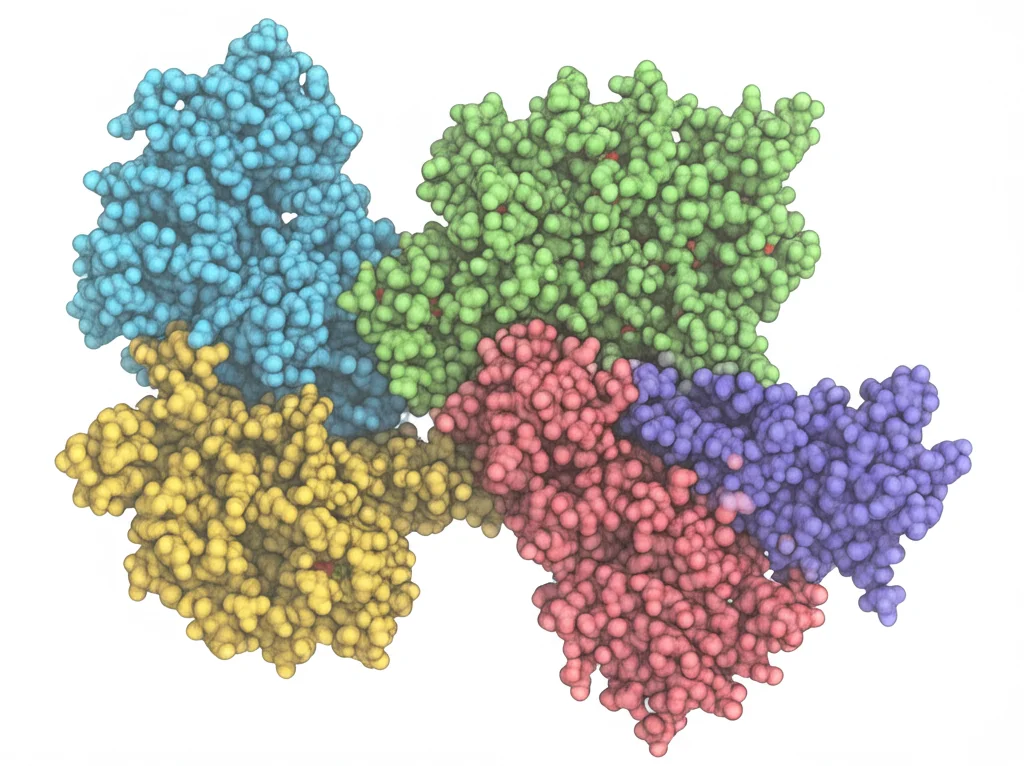
Un Dominio Acrobatico: Il Movimento di D5
Ma la sorpresa più grande è arrivata da un piccolo dominio di Polθ-hel chiamato D5. Nelle strutture senza DNA, o con un analogo dell’ATP (la molecola energetica che alimenta l’elicasi), il dominio D5 se ne sta attaccato al corpo principale dell’elicasi. Ma quando Polθ-hel lega il DNA destinato alla MMEJ, il dominio D5 fa qualcosa di incredibile: si stacca e si sposta completamente!
Abbiamo osservato che nello stato di “ricerca della microomologia”, D5 diventa addirittura invisibile nelle nostre mappe, suggerendo che sia molto flessibile e si sia allontanato. E nello stato in cui le microomologie sono appaiate (“MH annealed state”), D5 riappare in una posizione completamente nuova, incastonato nell’interfaccia tra le due unità del dimero. Non solo si sposta (di circa 42 Ångström, una distanza enorme a livello molecolare!), ma cambia anche parzialmente forma, ripiegandosi in modo diverso per adattarsi alla nuova posizione.
Perché questo movimento acrobatico? Sembra che lo spostamento iniziale di D5 sia necessario per fare spazio all’uscita del DNA dal canale e permettere la ricerca delle microomologie. Successivamente, quando D5 si riposiziona nell’interfaccia del dimero, aiuta a stabilizzare la struttura, tenendo ben salde le due estremità di DNA appaiate, pronte per le fasi successive della riparazione. È un interruttore molecolare essenziale!
Il Dimero si “Apre” per Lavorare
Oltre al movimento di D5, abbiamo notato che l’intero dimero di Polθ-hel subisce un cambiamento conformazionale quando lega il DNA. Le due unità ruotano leggermente l’una rispetto all’altra, passando da una forma più “chiusa” a una più “aperta”. Questo allarga lo spazio centrale (il “dimer cleft”), proprio dove avviene la magia della ricerca e dell’appaiamento delle microomologie. È come se il dimero si preparasse attivamente ad accogliere e processare le estremità del DNA rotto. Questa conformazione “aperta” è incompatibile con la formazione del tetramero, confermando ulteriormente che il dimero legato al DNA è l’unità funzionale.
La Prova del Nove: Mutazioni che Rompono il Dimero
Per essere sicuri che la forma dimerica fosse davvero essenziale per la funzione di Polθ-hel nella MMEJ, abbiamo fatto un esperimento cruciale. Abbiamo identificato gli amminoacidi chiave che tengono unite le due unità del dimero e li abbiamo modificati (mutati) per impedire la dimerizzazione. Abbiamo creato due mutanti (chiamati mutante 1 e mutante 2) che, come previsto, esistevano solo come monomeri (unità singole), sia da soli che legati al DNA.
Abbiamo poi testato la capacità di questi mutanti di eseguire il passo chiave della MMEJ: l’appaiamento delle microomologie. Utilizzando una tecnica basata sulla fluorescenza (FRET), abbiamo visto che mentre la Polθ-hel normale (WT, wild-type) era bravissima ad avvicinare e appaiare i DNA con microomologie, i nostri mutanti monomerici erano completamente incapaci di farlo, pur legando ancora il DNA. Questa è stata la prova definitiva: la struttura dimerica di Polθ-hel è assolutamente necessaria per la sinapsi e l’appaiamento delle estremità di DNA durante la MMEJ.
Un Modello per la MMEJ Mediata da Polθ
Mettendo insieme tutti questi pezzi del puzzle – le strutture cryo-EM, i movimenti conformazionali, i dati sulle mutazioni – possiamo ora proporre un modello più dettagliato di come Polθ avvii la MMEJ:
- Riconoscimento e Legame: Il dimero di Polθ-hel riconosce le estremità del DNA rotto con le code a singolo filamento (probabilmente dopo che la proteina RPA è stata rimossa, un compito che Polθ-hel può svolgere grazie alla sua attività ATPasica). Ogni unità del dimero lega una delle due estremità.
- Ricerca e Appaiamento: Polθ-hel, grazie alla sua capacità di traslocare lungo il DNA (alimentata da ATP) e alla flessibilità conformazionale (incluso il movimento di D5 e l’apertura del dimero), avvicina le due estremità e facilita la ricerca di brevi sequenze di microomologia. Una volta trovate, le appaia. Il riposizionamento di D5 stabilizza questo complesso.
- Estensione da parte della Polimerasi: A questo punto, il dominio polimerasi (Polθ-pol), collegato all’elicasi tramite il dominio centrale flessibile, entra in gioco. Si lega alle estremità appaiate e inizia a sintetizzare nuovo DNA per riempire le lacune.
- Completamento: Probabilmente in collaborazione con altre proteine (come ligasi e PCNA), la riparazione viene completata, saldando le interruzioni e ripristinando l’integrità del DNA.

Perché Tutto Questo è Importante?
Queste scoperte non sono solo affascinanti dal punto di vista della biologia molecolare fondamentale, ma hanno implicazioni concrete enormi. Capire nel dettaglio come funziona Polθ-hel ci permette di:
- Comprendere meglio un meccanismo cruciale di riparazione del DNA, specialmente rilevante in contesti tumorali.
- Identificare nuovi punti deboli nella struttura di Polθ-hel che possono essere bersagliati da farmaci.
- Sviluppare inibitori più specifici ed efficaci contro Polθ, aprendo la strada a terapie di precisione per i tumori HRD (mammella, ovaio, prostata, pancreas) che sfruttano la letalità sintetica.
Il nostro lavoro fornisce una mappa strutturale e meccanicistica dettagliata per Polθ-hel, illuminando i suoi primi passi critici nella MMEJ. È come aver finalmente capito come funziona un ingranaggio chiave in una macchina complessa. Ora possiamo pensare a come bloccare quell’ingranaggio in modo mirato nelle cellule che ne dipendono di più: le cellule tumorali. La strada è ancora lunga, ma aver svelato questi segreti strutturali di Polθ è un passo avanti fondamentale nella lotta contro il cancro.
Spero che questo viaggio nel cuore della riparazione del DNA vi abbia appassionato quanto ha appassionato noi scoprirlo!
Fonte: Springer