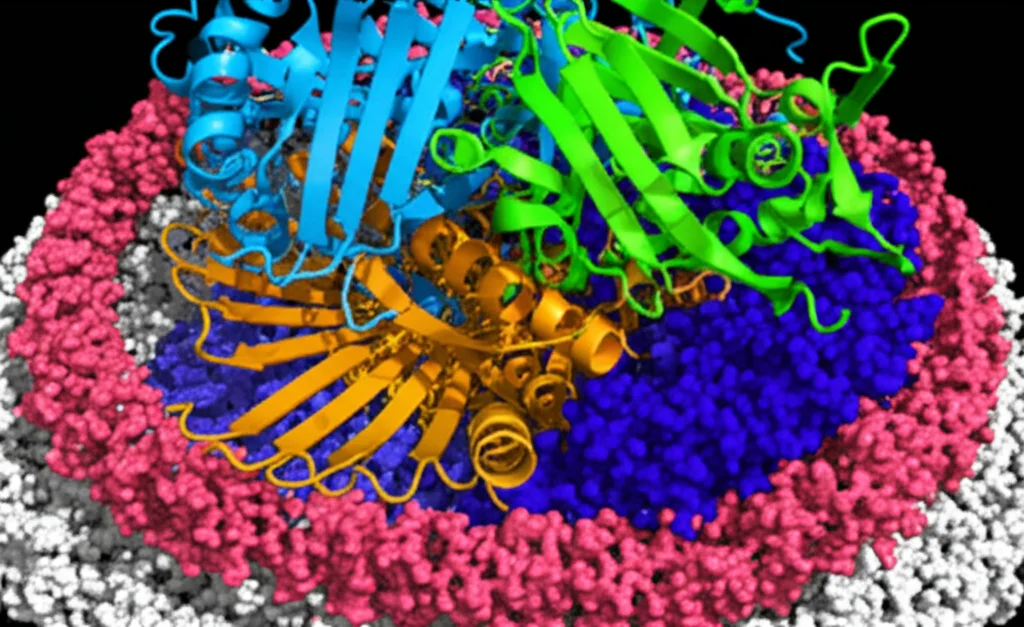
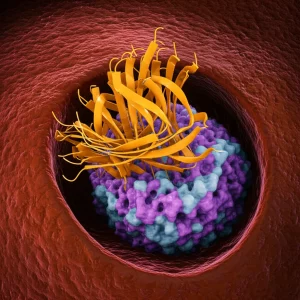
PimE: Viaggio nel Cuore dell’Enzima che Arma i Micobatteri
Ciao a tutti! Oggi voglio portarvi con me in un viaggio affascinante nel mondo microscopico dei batteri, in particolare di quelli che causano la tubercolosi (TB). Parleremo di come questi microrganismi costruiscono le loro difese e di come noi scienziati stiamo cercando di capire i loro segreti per poterli combattere meglio.
La Tubercolosi e la Corazza dei Micobatteri
La tubercolosi, causata dal batterio Mycobacterium tuberculosis (Mtb), è una malattia antica che purtroppo è ancora una delle principali cause di morte per malattie infettive a livello globale. Pensate che nel 2023 ha causato 1,25 milioni decessi! Ma cosa rende questi batteri così resistenti e difficili da debellare? Gran parte della risposta si trova nella loro complessa parete cellulare, una specie di corazza multistrato che li protegge dall’ambiente esterno e dal nostro sistema immunitario.
Questa parete è unica nel suo genere. Immaginate strati su strati: una membrana interna ricca di lipidi e glicolipidi speciali, poi uno strato di peptidoglicano legato ad arabinogalattano, e infine una sorta di “membrana esterna” chiamata micomembrana, ricca di acidi micolici a catena lunga e altri lipidi e glicolipidi complessi. Tra questi ultimi, spiccano i fosfatidil-mio-inositolo mannosidi, o più semplicemente PIM.
I PIM: Mattoncini Essenziali e Bersagli Potenziali
I PIM sono tra i glicolipidi più abbondanti nella membrana interna dei micobatteri. Sono fondamentali per mantenere l’integrità strutturale della parete, regolare cosa entra ed esce dalla cellula (permeabilità) e interagire con le cellule ospiti durante l’infezione. Non solo, alcuni PIM sono anche i precursori di molecole ancora più grandi e complesse come il lipomannano (LM) e il lipoarabinomannano (LAM), altri attori chiave nella virulenza dei micobatteri.
La sintesi dei PIM è un processo a tappe, un po’ come costruire con i LEGO. Diversi enzimi, chiamati mannosiltransferasi, aggiungono uno dopo l’altro residui di mannosio (uno zucchero) a una molecola base di fosfatidil-mio-inositolo (PI). I primi passaggi avvengono all’interno della cellula (nel citoplasma) grazie agli enzimi PimA e PimB. Poi intervengono altri enzimi, alcuni dei quali ancora sconosciuti, che aggiungono catene aciliche (grassi) e ulteriori mannosi, spostando la molecola in crescita verso l’esterno della membrana.
PimE: L’Operaio Specializzato Sotto la Lente
Il nostro protagonista di oggi è l’enzima PimE. Il suo compito specifico è aggiungere il quinto residuo di mannosio alla catena in crescita (partendo da un intermedio chiamato Ac1/2PIM4), formando così Ac1/2PIM5. Per fare questo, PimE utilizza un donatore di mannosio particolare, legato a un lungo lipide chiamato poliprenilfosfato (PPM). Questo passaggio è cruciale per la corretta costruzione della parete cellulare.
Perché ci interessa tanto PimE? Perché si è visto che i micobatteri senza PimE funzionante (mutanti ΔpimE) crescono male, hanno una forma anomala e, cosa importantissima, diventano più sensibili a diversi antibiotici e ad altri stress ambientali come il rame. Questo suggerisce che PimE potrebbe essere un ottimo bersaglio per sviluppare nuovi farmaci anti-TB. Il problema è che, finora, non sapevamo esattamente come funzionasse a livello molecolare: come riconosce i suoi “mattoncini” (substrati) e come catalizza la reazione?

Sbirciare Dentro PimE: La Criomicroscopia Elettronica
Per svelare i segreti di PimE, abbiamo deciso di “fotografarlo” ad altissima risoluzione. La tecnica scelta è la criomicroscopia elettronica (crio-EM) a particella singola. Questa tecnica permette di visualizzare la struttura tridimensionale delle molecole biologiche congelandole rapidamente e osservandole con un potente microscopio elettronico.
Abbiamo lavorato con la versione di PimE proveniente da un altro micobatterio, Mycobacterium abscessus (MaPimE), che si è dimostrato più facile da produrre e purificare in laboratorio. Poiché PimE è una proteina relativamente piccola per la crio-EM, abbiamo usato un “trucco”: l’abbiamo legata a un frammento di anticorpo (Fab) per renderla più grande e più facile da allineare durante l’analisi delle immagini.
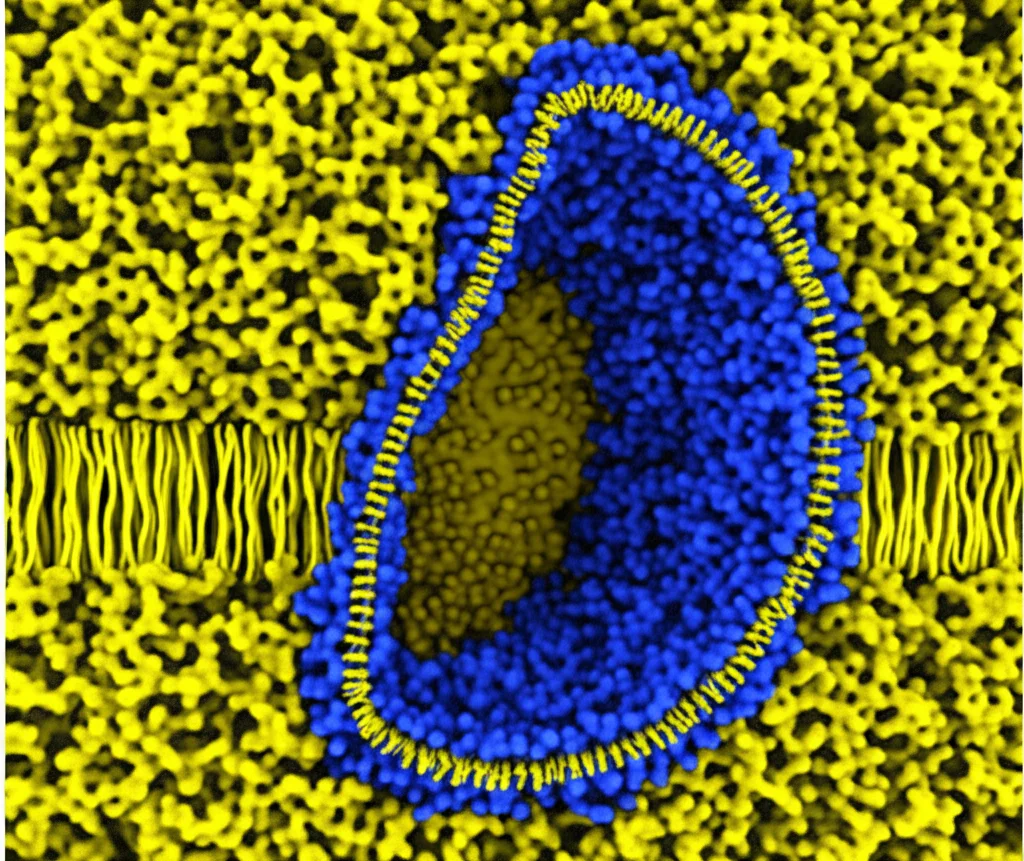
Dopo aver purificato PimE e averlo inserito in piccole “zattere” lipidiche chiamate nanodischi (per mimare il suo ambiente naturale nella membrana cellulare), abbiamo ottenuto due strutture ad alta risoluzione:
- La struttura di PimE “vuoto” (forma apo).
- La struttura di PimE con i prodotti della sua reazione legati all’interno (forma legata ai prodotti).
La Struttura Rivelata: Una Tasca su Misura
Cosa abbiamo visto? PimE è una proteina transmembrana, cioè attraversa la membrana cellulare, con ben dodici eliche che si avvolgono avanti e indietro. Entrambe le estremità della proteina (N- e C-terminale) si trovano sul lato citoplasmatico. La parte più interessante, però, è una cavità prominente situata all’interfaccia tra la porzione transmembrana e lo spazio esterno (periplasma).
Questa cavità ha una forma allungata, quasi “a anacardo”, ed è orientata quasi parallelamente alla membrana. Abbiamo subito ipotizzato che fosse il sito di legame per i substrati. Analizzandola più da vicino, abbiamo notato che:
- La parte centrale è prevalentemente idrofilica (ama l’acqua) ed è altamente conservata tra diverse specie di micobatteri. Qui si trova un residuo amminoacidico chiave, l’Aspartato 58 (D58), già sospettato di essere essenziale per la catalisi.
- Un’estremità della cavità (che abbiamo chiamato CE1) è più idrofobica (respinge l’acqua).
- L’altra estremità (CE2) è di nuovo più idrofilica.
- La distribuzione delle cariche elettriche non è uniforme: c’è una zona positiva vicino a CE1 e una negativa vicino a CE2 (dove si trova D58).
Queste caratteristiche ci hanno suggerito che la parte centrale fosse il vero e proprio centro di reazione, mentre CE1 potesse essere il punto d’ingresso per il donatore PPM (con la sua lunga coda idrofobica) e CE2 per l’accettore Ac1/2PIM4 (con la sua testa zuccherina idrofilica).
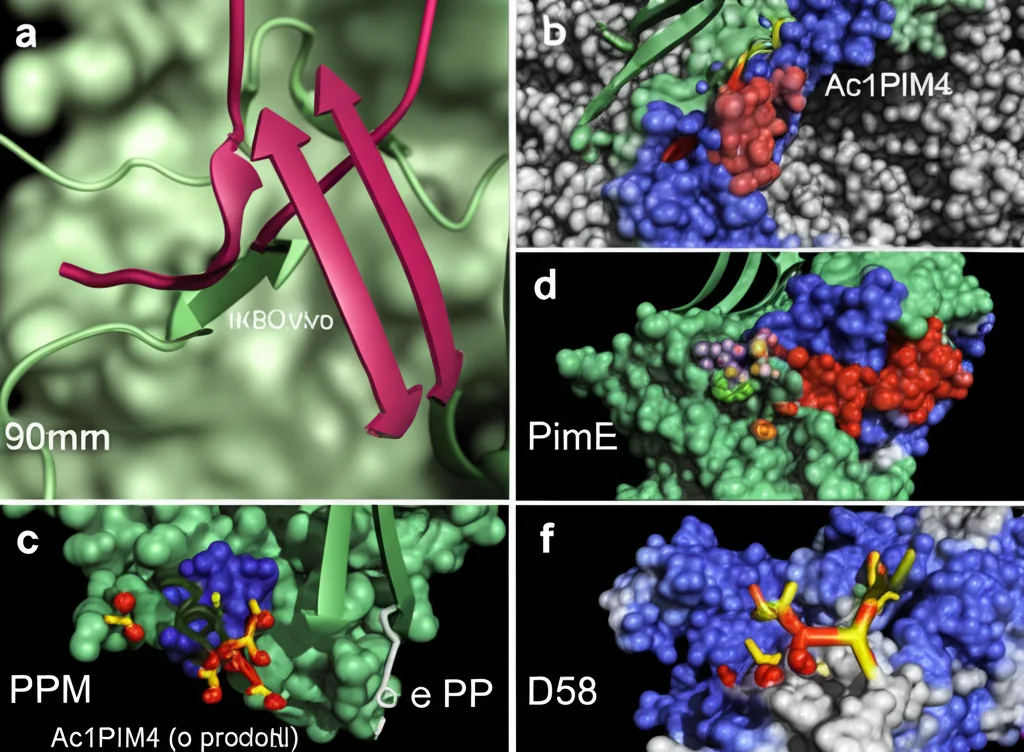
PimE Colto sul Fatto: La Struttura con i Prodotti
Per confermare le nostre ipotesi, abbiamo cercato di ottenere la struttura di PimE mentre era legato ai suoi substrati. Abbiamo aggiunto Ac1PIM4 (purificato da batteri) e PPM (prodotto enzimaticamente) durante la preparazione del campione per la crio-EM. Sorprendentemente, la struttura che abbiamo ottenuto non mostrava i substrati, ma i prodotti della reazione: Ac1PIM5 e il poliprenilfosfato (PP) rimasto dopo la cessione del mannosio! Questo significa che l’enzima era attivo anche nelle nostre condizioni sperimentali e aveva già completato la sua reazione.
Osservare dove si legano i prodotti ci ha dato informazioni preziose:
- Il PP si posiziona vicino a CE1, con la sua testa fosfato interagente con residui carichi positivamente (come K195 e H322) e la sua (corta) coda lipidica visibile che punta verso una scanalatura idrofobica tra le eliche transmembrana 6 e 9.
- Ac1PIM5 si trova dall’altro lato, vicino a CE2. La sua complessa testa zuccherina (con i 5 mannosi) occupa la parte centrale e idrofilica della cavità, interagendo con numerosi residui, tra cui il cruciale D58 (che lega il quinto mannosio appena aggiunto) e altri come Y62, H321, Y161, D160, W361 e R43. Le sue code aciliche, anche se non completamente visibili nella mappa di densità, sembrano estendersi verso la regione della membrana vicino all’elica 3.
Confrontando la struttura apo e quella legata ai prodotti, abbiamo notato piccoli ma significativi cambiamenti conformazionali, soprattutto in alcune anse (loop) della proteina che diventano più ordinate in presenza dei ligandi, suggerendo che si adattino per accogliere e posizionare correttamente i substrati/prodotti.
Come Funziona Esattamente? Meccanismo e Conferme Sperimentali
Grazie alle strutture e a simulazioni di dinamica molecolare (MD) – che ci permettono di vedere al computer come si muovono le molecole nel tempo – abbiamo potuto ricostruire il probabile meccanismo d’azione di PimE. Le simulazioni hanno confermato che PPM preferisce entrare da CE1 e Ac1PIM4 da CE2.
Abbiamo anche usato modelli computazionali (RoseTTAFold) per predire come i substrati completi (con le loro lunghe code lipidiche) si adattino nella cavità, confermando le interazioni viste sperimentalmente e suggerendone altre.
Ma la prova del nove viene sempre dagli esperimenti! Abbiamo creato diverse versioni mutate di PimE, cambiando specifici amminoacidi che la nostra struttura indicava come importanti. Poi abbiamo testato queste versioni mutanti:
- In vivo: Inserendo il gene mutato in ceppi di M. smegmatis (un “cugino” non patogeno di Mtb) privi del loro PimE e analizzando i PIM prodotti.
- In vitro: Usando membrane cellulari contenenti l’enzima mutato e fornendo i substrati (uno dei quali radioattivo per seguirne il destino).
I risultati sono stati chiarissimi e hanno confermato le nostre ipotesi strutturali:
- Mutare D58 (il presunto catalizzatore) o il suo vicino Y62 ha abolito completamente l’attività.
- Mutare residui che legano il fosfato del PPM/PP (come K195, W319, Y92, Q163) ha causato perdita totale o forte riduzione dell’attività.
- Mutare residui che interagiscono con la testa zuccherina di Ac1PIM4/5 (come Y161, W361, D160) ha anch’esso compromesso l’attività.
- Altri residui, pur non essendo strettamente essenziali, sembrano contribuire all’efficienza della reazione.
Un’altra scoperta interessante è che PimE sembra funzionare senza bisogno di ioni metallici, a differenza di molte altre glicosiltransferasi. Questo è supportato sia dai nostri saggi biochimici (l’attività non cambia con chelanti metallici o aggiunta di Mg²⁺) sia dall’assenza nella sua struttura di motivi tipici che legano metalli.
Quindi, il meccanismo proposto è questo: D58 agisce come una base catalitica, “strappa” un protone dal gruppo ossidrile (-OH) dell’accettore Ac1PIM4, rendendolo più reattivo per attaccare il mannosio del donatore PPM. Questo porta alla formazione del nuovo legame glicosidico (α1→2) e al rilascio di PP, stabilizzato da residui come K195 e H322.
Perché Tutto Questo è Importante? Verso Nuovi Farmaci
Capire in dettaglio come funziona PimE non è solo una curiosità scientifica. Come abbiamo detto, questo enzima è vitale per i micobatteri. La sua inibizione li rende più vulnerabili. Le nostre strutture ad alta risoluzione ci mostrano la “forma” esatta della tasca dove si legano i substrati e quali amminoacidi sono cruciali per la sua funzione.
Questa è un’informazione preziosissima per il disegno razionale di farmaci. Ora possiamo cercare o progettare piccole molecole che si incastrino perfettamente in quella cavità, bloccando l’attività di PimE. Poiché la via biosintetica dei PIM è altamente conservata tra le specie micobatteriche, un inibitore di PimE potrebbe essere efficace non solo contro M. tuberculosis, ma anche contro altri patogeni correlati come M. abscessus.
Il nostro studio fornisce quindi una solida base strutturale e meccanicistica per esplorare PimE come un nuovo bersaglio terapeutico. La lotta contro la tubercolosi è ancora lunga, ma ogni passo avanti nella comprensione di questi batteri così resistenti ci avvicina a nuove strategie per sconfiggerli. È un lavoro complesso, ma la speranza è quella di contribuire, un giorno, a rendere questa malattia un ricordo del passato.
Fonte: Springer