PAO-Sefarosa: La ‘Trappola’ Molecolare che Svela i Segreti Redox del Cuore
Ciao a tutti! Oggi voglio parlarvi di qualcosa che mi affascina da sempre nel mondo della ricerca: come fanno le nostre cellule, specialmente quelle del cuore, a “sentire” e rispondere ai segnali chimici? In particolare, come gestiscono lo stress ossidativo? Sapete, quelle molecole reattive dell’ossigeno (i famosi ROS) che sentiamo nominare spesso, a volte sono segnali utili, altre volte possono contribuire a malattie come quelle cardiovascolari. Capire come le proteine del nostro cuore percepiscono questi segnali è fondamentale.
I Sensori Nascosti: Le Cisteine nelle Proteine
Al centro di questo meccanismo ci sono spesso degli amminoacidi specifici nelle proteine: le cisteine. Immaginatele come delle piccole antenne sensibili. I loro gruppi tiolici (-SH) possono reagire con gli ossidanti. Una reazione comune è la formazione di un ponte disolfuro (-S-S-) tra due cisteine vicine. Questo cambiamento può modificare la forma della proteina, la sua attività o con chi interagisce. È un po’ come premere un interruttore molecolare!
Identificare quali proteine usano questo “interruttore redox” è una sfida. La proteomica redox ci ha dato grandi strumenti e liste sempre più lunghe di proteine candidate. Ma poi viene il bello: capire *come* l’ossidazione di una specifica proteina influenzi la sua funzione, specialmente in un organismo vivente, durante una malattia o una condizione fisiologica particolare. Tornare ogni volta a complesse analisi proteomiche per monitorare lo stato redox di *una* proteina specifica può essere dispendioso e non sempre praticabile.
Una “Trappola” Intelligente: La PAO-Sefarosa
Ed è qui che entra in gioco uno strumento che trovo particolarmente ingegnoso: la Fenilarsina Ossido (PAO) immobilizzata su Sefarosa. Sembra un nome complicato, ma l’idea è brillante nella sua semplicità. La PAO ha una caratteristica unica: si lega specificamente a coppie di gruppi tiolici (-SH) che sono vicini tra loro e *ridotti* (cioè non ossidati). Se queste due cisteine formano un ponte disolfuro a causa dello stress ossidativo, la PAO non riesce più a legarsi!
Quindi, cosa abbiamo fatto? Abbiamo usato questa PAO-Sefarosa come una sorta di “cromatografia di affinità redox”. Abbiamo preso campioni di cuore, li abbiamo messi a contatto con la PAO-Sefarosa e abbiamo visto quali proteine rimanevano “intrappolate”. Poi, abbiamo ripetuto l’esperimento dopo aver esposto i cuori a stress ossidativo (usando perossido di idrogeno o diamide, due noti ossidanti). Le proteine che venivano catturate meno dopo lo stress ossidativo erano proprio quelle che probabilmente avevano formato ponti disolfuro!
Esperimenti sul Cuore: Dalla Provetta all’Organismo
Inizialmente, abbiamo lavorato su cuori isolati di topo, perfusi in laboratorio (un modello chiamato Langendorff). Questo ci ha permesso di controllare l’ambiente e indurre uno stress ossidativo definito. Abbiamo identificato un bel po’ di proteine la cui cattura da parte della PAO-Sefarosa diminuiva significativamente dopo il trattamento con ossidanti.
Per avere un quadro ancora più completo, abbiamo usato anche un’altra strategia complementare: un modello di topo transgenico molto speciale. Questi topi esprimono nel cuore una versione modificata della Tioredossina-1 (Trx1), una proteina che normalmente riduce i ponti disolfuro. La nostra versione mutata (Cys35Ser) inizia il processo ma poi rimane “bloccata”, formando un complesso stabile con la proteina che stava cercando di ridurre. Marcando questa Trx1 mutata (con un tag FLAG-HA), siamo riusciti a “pescare” specificamente le proteine che si erano ossidate e avevano interagito con essa.
Confrontando i risultati dei due approcci (PAO-Sefarosa e Trx1 “trap-mutant”), abbiamo ottenuto una lista robusta di proteine cardiache candidate a essere dei sensori redox. Ma la domanda cruciale rimaneva: queste proteine si ossidano anche in vivo, in condizioni più fisiologiche o patologiche?
La Prova del Nove: Stress Ossidativo Reale
Per rispondere, siamo passati a modelli animali che mimano condizioni umane associate a stress ossidativo:
- Endotossiemia: Indotta iniettando lipopolisaccaride (LPS), un componente della parete batterica che scatena una forte infiammazione e stress ossidativo.
- Diabete di Tipo 1: Indotto con streptozotocina (STZ), che danneggia le cellule produttrici di insulina e causa iperglicemia e stress ossidativo cronico.
Abbiamo prelevato i cuori da topi sottoposti a questi trattamenti e abbiamo usato di nuovo la nostra fidata PAO-Sefarosa per vedere se alcune delle proteine candidate mostravano segni di ossidazione.
Risultati Sorprendenti (e Utili!)
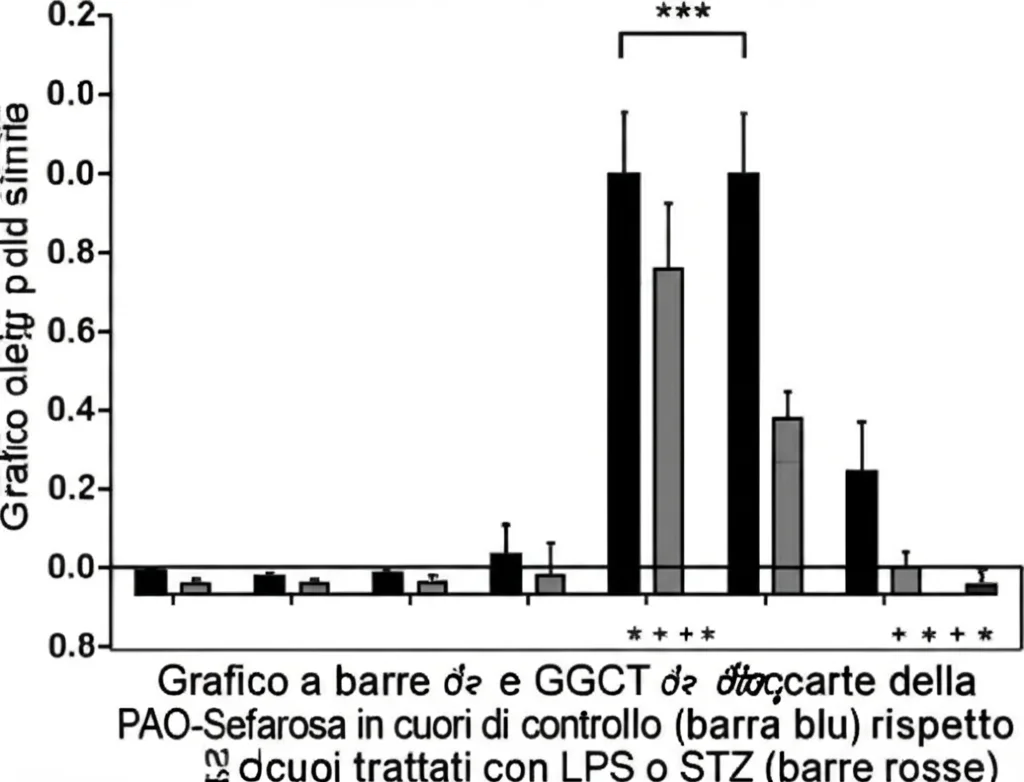
Abbiamo selezionato cinque proteine candidate dalle nostre liste iniziali (la scelta è stata guidata anche dalla disponibilità di buoni anticorpi, siamo pur sempre ricercatori pratici!). Ebbene, due di queste hanno mostrato una riduzione significativa della cattura da parte della PAO-Sefarosa nei cuori dei topi trattati con LPS o STZ. Queste due proteine sono:
- APIP (Apoptotic protease activating factor 1 interacting protein): Una proteina il cui coinvolgimento nello stress ossidativo era già stato suggerito da altri studi. Trovarla ossidata nei nostri modelli *in vivo* è una conferma importante!
- GGCT (γ-glutamylcyclotransferase): Un enzima coinvolto nel metabolismo del glutatione (un antiossidante chiave) e i cui livelli aumentano in diverse patologie cardiache. Il fatto che il suo stato redox cambi *in vivo* apre nuove domande sul suo ruolo funzionale.
Le altre tre proteine testate (TNNI3K, PHKG1, Nek7) non hanno mostrato cambiamenti significativi nel legame alla PAO-Sefarosa in questi modelli *in vivo*. Questo non significa che non siano sensibili all’ossidazione, ma probabilmente lo stress ossidativo generato *in vivo* da LPS e STZ è più specifico o meno intenso rispetto agli ossidanti chimici usati *ex vivo*, e colpisce un sottoinsieme diverso di proteine.
Perché la PAO-Sefarosa è un Alleato Prezioso?
Questo studio, secondo me, dimostra splendidamente l’utilità della PAO-Sefarosa. Non sostituisce le potenti tecniche di proteomica globale, che sono fantastiche per scoprire *nuovi* candidati. Ma una volta che hai una lista di sospetti, o vuoi testare una proteina specifica identificata magari da database come OxiMouse, la PAO-Sefarosa offre un metodo:
- Accessibile: Non richiede strumentazioni ultra-specialistiche come la spettrometria di massa per ogni singolo esperimento di follow-up.
- Relativamente Semplice: La procedura di cattura e analisi tramite immunoblotting è alla portata di molti laboratori.
- Efficace *in vivo*: Funziona bene anche su campioni complessi provenienti da modelli animali, dove lo stress ossidativo può essere più subdolo.
- Quantitativo (relativamente): Permette di confrontare lo stato di ossidazione tra diverse condizioni (es. controllo vs malattia).
Certo, ha i suoi limiti. Analizza principalmente proteine solubili e non ci dice *quali* specifiche cisteine sono coinvolte. Ma come strumento per monitorare lo stato redox di proteine d’interesse in contesti biologicamente rilevanti, è davvero prezioso.
Recentemente, è diventato disponibile anche uno strumento bioinformatico chiamato ReDisulphID, che predice potenziali ponti disolfuro redox-regolati. È stato confortante vedere che tutte e cinque le proteine che avevamo selezionato per i test *in vivo* sono state predette da ReDisulphID come contenenti cisteine vicinali potenzialmente ossidabili!
In conclusione, la “vecchia” chimica degli arsenicali trivalenti, intelligentemente immobilizzata su un supporto solido, ci offre ancora oggi un metodo elegante ed efficiente per sbirciare nel complesso mondo della segnalazione redox cardiaca. Ci permette di passare dalle grandi mappe proteomiche allo studio mirato di singole proteine in scenari che contano davvero per la salute e la malattia. E questo, per chi fa ricerca, è sempre entusiasmante!
Fonte: Springer







