Klebsiella Sotto Tiro: Vi Racconto il Nuovo Metodo PCR Super Veloce per Smascherarla!
Ciao a tutti! Oggi voglio parlarvi di una sfida che ci tiene sulle spine nel mondo della microbiologia clinica e della sanità pubblica: la lotta contro i batteri resistenti agli antibiotici. E in particolare, voglio puntare i riflettori su un “cattivo” piuttosto noto: Klebsiella pneumoniae.
L’incubo dell’Antibiotico-Resistenza e il Ruolo di Klebsiella
Sapete, l’antibiotico-resistenza è diventata un’emergenza globale. Le infezioni causate da batteri che se ne infischiano dei nostri farmaci più potenti, i cosiddetti batteri multi-resistenti (MDR), sono associate a malattie più gravi, degenze ospedaliere più lunghe e, purtroppo, a un aumento della mortalità. Costa vite e costa un sacco di soldi al sistema sanitario.
Mentre per alcuni batteri Gram-positivi sembra esserci una tregua, i Gram-negativi come le Enterobacteriaceae (la “famiglia” a cui appartiene Klebsiella) stanno diventando sempre più tosti. E Klebsiella pneumoniae è una delle protagoniste principali di questo dramma, specialmente nei reparti di terapia intensiva. Questo batterio è un osso duro perché ha sviluppato un arsenale di meccanismi di difesa:
- Produce biofilm protettivi.
- Utilizza pompe di efflusso per “sputare fuori” gli antibiotici.
- Ha una membrana esterna meno permeabile.
- Produce enzimi che inattivano gli antibiotici (come le famigerate carbapenemasi).
Insomma, un avversario formidabile.
L’Importanza di Essere Veloci: La Tipizzazione
Per controllare le infezioni da Klebsiella, le strategie classiche come l’igiene delle mani e l’uso appropriato degli antibiotici sono fondamentali. Ma c’è un altro pezzo del puzzle cruciale: capire chi abbiamo di fronte. Conoscere le caratteristiche epidemiologiche di questi batteri, cioè come si diffondono e quali “famiglie” (o ceppi) stanno circolando, è vitale per prevenire e controllare le epidemie negli ospedali e nella comunità.
Qui entra in gioco la tipizzazione molecolare. È come fare la carta d’identità genetica del batterio. Esistono diverse tecniche:
- L’elettroforesi su gel a campo pulsato (PFGE): per anni considerato il “gold standard” per la sua alta capacità di distinguere ceppi diversi.
- Tecniche basate sulla PCR come ERIC-PCR, RAPD, (GTG)5 PCR.
- Il sequenziamento dell’intero genoma (WGS): il metodo più completo, che ci dà una marea di informazioni.
Il WGS sarebbe fantastico, ma diciamocelo: è ancora costoso, lento e richiede sistemi complessi per interpretare i dati. Non proprio l’ideale per l’uso quotidiano in un laboratorio clinico che deve dare risposte rapide. Anche la PFGE, pur essendo potente, è laboriosa, richiede tempo, attrezzature specifiche e personale esperto.
La Svolta: Sfruttare i Siti XbaI con una PCR Multiplex
Ed è qui che arriva la parte affascinante! Ci siamo chiesti: non potremmo combinare l’affidabilità e il potere discriminante della PFGE con la semplicità e la velocità di una PCR? La risposta è sì, ed è nata l’idea del metodo XbaI-multiplex PCR.
L’idea di base è geniale nella sua semplicità. La PFGE spesso utilizza un enzima di restrizione chiamato XbaI, che taglia il DNA batterico in punti specifici (la sequenza 5′….T↓CTAGA….3′). Il numero e la dimensione dei frammenti ottenuti creano un profilo unico per ogni ceppo. Noi ci siamo concentrati proprio su questi siti di taglio *XbaI* e sulle piccole variazioni del DNA (i famosi SNP, Single-Nucleotide Polymorphism) che possono trovarsi proprio lì o nelle immediate vicinanze.
Invece di fare tutto il complesso processo di estrazione e digestione del DNA richiesto dalla PFGE, abbiamo sviluppato un metodo basato sulla PCR multiplex. Cosa significa? Significa che usiamo piccoli “inneschi” (primer) progettati specificamente per “agganciarsi” a 11 di queste regioni variabili intorno ai siti *XbaI* più discriminanti, cioè quelli che ci aiutano meglio a distinguere i diversi ceppi. Lanciamo una sola reazione PCR che amplifica contemporaneamente tutte queste 11 regioni.
Come Funziona e Cosa Abbiamo Scoperto
Il processo è molto più snello:
- Si estrae il DNA batterico (un processo standardizzato).
- Si esegue la reazione di XbaI-multiplex PCR (ci vogliono poche ore).
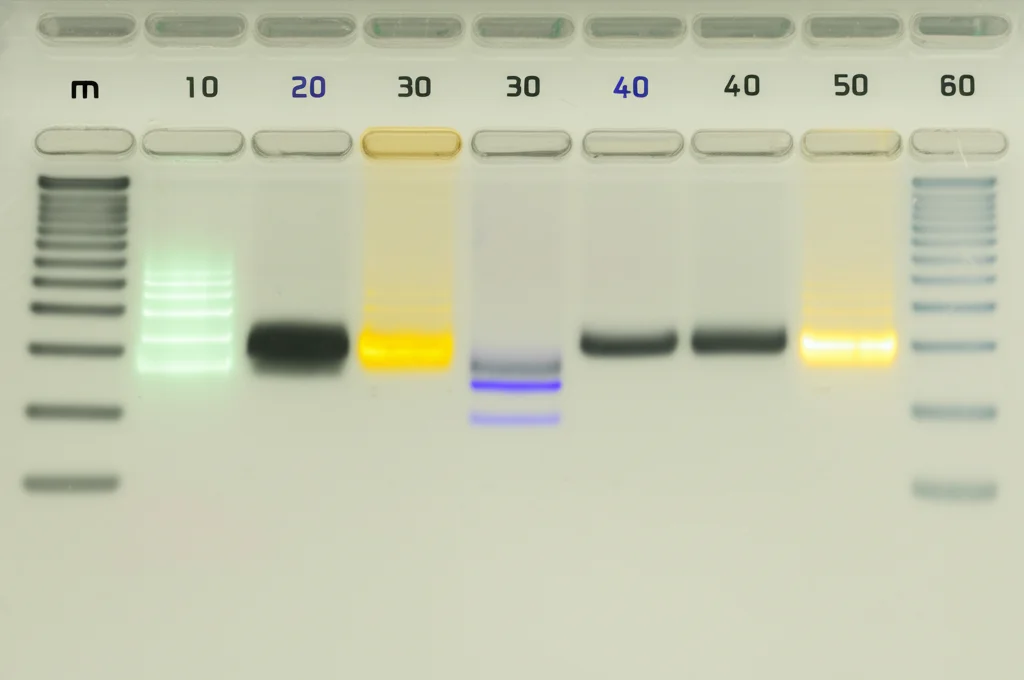
- Si analizzano i risultati su un semplice gel di agarosio: la presenza o l’assenza di una banda di una certa dimensione ci dice se quel particolare sito *XbaI* (o la regione vicina) è presente o assente in quel ceppo.
Il profilo risultante, cioè l’insieme delle bande presenti, è la “carta d’identità” del ceppo secondo questo metodo.
Abbiamo iniziato con un’analisi in silico, cioè al computer, usando i genomi di 30 ceppi noti di K. pneumoniae. Abbiamo mappato tutti i siti *XbaI* e identificato le regioni più variabili e utili per la discriminazione. Abbiamo poi progettato i primer per le 11 regioni più promettenti, sparse in tutto il genoma (il che è buono, perché riflette variazioni globali, non solo in un singolo gene).
Poi abbiamo testato virtualmente il metodo su 10 ceppi di riferimento per cui erano disponibili anche i dati PFGE-*XbaI*. I risultati sono stati incoraggianti! Il metodo XbaI-multiplex PCR è riuscito a tipizzare tutti i ceppi, mostrando profili distinti per quasi tutti (tranne due ceppi noti per essere molto simili). La cosa interessante è che i raggruppamenti ottenuti con il nostro metodo erano molto simili a quelli ottenuti con PFGE e con un altro metodo di riferimento, l’MLST (Multi-Locus Sequence Typing). Anzi, in un caso (due ceppi con lo stesso ST, ST11), il nostro metodo è riuscito a distinguerli, cosa che l’MLST non fa!
Una scoperta curiosa: uno dei set di primer (chiamato *XbaI*-3) ha prodotto frammenti di dimensioni diverse in ceppi diversi. Abbiamo scoperto che ciò è dovuto a un numero variabile di sequenze ripetute in quella regione del genoma. Questa variabilità inaspettata potrebbe addirittura aumentare il potere discriminante del nostro metodo!
La Prova sul Campo (Quasi): I Ceppi Clinici
Dopo i test virtuali, siamo passati a 29 isolati clinici reali di K. pneumoniae, provenienti da pazienti e già caratterizzati con MLST (appartenevano a 9 Sequence Types, ST, diversi). Anche qui, il metodo XbaI-multiplex PCR ha funzionato su tutti gli isolati, dividendoli in 4 profili distinti.
La maggior parte degli isolati (24 su 29) mostrava lo stesso profilo PCR. Tra questi, c’erano molti appartenenti al clone ad alto rischio ST14, ma anche isolati ST15 e ST2096 (che sono varianti molto vicine a ST14, fanno parte dello stesso “complesso clonale” CC14). Interessante notare che anche due isolati ST147 avevano lo stesso profilo. Questo ci dice che il metodo raggruppa bene ceppi strettamente correlati.
Abbiamo però notato una cosa: alcuni isolati ST101 mostravano lo stesso profilo degli ST14. Questo è stato un po’ inaspettato, perché MLST li distingue. Tuttavia, non è una contraddizione: il nostro metodo non guarda geni specifici come l’MLST, ma siti sparsi nel genoma. È pensato come uno strumento di screening rapido, non necessariamente per la caratterizzazione fine che fa l’MLST. Altri ceppi, come due isolati ST383 (un clone ipervirulento) e due isolati ST147, mostravano profili unici e coerenti tra loro.
I Vantaggi Innegabili (e Qualche Limite)
Allora, perché questo metodo XbaI-multiplex PCR è così promettente?
- Velocità: I risultati si ottengono in 4-6 ore, contro i giorni richiesti da PFGE o WGS.
- Costo-efficacia: È molto più economico delle tecniche di sequenziamento o della PFGE.
- Semplicità: Non richiede strumenti super complessi (bastano un termociclatore per PCR e un sistema per elettroforesi su gel, presenti in quasi tutti i laboratori) né competenze ultra-specialistiche.
- Buon potere discriminante: Ha dimostrato di poter distinguere bene molti ceppi, anche alcuni strettamente correlati.
- Potenziale di automazione: Potrebbe essere adattato per la PCR real-time (RT-PCR), riducendo ulteriormente i tempi (meno di 60 minuti!) e permettendo l’analisi automatica dei risultati.
Questo lo rende ideale per l’uso di routine nei laboratori di microbiologia clinica, specialmente nei paesi in via di sviluppo dove le risorse sono limitate.
Certo, come ogni nuovo metodo, ha bisogno di ulteriori conferme. Lo studio ha avuto dei limiti, principalmente legati alla disponibilità di fondi e di un numero maggiore e più vario di isolati clinici, magari provenienti da focolai epidemici ospedalieri. Non abbiamo potuto confrontarlo direttamente con PFGE o ERIC-PCR sugli isolati clinici.
In Conclusione
Nonostante i limiti, credo che il metodo XbaI-multiplex PCR rappresenti un passo avanti significativo. È uno strumento rapido, economico e accessibile per tenere d’occhio la diffusione di Klebsiella pneumoniae, aiutandoci a identificare rapidamente i ceppi circolanti e a mettere in atto misure di controllo più efficaci. In un’epoca in cui l’antibiotico-resistenza ci minaccia seriamente, avere strumenti diagnostici migliori e più veloci è fondamentale. E questa nuova tecnica sembra avere tutte le carte in regola per dare un contributo importante!
Fonte: Springer







