Micronuclei nel Mais: Frammenti di DNA Raccontano Storie Affascinanti di Danno Genetico
Ciao a tutti, appassionati di scienza e curiosi del mondo vegetale! Oggi voglio portarvi con me in un viaggio affascinante nel cuore delle cellule di mais, per scoprire dei piccoli protagonisti spesso trascurati: i micronuclei. Sembrano dettagli insignificanti, ma credetemi, sono come delle piccole capsule del tempo che ci raccontano storie incredibili sul DNA e sui danni che può subire.
Avete presente il mais, Zea mays? Quella pianta così importante per la nostra alimentazione e non solo. Bene, anche le sue cellule, come le nostre, possono subire stress e danni al loro prezioso materiale genetico. Quando questo accade, a volte si formano queste strutture extra, i micronuclei appunto. Ma da dove vengono? E cosa contengono esattamente? Sono tutti uguali? Queste sono le domande che ci siamo posti, e le risposte sono state più intriganti del previsto!
Ma cosa sono esattamente questi Micronuclei?
Immaginate il nucleo di una cellula come la biblioteca centrale che contiene tutte le istruzioni (i cromosomi, fatti di DNA) per far funzionare l’organismo. A volte, a causa di agenti “disturbatori” (chiamati mutageni, come sostanze chimiche o radiazioni), possono succedere due cose principali:
- Effetto Clastogenico: Un cromosoma si spezza! Un frammento senza una parte essenziale (il centromero, che serve per agganciarsi durante la divisione cellulare) rimane “orfano” e non viene incluso nel nuovo nucleo.
- Effetto Aneugenico: Un intero cromosoma non riesce ad agganciarsi correttamente durante la divisione cellulare e rimane indietro, escluso dal nucleo principale.
Questi frammenti o cromosomi “ritardatari” vengono poi racchiusi in una loro piccola membrana, formando un micronucleo separato dal nucleo principale. Sono la prova visibile che qualcosa è andato storto a livello del DNA.
Quanto DNA si perde? La Sorpresa delle Dimensioni
Una delle prime cose che abbiamo voluto capire è: quanto DNA finisce in questi micronuclei? È sempre la stessa quantità? Per scoprirlo, abbiamo trattato delle radici di mais con una sostanza chimica nota per causare danni al DNA, il metil metansolfonato (MMS), un vero “cattivo” per il genoma. Poi, usando una tecnica chiamata citometria d’immagine (che permette di misurare la quantità di DNA colorando i nuclei e analizzando l’intensità del colore al microscopio), abbiamo misurato il contenuto di DNA di tantissimi micronuclei.
E qui la sorpresa: i micronuclei non sono affatto tutti uguali! Abbiamo trovato una varietà incredibile di contenuti di DNA. Alcuni erano piccolissimi, contenendo solo una frazione minima del genoma totale del mais, mentre altri erano sorprendentemente grandi, arrivando a contenere l’equivalente di diversi frammenti cromosomici messi insieme. La maggior parte (circa il 69%) aveva un contenuto di DNA tra 0.5 e 1.5 picogrammi (pg), ma ne abbiamo trovati anche con valori fino a 3.0 pg! Considerando che il contenuto “base” (1C) del mais che abbiamo usato è circa 3.05 pg, significa che in alcuni casi si può perdere una porzione significativa del genoma, fino al 50% o più del valore di riferimento 1C, a causa della formazione di questi micronuclei. Questo ci dice quanto può essere esteso il danno causato da un mutageno come l’MMS.
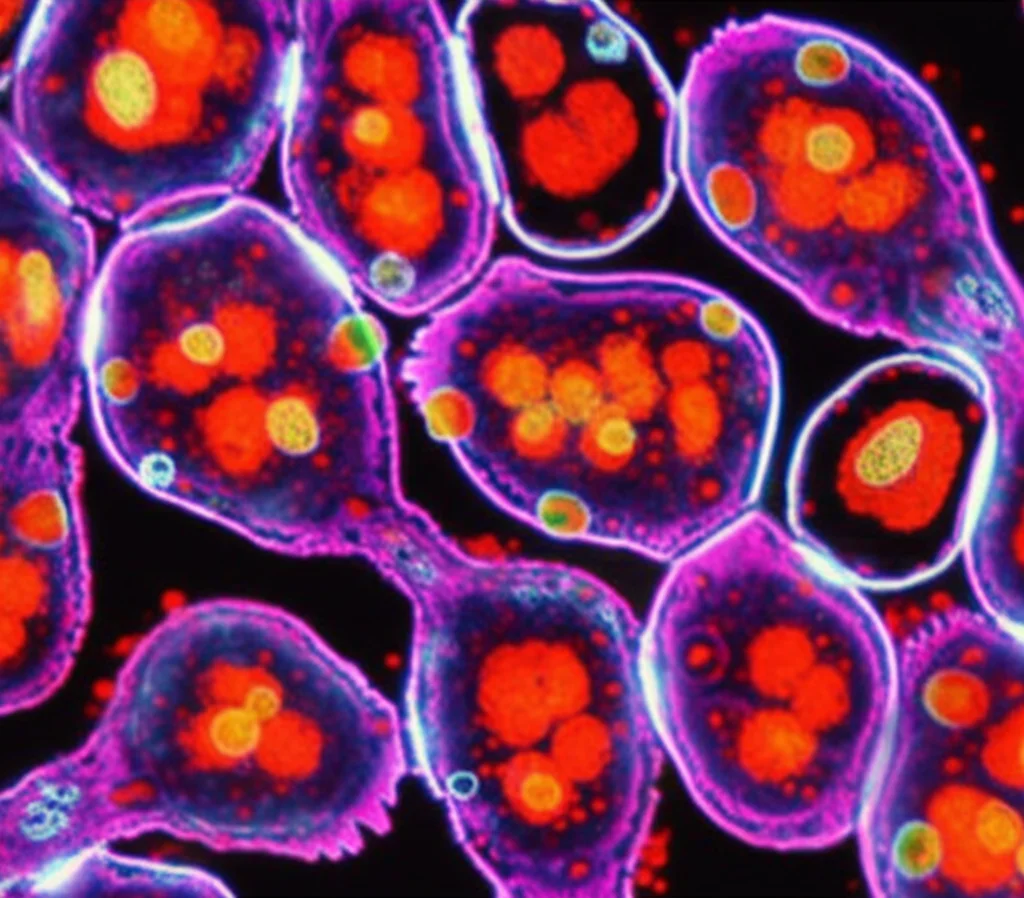
Cosa c’è dentro? Un’indagine sul Contenuto Specifico
Ok, i micronuclei hanno dimensioni diverse, ma cosa contengono nello specifico? Solo pezzi casuali di DNA o ci sono delle sequenze preferenziali? Per rispondere, abbiamo usato un’altra tecnica potentissima: la Fluorescenza In Situ Hybridization (FISH). In pratica, si usano delle “sonde” molecolari marcate con colori fluorescenti che si legano specificamente a determinate sequenze di DNA. È come usare degli evidenziatori molecolari per vedere dove si trovano certe parti del genoma.
Abbiamo cercato diverse sequenze ripetitive, che sono molto abbondanti nel genoma del mais e a volte considerate “punti caldi” per le rotture:
- DNA ribosomiale (rDNA) 5S e 18S: Sequenze importanti per produrre i ribosomi, le fabbriche di proteine della cellula. Le abbiamo trovate in circa il 22-25% dei micronuclei.
- Knob da 180-bp: Sequenze ripetute che formano regioni dense (eterocromatina) sui cromosomi. Trovate in quasi tutti i micronuclei (>97%), ma con intensità e distribuzione variabili!
- Retrotrasposone LTR Grande: Un tipo di “elemento genetico mobile”, DNA che può “saltare” nel genoma. Anche questo presente in oltre il 97% dei micronuclei, con segnali sparsi.
Abbiamo anche usato un colorante chiamato DAPI, che si lega preferenzialmente alle regioni ricche di Adenina (A) e Timina (T), spesso associate all’eterocromatina. Anche qui, abbiamo visto una grande variabilità nei segnali DAPI tra i micronuclei.
Cosa ci dice tutto questo? Che i micronuclei hanno una composizione molto varia! Contengono frammenti provenienti da diverse parti dei cromosomi, e sembra che alcune sequenze, come quelle dei knob e del retrotrasposone Grande, siano particolarmente inclini a finire nei micronuclei dopo il trattamento con MMS. Poiché l’MMS attacca preferenzialmente le basi Guanina (G) e Adenina (A), e queste sequenze sono ricche proprio di queste basi (l’rDNA e Grande sono ricchi di G, i knob di A), ha perfettamente senso che siano dei bersagli privilegiati!
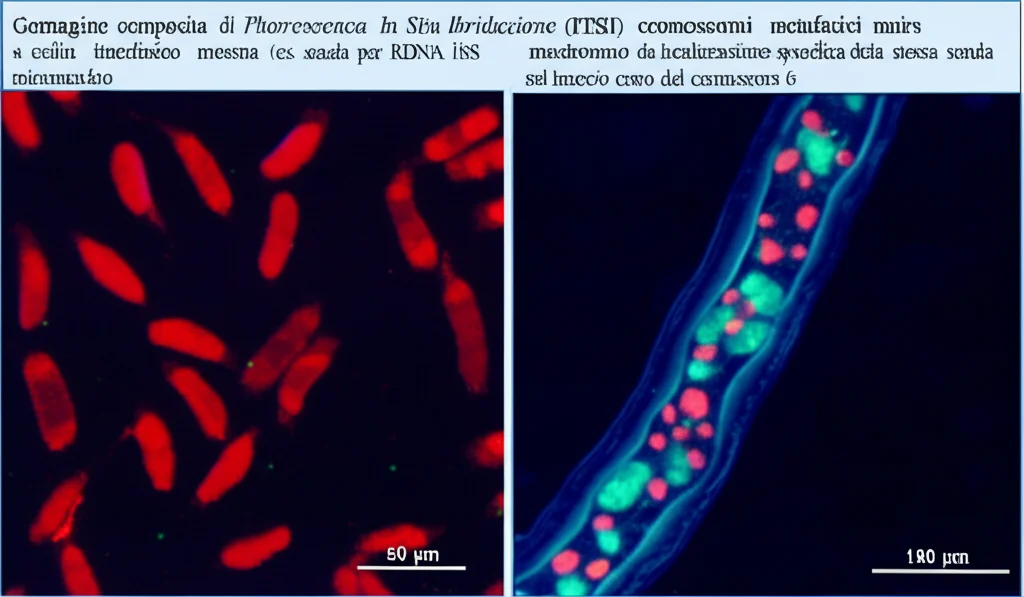
Da dove vengono esattamente? Il micro-detective genetico
L’ultimo pezzo del puzzle era capire se micronuclei diversi provenissero da regioni cromosomiche diverse. Qui abbiamo fatto qualcosa di veramente “micro”: abbiamo usato un micromanipolatore per isolare fisicamente singoli micronuclei dalle cellule! Un lavoro di precisione incredibile.
Una volta isolati, abbiamo amplificato il pochissimo DNA contenuto in due di questi micronuclei (chiamiamoli Micronucleo 1 e Micronucleo 2) e lo abbiamo usato per creare delle sonde specifiche. Abbiamo poi usato queste sonde “personalizzate” per fare di nuovo FISH sui cromosomi normali del mais. L’idea era: se la sonda del Micronucleo 1 si attacca a certe regioni dei cromosomi, significa che quel micronucleo proveniva da lì.
I risultati sono stati illuminanti! Entrambe le sonde si sono attaccate un po’ a tutti i cromosomi, confermando che sequenze comuni (come forse i retrotrasposoni) erano presenti in entrambi. MA, il pattern di ibridazione era diverso!
- La sonda del Micronucleo 1 si è attaccata lungo quasi tutta la lunghezza dei cromosomi, ma con meno intensità su alcune regioni specifiche (cromosoma 6, bracci corti dei cromosomi 3, 4 e 9).
- La sonda del Micronucleo 2 si è attaccata anch’essa diffusamente, ma ha evitato specificamente le regioni dei centromeri, dei knob e delle costrizioni secondarie (dove si trova l’rDNA 18S)!
Questa è la prova definitiva: i micronuclei indotti dall’MMS non solo hanno contenuto e composizione diversi, ma provengono anche da frammenti cromosomici differenti!
Perché tutto questo è affascinante (e importante)?
Questo studio ci mostra che i micronuclei sono molto più che semplici indicatori della presenza di danno al DNA. Sono delle finestre sulla diversità di questo danno. Ci dicono:
- Quanto DNA viene perso (variabilità nel contenuto).
- Quali tipi di sequenze sono coinvolte (composizione variabile, con possibili hotspot).
- Da quali regioni del genoma provengono questi frammenti (origine diversa).
Tutto questo è direttamente collegato al modo in cui il mutageno (in questo caso l’MMS) interagisce con il DNA. Abbiamo dimostrato l’estensione del danno genotossico e capito un po’ meglio la struttura e la diversità di queste piccole, ma informative, strutture cellulari.
La prossima volta che sentirete parlare di mutagenesi o di test di genotossicità, ricordatevi dei micronuclei. Non sono solo un “sintomo”, ma una fonte ricca di informazioni che aspetta solo di essere esplorata più a fondo per capire come proteggere il nostro prezioso DNA, sia esso umano o… di mais!
Fonte: Springer