Mielofibrosi Primaria: Quando la Metilazione del DNA Racconta una Storia Diversa dalle Mutazioni Driver
Ciao a tutti! Oggi voglio portarvi con me in un viaggio affascinante nel mondo microscopico delle nostre cellule, precisamente nel midollo osseo, dove nascono le cellule del sangue. Parleremo di mielofibrosi primaria (PMF), una malattia un po’ complessa, e di come un meccanismo chiamato metilazione del DNA stia svelando nuovi indizi sulla sua natura, a volte indipendenti dalle famose “mutazioni driver”. Pronti a scoprire qualcosa di nuovo?
Cos’è la Mielofibrosi Primaria e Cosa C’entrano le Mutazioni Driver?
Immaginate il midollo osseo come una fabbrica super efficiente che produce globuli rossi, globuli bianchi e piastrine. Nella mielofibrosi primaria, questa fabbrica inizia a funzionare male. Si sviluppa una sorta di cicatrice fibrosa nel midollo, che ostacola la normale produzione di cellule del sangue. È una malattia clonale, il che significa che deriva da una singola cellula “impazzita” che inizia a moltiplicarsi.
Da tempo sappiamo che alla base di questa “follia” ci sono spesso delle mutazioni genetiche specifiche, chiamate mutazioni driver perché sembrano proprio guidare lo sviluppo della malattia. Le più note nella PMF (e in altre malattie simili, le neoplasie mieloproliferative o MPN) sono a carico dei geni:
- JAK2 (soprattutto la variante V617F)
- CALR (calreticulina)
- MPL
Queste mutazioni sono quasi sempre mutuamente esclusive: se un paziente ha la mutazione JAK2, di solito non ha quella CALR o MPL, e viceversa. Questo ci ha fatto pensare: forse ogni mutazione dà un’impronta specifica alla malattia? Magari anche a livello epigenetico?
L’Epigenetica Entra in Scena: La Metilazione del DNA
Qui entra in gioco l’epigenetica. Pensate al DNA come a un enorme libro di istruzioni. Le mutazioni sono come errori di battitura nel testo. L’epigenetica, invece, è come aggiungere note adesive o sottolineature che dicono quali parti del libro leggere e quali ignorare, senza cambiare il testo stesso. La metilazione del DNA è uno di questi meccanismi epigenetici: piccoli “cappucci” chimici (gruppi metile) si attaccano a specifiche basi del DNA (le citosine, specialmente quando seguite da una guanina – i famosi siti CpG). Questo può accendere o spegnere i geni vicini.
Ci siamo chiesti: nella PMF, questi pattern di metilazione sono diversi rispetto a una persona sana? E soprattutto, le diverse mutazioni driver (JAK2, CALR, MPL) lasciano “firme” di metilazione distinte?
Cosa Abbiamo Scoperto: Somiglianze Inaspettate e Differenze Sottili
Analizzando i profili di metilazione del DNA in pazienti con PMF, abbiamo fatto scoperte interessanti. Prima di tutto, sì, i pazienti con PMF hanno profili di metilazione decisamente diversi dai controlli sani. Abbiamo identificato migliaia di siti CpG che erano “ipermetilati” (più cappucci del normale) o “ipometilati” (meno cappucci) nella PMF. Alcuni di questi cambiamenti riguardavano geni già noti per essere coinvolti in malattie del sangue, come RUNX1, BRD4, SRSF2.
Ma la vera sorpresa è arrivata confrontando i pazienti in base alla loro mutazione driver:
- I pazienti con mutazione JAK2 e quelli con mutazione CALR avevano profili di metilazione molto simili tra loro! Pochissime differenze significative.
- I pazienti con mutazione MPL, invece, mostravano cambiamenti di metilazione meno marcati e con pattern un po’ diversi rispetto agli altri due gruppi.
Questo suggerisce che, almeno per JAK2 e CALR, l’impatto diretto sulla “mappa” epigenetica della metilazione potrebbe non essere così diverso come pensavamo.

Le Mutazioni Driver Causano Direttamente Questi Cambiamenti? Il Test delle Cellule Staminali
Per capire se fosse proprio la mutazione JAK2V617F a causare direttamente i cambiamenti di metilazione, abbiamo usato un modello affascinante: le cellule staminali pluripotenti indotte (iPSC). In pratica, abbiamo preso cellule da pazienti con policitemia vera (un’altra MPN strettamente legata alla PMF e quasi sempre con JAK2 mutato), le abbiamo “riprogrammate” a uno stato staminale e poi abbiamo selezionato cloni che fossero senza mutazione (wild type), con una copia mutata (eterozigoti) o con entrambe le copie mutate (omozigoti).
Abbiamo poi fatto differenziare queste iPSC in precursori delle cellule del sangue (iHPC) e abbiamo guardato la loro metilazione. Risultato? Anche nelle cellule con la mutazione JAK2V617F, i pattern di metilazione visti nei pazienti PMF non venivano replicati in modo significativo. C’era solo una debole associazione, soprattutto per i siti ipometilati.
Questo è un indizio forte: forse i grandi cambiamenti epigenetici che vediamo nei pazienti non sono una conseguenza *immediata* e *diretta* della mutazione driver stessa, ma si sviluppano nel tempo, magari influenzati da altri fattori o dall’evoluzione della malattia nel complesso ambiente del midollo osseo.
Età Epigenetica Accelerata e il Ruolo del Carico Allelico
Un altro aspetto che abbiamo esaminato è l'”età epigenetica”. Esistono “orologi” basati sulla metilazione che possono stimare l’età biologica di una persona. Ebbene, nei pazienti con PMF, questa età epigenetica risulta significativamente accelerata rispetto all’età anagrafica, e questo accade indipendentemente dalla mutazione driver (JAK2, CALR o MPL).
Abbiamo anche verificato se la quantità di mutazione presente (il cosiddetto carico allelico, cioè la percentuale di cellule che portano la mutazione) fosse correlata all’entità dei cambiamenti di metilazione. Sorprendentemente, la correlazione era molto debole. Questo suggerisce che i cambiamenti epigenetici potrebbero non essere uniformi in tutte le cellule maligne, o che altri fattori siano più importanti nel determinarli.
Il Fattore “Confondente”: La Composizione Cellulare
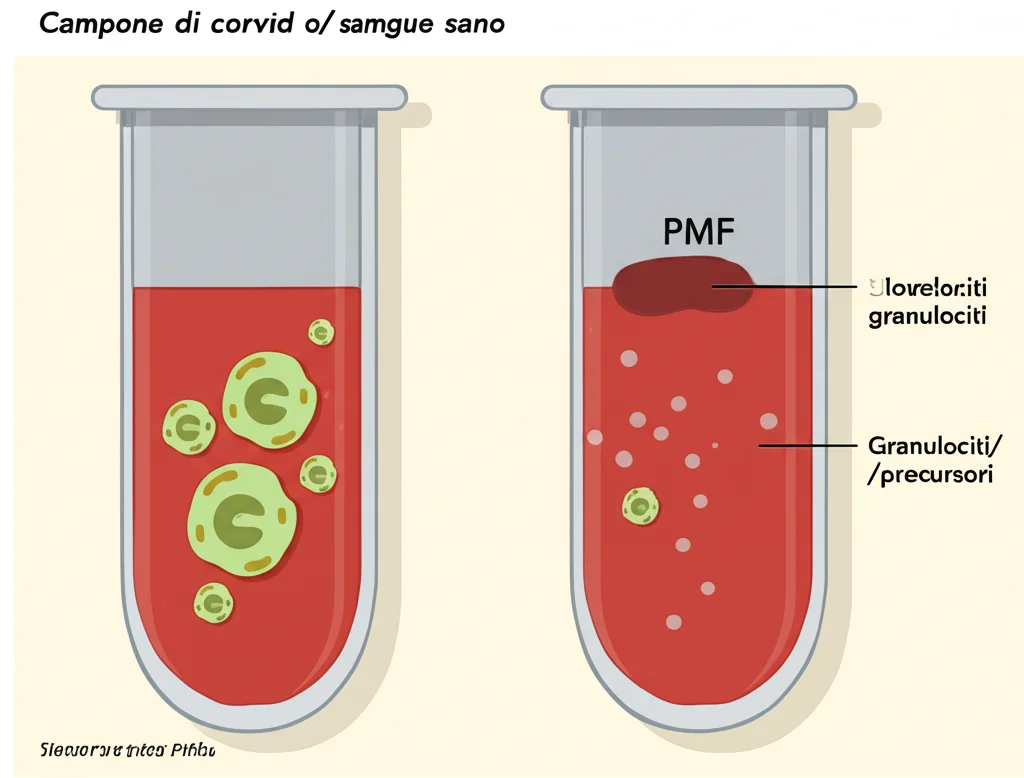
Qui arriviamo a un punto cruciale, spesso trascurato: la composizione cellulare del campione analizzato. Il sangue (o il midollo) è un mix di tanti tipi di cellule (granulociti, linfociti, monociti, ecc.), e ogni tipo cellulare ha un suo specifico pattern di metilazione di base. Nei pazienti con PMF, le proporzioni di questi tipi cellulari sono spesso alterate rispetto a una persona sana (ad esempio, può esserci un aumento di precursori mieloidi o granulociti).
Usando algoritmi bioinformatici per stimare la composizione cellulare dai dati di metilazione, abbiamo visto che i campioni PMF mostravano un “bias” verso i granulociti (o cellule progenitrici con profili simili). Quando abbiamo rifatto le analisi escludendo i siti CpG la cui metilazione varia molto tra i diversi tipi di cellule sane, il numero di differenze significative tra PMF e controlli si è drasticamente ridotto!
Questo significa che una buona parte dei cambiamenti di metilazione osservati nella PMF rispetto ai sani è probabilmente dovuta alla diversa miscela di cellule presenti nel campione, piuttosto che a cambiamenti intrinseci avvenuti *dentro* un tipo specifico di cellula maligna. Attenzione però: alcuni cambiamenti specifici della malattia rimangono anche dopo questa “correzione”.
PMF vs Altre Malattie Mieloidi: Un Profilo Unico?
E come si colloca la PMF rispetto ad altre malattie mieloidi come la leucemia mieloide acuta (LMA), le sindromi mielodisplastiche (SMD) o la leucemia mielomonocitica giovanile (LMMG)? Abbiamo confrontato i profili di metilazione.
Anche qui, la composizione cellulare gioca un ruolo enorme. Inizialmente, sembrava esserci una notevole sovrapposizione nei pattern di metilazione alterati tra tutte queste malattie. Tuttavia, una volta esclusi i siti CpG sensibili al tipo cellulare, le firme di metilazione per ciascuna malattia diventavano più distinte, con molte meno sovrapposizioni. Solo un sito CpG è risultato ipermetilato in *tutte* queste condizioni.
Questo ci dice due cose:
1. Molte alterazioni epigenetiche “generali” nelle malattie mieloidi potrebbero riflettere uno stato mieloide alterato comune o cambiamenti nella composizione cellulare.
2. Esistono però anche pattern più specifici per ogni malattia, una volta che si riesce a “filtrare” il rumore di fondo dato dalla varietà cellulare.
Verso una “Firma” Epigenetica per la PMF?
Sebbene distinguere la PMF dalle altre malattie mieloidi basandosi su singoli siti CpG sia difficile, abbiamo provato a vedere se una combinazione di pochi siti potesse funzionare meglio. Abbiamo identificato 5 siti CpG la cui metilazione (in particolare, una bassa metilazione) sembrava più caratteristica della PMF rispetto ai controlli e alle altre malattie analizzate (LMA, SMD, LMMG).
Abbiamo creato un semplice “PMF score” basato su questi 5 siti. Questo score riusciva a distinguere abbastanza bene i campioni PMF (e anche campioni di mielofibrosi secondaria e policitemia vera da set di dati di validazione) dai controlli e dalla maggior parte delle altre malattie mieloidi. È interessante notare che questo score non correlava con il carico allelico della mutazione driver, rafforzando l’idea che questi cambiamenti epigenetici abbiano una dinamica in parte indipendente. Inoltre, non c’erano grandi differenze nello score tra PMF con JAK2, CALR o MPL nei nostri campioni, anche se dati pubblici suggeriscono che le forme “triplo negative” (senza nessuna delle tre mutazioni classiche) potrebbero avere uno score leggermente più basso.
Attenzione: questo score è una prova di concetto, non uno strumento diagnostico pronto all’uso! Ma dimostra che, nonostante le complessità, i pattern di metilazione del DNA contengono informazioni che potrebbero, in futuro, aiutare a classificare meglio queste malattie.
Conclusioni: Un Quadro Complesso ma Affascinante
Insomma, cosa ci portiamo a casa da questo viaggio nella metilazione della PMF?
- La metilazione del DNA nella PMF è decisamente diversa da quella dei soggetti sani.
- Le mutazioni driver JAK2 e CALR sembrano associate a profili di metilazione molto simili, mentre MPL è un po’ a sé stante.
- Le mutazioni driver, da sole, potrebbero non essere la causa diretta dei principali cambiamenti epigenetici osservati nei pazienti; questi sembrano evolvere nel tempo.
- Gran parte delle differenze di metilazione osservate (sia rispetto ai sani che tra diverse malattie mieloidi) è influenzata dalla diversa composizione cellulare dei campioni. È fondamentale tenerne conto!
- Nonostante le sovrapposizioni e le complessità, esistono pattern di metilazione che sembrano più specifici per la PMF e che, combinati in “firme” o “score”, potrebbero avere un potenziale diagnostico o prognostico futuro.
La ricerca sull’epigenetica nelle malattie del sangue è in pieno svolgimento. Capire l’interazione tra genetica (le mutazioni) ed epigenetica (come la metilazione) è fondamentale per svelare i meccanismi profondi di malattie come la mielofibrosi primaria e, speriamo, per trovare nuove strategie terapeutiche. Il libro del DNA ha ancora tanti segreti da svelare, e le note epigenetiche sono una chiave di lettura sempre più importante!
Fonte: Springer