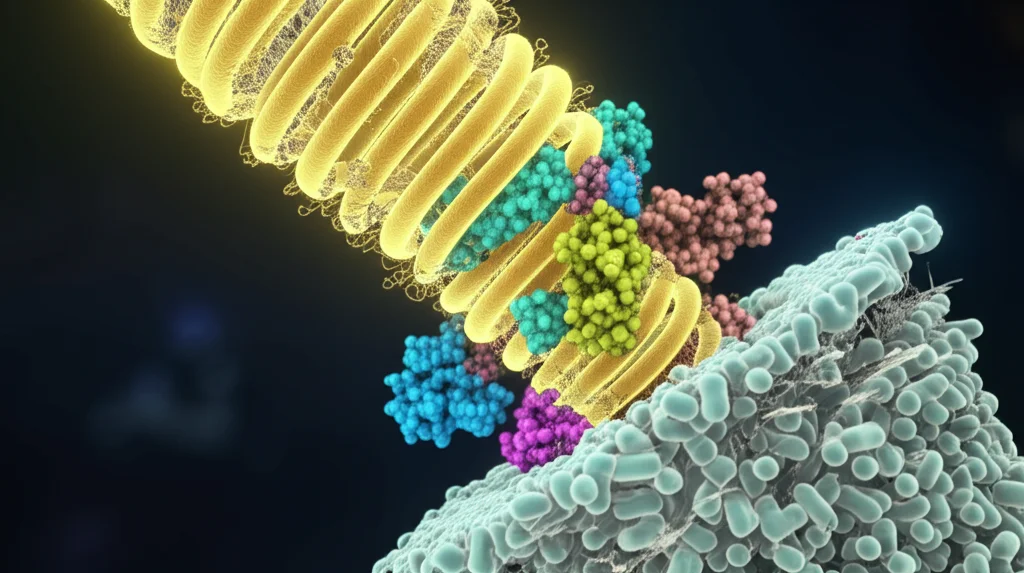
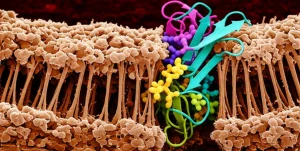
CpaF: La Danza Rotante che Costruisce le Nanomacchine Batteriche!
Ciao a tutti, appassionati di scienza e curiosi del mondo microscopico! Oggi voglio portarvi con me in un viaggio affascinante nel cuore delle cellule batteriche, alla scoperta di nanomacchine incredibilmente sofisticate. Parleremo di pili, quelle specie di “tentacoli” o “arpioni” che i batteri usano per un sacco di cose: muoversi, attaccarsi alle superfici (a volte anche alle nostre cellule, ahimè!), scambiarsi materiale genetico e persino percepire l’ambiente circostante.
In particolare, ci concentreremo su una famiglia specifica, i pili di tipo IV (T4P), e ancora più nello specifico sui pili Tad (Tight Adherence). Quello che rende i T4P così speciali è la loro dinamicità: possono allungarsi (assemblaggio) e ritirarsi (disassemblaggio) molto rapidamente. Ma come fanno? Serve energia, ovviamente, e qui entra in gioco il nostro protagonista: un motore molecolare chiamato ATPasi.
Il Motore Tuttofare: CpaF
Molti sistemi T4P usano due motori distinti, uno per costruire (come PilB) e uno per smontare (come PilT). Ma il sistema Tad, come quello che abbiamo studiato nel batterio d’acqua dolce Caulobacter crescentus, è più “minimalista”: usa un solo motore, chiamato CpaF, che è bifunzionale. Esatto, fa entrambe le cose: costruisce e smonta il pilo! Una bella sfida per un singolo enzima, non trovate?
La domanda che ci siamo posti è stata: come diavolo fa CpaF a gestire entrambi i compiti? Come fa un singolo motore a invertire la marcia, per così dire? Per capirlo, dovevamo “vedere” CpaF in azione. E qui entra in gioco la magia della crio-microscopia elettronica (cryo-EM). Questa tecnica pazzesca ci permette di congelare le molecole in un istante e di osservarle con una risoluzione incredibile, quasi atomica. È come scattare fotografie super dettagliate di queste macchine minuscole mentre lavorano.
Abbiamo purificato la proteina CpaF e l’abbiamo preparata in diverse condizioni: senza “carburante” (nucleotidi come ATP e ADP), con poco carburante e con il pieno. E indovinate un po’? Abbiamo beccato CpaF in tre diverse “pose”, tre conformazioni distinte che abbiamo chiamato: “chiusa”, “compatta” ed “espansa”.
La cosa interessante è che la quantità di ATP e ADP (le molecole che forniscono energia) influenzava quale forma CpaF preferiva assumere. Senza carburante, trovavamo sia la forma chiusa che quella compatta. Aggiungendo un po’ di ATP/ADP, la forma chiusa spariva e vedevamo quella compatta e quella nuova, espansa. Con tanto ATP, quasi tutte le molecole di CpaF erano nella forma espansa. Questo ci ha subito suggerito che queste forme diverse fossero legate al ciclo di utilizzo dell’energia (il ciclo catalitico).
Una Rotazione Controllata dall’Energia
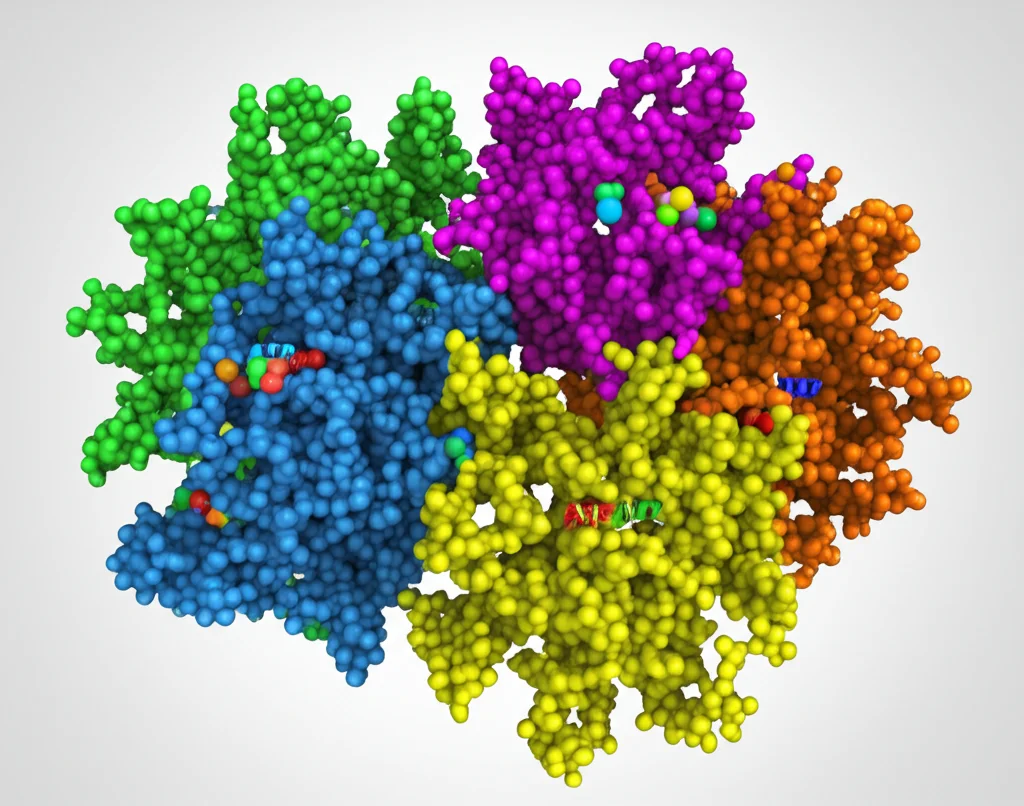
Analizzando nel dettaglio queste strutture (la forma compatta l’abbiamo vista a una risoluzione pazzesca, la più alta mai ottenuta per un’ATPasi di questo tipo!), abbiamo capito come funziona il motore. Immaginate CpaF come un anello formato da sei parti (un esamero). Ogni parte ha una tasca per legare l’ATP o l’ADP.
Abbiamo scoperto una cosa fondamentale:
- Quando l’ATP si lega a una subunità, l’intero anello si espande e, cosa cruciale, ruota leggermente in senso orario.
- Quando l’ATP viene usato (idrolizzato ad ADP), questo contribuisce al movimento.
- Quando l’ADP viene rilasciato, l’anello si contrae, tornando alla forma compatta, pronto per un nuovo ciclo.
In pratica, CpaF non spinge o tira in linea retta, ma usa un meccanismo rotante! L’energia dell’ATP viene convertita in un movimento di torsione. Pensate a un motore rotativo in miniatura. Ogni ciclo di legame, idrolisi e rilascio del nucleotide fa fare uno “scatto” rotatorio all’intero complesso. E questa rotazione, trasmessa ad altre parti della macchina di assemblaggio del pilo (le proteine della “piattaforma” nella membrana), è probabilmente ciò che spinge le subunità del pilo (chiamate PilA) a impilarsi una sull’altra, formando il filamento elicoidale. Dato che il pilo Tad ha una struttura elicoidale destrorsa, questo movimento rotatorio orario del motore CpaF si adatta perfettamente!
Il Mistero della “Coda” Disordinata (IDR)
CpaF ha anche una parte iniziale, una specie di “coda” (chiamata IDR, Regione Intrinsecamente Disordinata) che, come dice il nome, non ha una struttura ben definita. Ci siamo chiesti: a cosa serve? È lei a decidere se il motore deve costruire o smontare, come era stato ipotizzato da altri?
Per scoprirlo, abbiamo fatto un po’ di “chirurgia molecolare”: abbiamo creato versioni di CpaF senza questa coda, o con code più corte, direttamente nel batterio C. crescentus. Poi abbiamo usato la microscopia a fluorescenza per vedere quanti pili venivano costruiti e ritirati.
I risultati sono stati sorprendenti:
- Togliere la coda IDR riduceva sia l’estensione che la retrazione dei pili in modo simile. Non bloccava una funzione a favore dell’altra.
- In provetta, CpaF senza coda funzionava altrettanto bene (anzi, a volte anche un po’ meglio!) come motore ATPasi, e non era meno stabile.
Inoltre, guardando altri batteri, abbiamo visto che molte versioni di CpaF in giro per il mondo microbico non ce l’hanno proprio questa coda IDR! Eppure, alcuni di questi batteri devono per forza essere in grado sia di estendere che di ritrarre i loro pili Tad.
Quindi, la nostra conclusione è che l’IDR è importante, probabilmente aiuta a ottimizzare l’attività del pilo, magari interagendo con altre proteine o regolando la velocità, ma non è lei l’interruttore che decide la direzione del motore. Il meccanismo che permette a CpaF di essere bifunzionale deve essere più sottile e forse legato a come interagisce con il resto della macchina o con il pilo stesso.

Mettere Insieme i Pezzi: Il Modello con la Piattaforma
Ok, CpaF ruota. Ma come trasmette questo movimento per costruire il pilo? Qui entra in gioco la “piattaforma”, un complesso di proteine (CpaG e CpaH nel nostro caso) ancorato nella membrana interna del batterio, proprio sotto CpaF.
Ricostruire l’intera macchina è difficilissimo, ma oggi abbiamo strumenti potentissimi come AlphaFold3, un’intelligenza artificiale che predice la struttura delle proteine e come interagiscono. L’abbiamo usata per modellare come CpaF (nella sua forma espansa) si incastra con la piattaforma CpaG/CpaH.
Il modello suggerisce che specifiche parti di CpaF, in particolare delle anse e delle eliche che sporgono nel suo poro centrale, contattano direttamente le proteine della piattaforma. E indovinate quali parti? Proprio quelle che cambiano posizione durante il ciclo ATP! In particolare, un’ansa chiamata “extended pore loop” sembra cruciale: si alza e si abbassa a seconda che ci sia legato ATP o meno. Pensiamo che quando l’ATP si lega e l’ansa si alza, questa possa “agganciare” la piattaforma, trasmettendo la rotazione.
Il modello mostra anche come la piattaforma formi una specie di canale centrale attraverso cui le subunità PilA potrebbero passare dalla membrana al filamento in crescita. La rotazione indotta da CpaF potrebbe quindi non solo spingere le subunità ma anche aprire e chiudere dei “cancelli” nella piattaforma per farle entrare al momento giusto.
La cosa ancora più eccitante è che quando abbiamo usato AlphaFold3 per guardare sistemi simili in altri batteri (T4aP, T4bP, T2SS) e persino negli archea (archaellum), abbiamo visto che i motori ATPasi usano regioni molto simili per interagire con le loro piattaforme! Questo suggerisce che il meccanismo di base, questa interfaccia motore-piattaforma, potrebbe essere un tema comune, conservato nel corso dell’evoluzione per costruire tutte queste diverse nanomacchine filamentose.

Cosa Abbiamo Imparato e Cosa Resta da Scoprire
Quindi, ricapitolando: abbiamo “spiato” CpaF al lavoro, scoprendo tre sue conformazioni chiave. Abbiamo capito che usa un affascinante meccanismo rotatorio orario, alimentato dall’ATP, per guidare l’assemblaggio del pilo Tad. Abbiamo visto che la sua coda disordinata (IDR) ha un ruolo nel regolare l’efficienza, ma non nel decidere la direzione. E abbiamo un modello promettente di come CpaF interagisce con la piattaforma CpaG/CpaH, un’interazione che sembra conservata in molte altre nanomacchine batteriche.
Certo, restano domande aperte. La più grande è: come fa CpaF a invertire la rotazione per smontare il pilo? Il nostro lavoro suggerisce che non è l’IDR. Forse dipende da segnali esterni? Dalla tensione del pilo quando tocca una superficie? O da qualche altro componente della macchina che ancora non conosciamo?
Capire questi meccanismi non è solo affascinante dal punto di vista della biologia fondamentale, ma potrebbe anche aprire nuove strade per combattere i batteri patogeni. Se riusciamo a bloccare questi motori, potremmo impedire ai batteri di attaccarsi, muoversi o causare infezioni.
Il mondo delle nanomacchine biologiche è pieno di sorprese e meccanismi ingegnosi. Noi abbiamo aggiunto un altro tassello al puzzle, svelando la danza rotante di CpaF. E la ricerca continua!
Fonte: Springer