Mappatura Spaziale del Cervello: Un Viaggio Rivoluzionario tra Metaboliti, Lipidi e Glicani
Avete mai pensato a quanto sia incredibilmente complesso il nostro cervello? È una macchina biologica straordinaria, e per capirla a fondo, noi scienziati dobbiamo esplorare i suoi componenti più piccoli: le molecole. Oggi voglio raccontarvi di un’avventura affascinante che stiamo vivendo nei nostri laboratori, un nuovo modo per “vedere” come sono distribuite tre classi fondamentali di biomolecole – metaboliti, lipidi e glicani – direttamente nel tessuto cerebrale. È come avere una mappa super dettagliata che ci svela i segreti metabolici del cervello, sia in condizioni di salute che di malattia.
Un Trio Fondamentale: Metaboliti, Lipidi e Glicani
Immaginate il metabolismo come una vastissima rete stradale all’interno delle nostre cellule e dei nostri tessuti. I metaboliti sono come le piccole auto e i furgoni che trasportano energia e materiali da costruzione. I lipidi (i grassi, per intenderci) sono cruciali per costruire le membrane cellulari, immagazzinare energia e inviare segnali. Infine, i glicani (zuccheri complessi) sono come le indicazioni stradali e i semafori, essenziali per la comunicazione tra cellule e per molte altre funzioni vitali. Tradizionalmente, studiare queste tre famiglie di molecole richiedeva metodi separati, un po’ come guardare la mappa di una città un quartiere alla volta, perdendo la visione d’insieme. E se vi dicessi che ora possiamo guardare l’intera mappa contemporaneamente, e per di più in 3D (o quasi)?
La Sfida: Vedere Tutto Insieme e Nello Spazio Giusto
Il problema principale è che queste molecole sono molto diverse tra loro per dimensioni, solubilità e carica elettrica. È come cercare di fotografare contemporaneamente una formica, un elefante e un uccello in volo con la stessa macchina fotografica e le stesse impostazioni: una vera sfida! Inoltre, quando analizziamo un tessuto, spesso lo “frulliamo”, perdendo così tutte le informazioni su dove esattamente si trovavano quelle molecole. È un po’ come avere la lista di tutti gli abitanti di una città, ma non sapere in quale casa vivono. Per capire veramente come funziona il cervello, o cosa va storto in malattie come l’Alzheimer, abbiamo bisogno di sapere non solo quali molecole ci sono, ma anche dove sono localizzate.
Qui entra in gioco una tecnica potentissima chiamata imaging mediante spettrometria di massa MALDI (Matrix-Assisted Laser Desorption/Ionization). Pensatela come uno scanner super avanzato che, bombardando sottilissime fette di tessuto con un laser, ci permette di identificare migliaia di molecole diverse e, soprattutto, di sapere esattamente da quale punto del tessuto provengono. È come creare una mappa molecolare ad alta risoluzione.
La Nostra Soluzione: Un Flusso di Lavoro Integrato e Sami
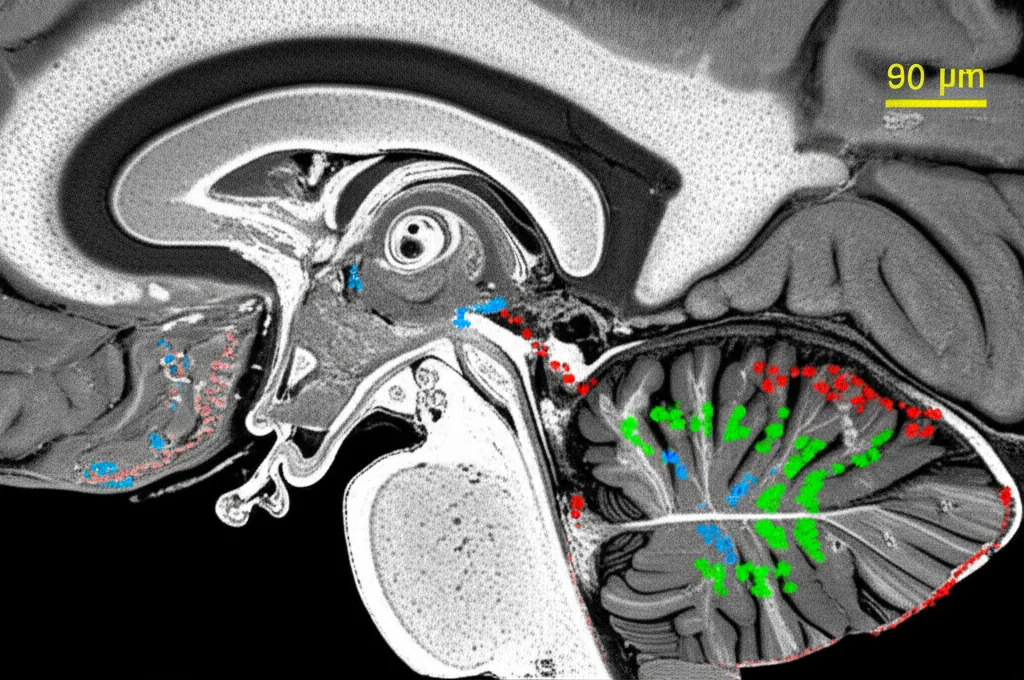
Nel nostro studio, abbiamo messo a punto un flusso di lavoro che ci permette, per la prima volta, di analizzare metaboliti, lipidi e glicani dalla stessa singola sezione di tessuto cerebrale. Questo è un passo da gigante! In pratica, prendiamo una fettina sottilissima di cervello di topo (spessa solo 10 micrometri, circa un decimo dello spessore di un capello), la trattiamo con una speciale “matrice” chimica e la analizziamo con il MALDI per i metaboliti e poi per i lipidi. Successivamente, sulla stessa fettina, rimuoviamo la prima matrice, applichiamo degli enzimi specifici per liberare i glicani e il glicogeno (una forma di zucchero immagazzinato), aggiungiamo un’altra matrice e rianalizziamo tutto. In questo modo, preserviamo l’integrità spaziale del tessuto e otteniamo un quadro completo.
Ma raccogliere tutti questi dati è solo metà del lavoro. Immaginate di avere migliaia di mappe sovrapposte: come si fa a dare un senso a tutto? Per questo, abbiamo sviluppato una piattaforma computazionale che abbiamo chiamato Sami (Spatial Augmented Multiomics Interface). Sami è il nostro “cervellone” digitale: integra i dati provenienti dalle tre analisi (metabolomica, lipidomica e glicomica), li armonizza e utilizza algoritmi sofisticati per identificare pattern spaziali, raggruppare regioni con profili molecolari simili (clustering) e persino suggerire quali vie metaboliche sono più attive in quelle specifiche aree. È come avere un navigatore GPS super intelligente per esplorare il paesaggio metabolico del cervello.
Abbiamo testato Sami su cervelli di topo sani e su un modello murino della malattia di Alzheimer (i topi Ps19, che sviluppano una patologia simile alla demenza frontotemporale umana). I risultati sono stati strabilianti! Siamo riusciti a vedere come diverse regioni del cervello sano abbiano “firme” metaboliche uniche, come se ogni quartiere della città avesse le sue attività commerciali e i suoi ritmi specifici. Ad esempio, l’ippocampo, cruciale per la memoria, ha mostrato un’intensa attività del ciclo dell’acido citrico e del metabolismo del glutammato, un neurotrasmettitore eccitatorio fondamentale. Il corpo calloso, invece, che connette i due emisferi cerebrali, ha mostrato un’attività prevalente legata alla glicolisi e all’effetto Warburg, un modo particolare di utilizzare il glucosio.
Svelare i Cambiamenti nell’Alzheimer
Quando abbiamo confrontato i cervelli sani con quelli dei topi Ps19, Sami ci ha aiutato a individuare le aree dove il metabolismo era andato in tilt. Nella corteccia dei topi malati, ad esempio, abbiamo osservato alterazioni significative nel ciclo dell’acido citrico, nell’effetto Warburg e nel metabolismo della carnitina. È interessante notare che, sebbene alcune vie metaboliche fossero alterate sia nelle diverse regioni del cervello sano sia nel confronto tra cervello sano e malato, i cambiamenti specifici a livello dei singoli metaboliti erano spesso diversi, a volte persino opposti! Questo ci dice quanto sia importante non solo identificare le vie coinvolte, ma anche capire come sono alterate nel dettaglio.
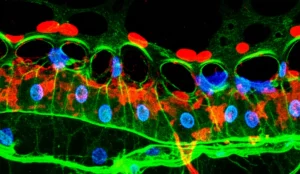
Un’altra scoperta importante, grazie all’analisi glicomica, è stata l’aumento significativo di glicogeno nella corteccia dei topi Ps19. Per confermare queste scoperte “multiomiche”, abbiamo usato una tecnica classica, l’immunofluorescenza, per visualizzare direttamente alcune proteine chiave del ciclo dell’acido citrico e il glicogeno. Ebbene, i risultati hanno confermato ciò che Sami ci aveva mostrato: una riduzione di enzimi come la citrato sintasi e l’aconitasi, e un aumento del glicogeno nel cervello dei topi malati. È stata una grande soddisfazione vedere come queste tecniche diverse si supportassero a vicenda!
Perché Tutto Questo è Importante?
Questo approccio integrato, che combina l’imaging MALDI sulla stessa sezione di tessuto con la potenza analitica di Sami, apre scenari incredibili per la ricerca. Possiamo finalmente ottenere una visione molto più olistica e spazialmente definita del metabolismo nei tessuti complessi. Questo non solo ci aiuta a capire meglio come funziona il cervello in condizioni normali, ma ci fornisce anche nuovi indizi sulle cause e sulla progressione di malattie neurologiche come l’Alzheimer, la sclerosi multipla o i tumori cerebrali. Identificare specifiche alterazioni metaboliche in determinate regioni del cervello potrebbe portare, in futuro, a sviluppare nuove strategie diagnostiche e terapeutiche più mirate.
Certo, c’è ancora strada da fare. Ad esempio, al momento la nostra risoluzione non ci permette di scendere al livello della singola cellula, ma le tecnologie stanno avanzando rapidamente. Stiamo già pensando a come integrare queste analisi con altre tecniche di imaging cellulare e a come espandere il numero di molecole che possiamo “vedere”. L’obiettivo è creare mappe metaboliche sempre più dettagliate e complete, non solo per il cervello ma per tutti i tessuti e organi del corpo.
In conclusione, quello che abbiamo sviluppato è più di una semplice tecnica: è una nuova finestra sul mondo invisibile del metabolismo. È un po’ come passare da una vecchia mappa sbiadita a Google Earth, con la possibilità di zoomare e vedere i dettagli più nascosti. Siamo convinti che questo approccio, che abbiamo chiamato “triple-omics” spaziale, guiderà la prossima ondata di scoperte nel campo del metabolismo, aiutandoci a svelare i meccanismi alla base della salute e della malattia. E per me, far parte di questa avventura è incredibilmente stimolante!
Fonte: Springer