LMA e Disulfidptosi: Ho Trovato 7 LncRNA Che Potrebbero Prevedere il Futuro dei Pazienti!
Ciao a tutti! Oggi voglio parlarvi di qualcosa di veramente affascinante che sta emergendo nel campo della ricerca sulla Leucemia Mieloide Acuta (LMA). Come sapete, la LMA è un osso duro, una neoplasia ematologica aggressiva con una prognosi spesso sfavorevole, specialmente negli adulti dove il tasso di sopravvivenza globale si aggira tristemente intorno al 30%. È una malattia che nasce dalle cellule staminali/progenitrici mieloidi del nostro sangue, caratterizzata da una proliferazione incontrollata e da un blocco della morte cellulare programmata (apoptosi) delle cellule immature, soffocando così la normale produzione di cellule del sangue.
Nonostante i progressi, come la chemioterapia standard “3+7”, farmaci mirati come venetoclax e azacitidina, e il trapianto di cellule staminali ematopoietiche, troppi pazienti vanno incontro a recidive refrattarie. La LMA è incredibilmente eterogenea, il che significa che diversi sottotipi rispondono in modo diverso alle cure. Ecco perché è fondamentale trovare nuovi modi per valutare la prognosi a livello molecolare, per offrire trattamenti davvero personalizzati e migliorare la sopravvivenza e la qualità della vita.
Ma cos’è questa Disulfidptosi?
Qui le cose si fanno interessanti. Recentemente, è stato descritto un nuovo tipo di morte cellulare regolata (RCD), chiamata disulfidptosi. Immaginate una cellula tumorale che, in condizioni di “fame” di glucosio, accumula uno stress eccessivo dovuto ai disolfuri, soprattutto se esprime alti livelli di una proteina chiamata SLC7A11. Questo porta a un accumulo anomalo di legami disolfuro nel citoscheletro di actina, causando una contrazione eccessiva dei filamenti di actina, la rottura della struttura cellulare e, alla fine, la morte della cellula tumorale. Sembra promettente come strategia terapeutica, vero? Soprattutto perché le cellule leucemiche nella LMA sono note per “dirottare” il metabolismo del glucosio a loro vantaggio. Indurre la disulfidptosi potrebbe essere una nuova arma contro la LMA, ma i meccanismi precisi e la sua regolazione in questa malattia sono ancora poco chiari.
Entrano in Scena gli LncRNA: Registi Nascosti del Genoma
Per anni, la ricerca genetica sulla LMA si è concentrata sui geni che codificano per proteine. Ma il nostro genoma è molto più complesso! Stiamo scoprendo sempre più che anche le parti non codificanti, in particolare i lunghi RNA non codificanti (LncRNA), giocano ruoli cruciali nel cancro. Gli LncRNA sono molecole di RNA lunghe più di 200 nucleotidi che non producono proteine, ma agiscono come regolatori maestri dell’espressione genica a vari livelli (epigenetico, trascrizionale, post-trascrizionale), interagendo con DNA, RNA o proteine.
Nella LMA, gli LncRNA sono coinvolti in tantissimi processi: guidano la proliferazione, bloccano la differenziazione cellulare, contribuiscono alla resistenza ai farmaci e possono persino servire come biomarcatori per predire la prognosi. Ad esempio, alcuni LncRNA specifici sono stati collegati a mutazioni particolari o a specifici sottotipi di LMA. Già esistono modelli prognostici basati su LncRNA legati ad altri tipi di morte cellulare, come la cuproptosi. Ma nessuno aveva ancora esplorato il legame tra LncRNA e disulfidptosi nella LMA.

La Mia Ricerca: Sulle Tracce dei DRL nella LMA
Ed è qui che entra in gioco il mio studio. Mi sono chiesto: possiamo identificare degli LncRNA specificamente legati alla disulfidptosi (li chiameremo DRL, Disulfidptosis-Related LncRNAs) che possano aiutarci a predire come andrà un paziente con LMA?
Per farlo, mi sono tuffato nei dati del The Cancer Genome Atlas (TCGA), un enorme database pubblico. Ho analizzato i dati di RNA-seq e clinici di 128 pazienti con LMA (escludendo il sottotipo M3 e i casi con dati incompleti). Ho iniziato identificando 28 geni noti per essere coinvolti nella disulfidptosi (DRG, Disulfidptosis-Related Genes). Curiosamente, questi geni non mostravano molte alterazioni genetiche nei pazienti LMA, suggerendo che le loro sequenze fossero abbastanza stabili, una buona base per il passo successivo.
Usando analisi di correlazione (Pearson), ho cercato gli LncRNA la cui espressione fosse fortemente legata a quella dei DRG. Ne ho trovati ben 344! Un bel numero da cui partire.
Costruire la Sfera di Cristallo: La Firma Prognostica a 7 LncRNA
Avere 344 candidati è tanto. Dovevo restringerli a quelli veramente importanti per la prognosi. Ho diviso i pazienti in un set di addestramento (96 pazienti) e uno di validazione (32 pazienti).
1. Primo Filtro (Analisi Univariata di Cox): Ho cercato quali dei 344 DRL fossero significativamente correlati alla sopravvivenza globale (OS). Ne sono emersi 26, tutti associati a un rischio maggiore.
2. Secondo Filtro (Analisi LASSO): Per evitare che il modello diventasse troppo complesso e “imparasse a memoria” i dati di addestramento (overfitting), ho usato una tecnica chiamata LASSO (Least Absolute Shrinkage and Selection Operator). Questa ha selezionato 12 LncRNA promettenti.
3. Filtro Finale (Analisi Multivariata di Cox): Per costruire il modello definitivo e assicurarmi della sua robustezza, ho applicato un’ulteriore analisi statistica. Risultato? Una firma prognostica composta da 7 specifici LncRNA: FAM30A, MRPL20-AS1, AC022182.2, AL391704.1, LINC01539, AP000439.1, AC113423.1.
Ho quindi creato una formula per calcolare un “punteggio di rischio” per ogni paziente, basato sull’espressione di questi 7 LncRNA.
Alto Rischio vs Basso Rischio: Cosa Ci Dice la Firma?
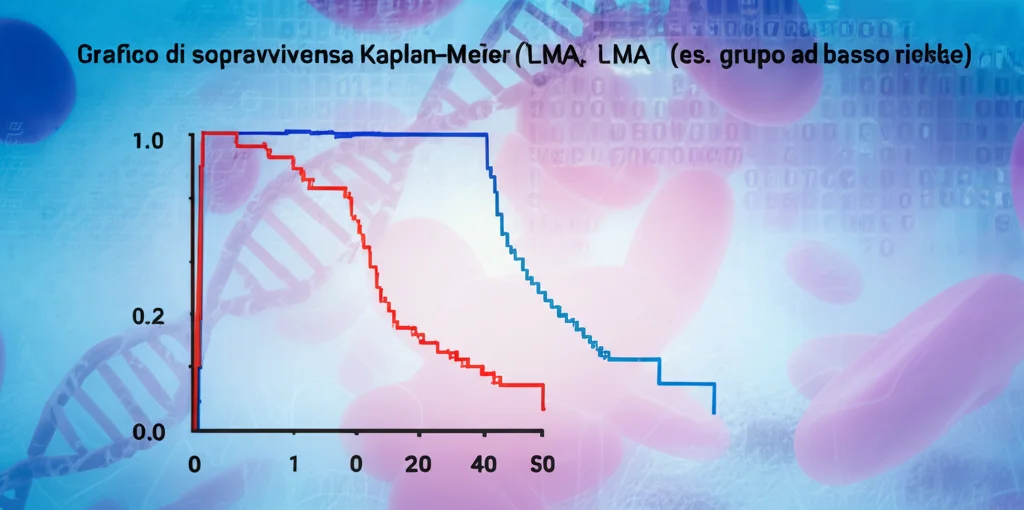
Usando il punteggio di rischio, ho diviso i pazienti del set di addestramento in due gruppi: alto rischio e basso rischio. I risultati sono stati netti:
- Il gruppo ad alto rischio aveva una sopravvivenza globale (OS) significativamente peggiore e tempi di sopravvivenza più brevi.
- Le curve ROC (Receiver Operating Characteristic), che misurano l’accuratezza predittiva, hanno mostrato ottimi valori AUC (Area Under the Curve) a 1, 3 e 5 anni (0.82, 0.88, 0.90), indicando un’elevata capacità predittiva del modello nel set di addestramento.
Ho poi validato il modello sia nel set di validazione che nell’intero set di pazienti, ottenendo risultati consistenti: la firma era in grado di distinguere i pazienti con prognosi diverse anche in gruppi indipendenti, sebbene con AUC leggermente inferiori nel set di validazione (come spesso accade), ma comunque buoni nell’intero set (0.76, 0.80, 0.89 a 1, 3, 5 anni).
Non Solo LncRNA: Fattori Clinici e Validazione nel Mondo Reale
Un modello è ancora più utile se si dimostra indipendente da altri fattori prognostici noti. Ho confrontato il mio punteggio di rischio con fattori clinici come età, sesso, classificazione FAB, percentuale di blasti nel midollo, conta dei globuli bianchi (WBC) e delle piastrine. L’analisi multivariata ha confermato che il punteggio di rischio basato sui 7 LncRNA e l’età erano i due predittori indipendenti più forti della sopravvivenza. Ho anche creato un nomogramma che combina il punteggio di rischio e i fattori clinici per predire la probabilità di sopravvivenza a 1, 3 e 5 anni, e le curve di calibrazione hanno mostrato una buona corrispondenza tra le previsioni e i dati reali.
Analizzando le mutazioni genetiche, ho notato differenze interessanti: nel gruppo ad alto rischio, la mutazione più frequente era nel gene KIT, mentre nel gruppo a basso rischio era in NPM1. Inoltre, il punteggio di rischio era significativamente più alto nei pazienti con più di 60 anni.
Ma la prova del nove è il mondo reale. Ho preso campioni di sangue da 40 pazienti LMA e 10 controlli sani e ho misurato l’espressione dei 7 LncRNA tramite qPCR. I risultati hanno confermato che questi LncRNA erano generalmente più espressi nei campioni LMA rispetto ai controlli. In particolare, FAM30A, MRPL20-AS1 e AC113423.1 erano significativamente sovraespressi nei pazienti. Ho anche verificato che il gene chiave della disulfidptosi, SLC7A11, fosse più espresso nel gruppo ad alto rischio (identificato dal mio modello) e che il rapporto NADP+/NADPH (un indicatore dello stato redox legato alla disulfidptosi) fosse anch’esso significativamente elevato in questo gruppo. Bingo! Questo rafforza la connessione tra la mia firma LncRNA e il meccanismo biologico della disulfidptosi.
Uno Sguardo al Microambiente Immunitario
La disulfidptosi e gli LncRNA non agiscono nel vuoto. Il microambiente tumorale (TME), specialmente la componente immunitaria, è cruciale nella LMA. Ho usato analisi bioinformatiche (GSEA, ssGSEA) per vedere se ci fossero differenze immunologiche tra i gruppi di rischio.
I risultati sono stati illuminanti:
- Il gruppo ad alto rischio mostrava un arricchimento di pathway legati all’attivazione, differenziazione e migrazione delle cellule immunitarie.
- C’era una maggiore infiltrazione di diverse cellule immunitarie nel TME dei pazienti ad alto rischio, tra cui cellule B, neutrofili, cellule T helper follicolari (Tfh), cellule T CD8+ attivate e cellule T CD4+ di memoria centrale.
- Funzioni immunitarie come la presentazione dell’antigene (MHC classe I) e la co-stimolazione delle cellule T erano potenziate nel gruppo ad alto rischio.
- Tuttavia, nonostante questa apparente “attivazione” immunitaria, questi pazienti hanno una prognosi peggiore. Questo suggerisce che l’attività antitumorale di queste cellule potrebbe essere compromessa, o che si instauri un meccanismo di fuga immunitaria.
- Coerentemente con l’ipotesi della fuga immunitaria, i pazienti ad alto rischio mostravano un’espressione significativamente più alta di diversi checkpoint immunitari inibitori, come PD-1 (PDCD1), CTLA4, LAG-3 e altri. Questi sono proprio i bersagli delle moderne immunoterapie (inibitori dei checkpoint).
- L’analisi ESTIMATE ha mostrato che i pazienti ad alto rischio avevano punteggi TME più alti ma anche una maggiore “purezza” tumorale, suggerendo un microambiente complesso che favorisce la crescita del tumore nonostante l’infiltrazione immunitaria.
Implicazioni per le Terapie Personalizzate
Questi risultati non sono solo biologicamente interessanti, ma hanno potenziali implicazioni cliniche enormi. La firma DRL potrebbe non solo predire la prognosi, ma anche indicare quali pazienti potrebbero beneficiare maggiormente dall’immunoterapia con inibitori dei checkpoint, dato che esprimono livelli più alti di questi bersagli.
Inoltre, ho esplorato la sensibilità a diversi farmaci. Usando database come GDSC, ho predetto la risposta potenziale dei pazienti nei due gruppi di rischio:
- I pazienti ad alto rischio sembravano essere meno sensibili ai chemioterapici standard come citarabina e doxorubicina.
- Al contrario, mostravano una maggiore sensibilità a farmaci mirati come imatinib, cyclopamine, rapamicina, sorafenib e WZ-1-84.
Questo suggerisce che la firma DRL potrebbe aiutare a guidare la scelta terapeutica, indirizzando i pazienti ad alto rischio verso terapie alternative o combinate, potenzialmente includendo l’immunoterapia.
Cosa Ci Riserva il Futuro?
Ovviamente, come ogni studio, anche questo ha i suoi limiti. L’analisi si basa sui dati TCGA, che non sono numerosissimi. Serviranno validazioni su coorti più ampie e indipendenti, magari con dati clinici raccolti prospetticamente nel mondo reale. Inoltre, anche se abbiamo mostrato una correlazione tra i 7 LncRNA e i geni della disulfidptosi, i meccanismi molecolari precisi con cui questi LncRNA regolano la disulfidptosi e lo sviluppo della LMA richiedono ulteriori indagini sperimentali in laboratorio.
Nonostante ciò, credo che questo sia il primo studio a costruire una firma prognostica basata sugli LncRNA legati alla disulfidptosi specificamente per la LMA. Abbiamo dimostrato che ha un buon potere predittivo e abbiamo svelato un’interessante connessione con il microambiente immunitario e la sensibilità ai farmaci.
Spero che questo lavoro apra nuove strade per una valutazione prognostica più accurata e per lo sviluppo di strategie terapeutiche davvero personalizzate per i pazienti affetti da questa difficile malattia. La ricerca continua, e ogni passo avanti ci avvicina a comprendere meglio e a combattere più efficacemente la LMA!
Fonte: Springer







