H6PD e AGR2: L’Inaspettata Alleanza nel Cancro al Seno Svelata da un Approccio “Detective” Molecolare
Ciao a tutti! Oggi voglio portarvi con me in un viaggio affascinante nel cuore delle cellule tumorali del seno, un mondo complesso dove le proteine giocano ruoli cruciali, a volte in modi che non ci aspetteremmo mai. Parleremo di un enzima chiamato Esoso-6-fosfato deidrogenasi (H6PD) e di come siamo andati a caccia dei suoi “compagni di squadra” molecolari.
Chi è H6PD e perché ci interessa?
Immaginate il reticolo endoplasmatico (ER) come una sorta di fabbrica super organizzata all’interno della cellula. H6PD lavora proprio lì, catalizzando i primi due passaggi di una via metabolica chiamata via del pentoso fosfato (PPP). Il suo compito principale? Produrre NADPH, una molecola fondamentale, una sorta di “batteria” energetica e antiossidante per la cellula.
Sappiamo che H6PD è importante per processi fisiologici essenziali, come il metabolismo energetico e l’equilibrio redox (la gestione dello stress ossidativo). Finora, l’unico partner di interazione conosciuto di H6PD era un altro enzima, l’11β-idrossisteroide deidrogenasi 1 (11β-HSD1), che usa il NADPH prodotto da H6PD per riattivare i glucocorticoidi (ormoni come il cortisolo). Questo collega lo stato energetico della cellula alla risposta ormonale.
Ma ecco il punto interessante: studi precedenti avevano mostrato che la perdita di H6PD influenzava le proprietà delle cellule di cancro al seno, indipendentemente dalla presenza di 11β-HSD1. Questo ci ha fatto grattare la testa. Come faceva H6PD a esercitare questi effetti senza il suo partner noto? Era forse dovuto a una carenza di NADPH o dei prodotti della via PPP generati da H6PD? O c’era qualcos’altro sotto? Per capirlo, dovevamo scoprire con chi altro interagiva H6PD.
La nostra strategia: BioID, l’investigatore molecolare
Per scovare i nuovi partner di H6PD, abbiamo deciso di usare una tecnica super intelligente chiamata BioID (Biotin Identification), che funziona per prossimità. Immaginatela così: abbiamo “attaccato” a H6PD un enzima speciale (una ligasi della biotina mutata, chiamata BirA*) che è un po’ “distratto”. In presenza di biotina (una vitamina), questo BirA* inizia ad appiccicare etichette di biotina non solo su H6PD, ma anche su tutte le proteine che si trovano nelle sue immediate vicinanze. È come se H6PD andasse in giro con un pennarello indelebile e marchiasse tutti i suoi vicini!
Prima di lanciarci alla scoperta, dovevamo essere sicuri che il metodo funzionasse nel nostro contesto, cioè all’interno del reticolo endoplasmatico. Abbiamo quindi validato l’approccio confermando l’interazione già nota tra H6PD e 11β-HSD1 in cellule appositamente ingegnerizzate. Missione compiuta: il nostro “investigatore” funzionava!

A caccia di indizi nelle cellule di cancro al seno triplo negativo
A questo punto, eravamo pronti per la vera caccia. Abbiamo creato una linea cellulare stabile di cancro al seno triplo negativo (MDA-MB-231) – un tipo di tumore particolarmente aggressivo – che esprimeva la nostra proteina “esca” H6PD-BirA*. Abbiamo coltivato queste cellule in presenza di biotina, permettendo alla nostra ligasi di “etichettare” i vicini di H6PD.
Poi è arrivato il momento clou: abbiamo “pescato” tutte le proteine etichettate con la biotina usando delle perline magnetiche rivestite di streptavidina (che lega fortissimamente la biotina) e le abbiamo analizzate con una tecnica super potente chiamata spettrometria di massa (MS). Questa tecnica ci permette di identificare esattamente quali proteine sono state etichettate.
Le scoperte: un network inaspettato
I risultati sono stati elettrizzanti! L’analisi di spettrometria di massa ha rivelato un intero “interactoma” per H6PD, una rete di potenziali partner. Tra i candidati più promettenti c’erano proteine coinvolte in processi fondamentali all’interno del reticolo endoplasmatico:
- Proteine del ciclo calreticulina/calnexina (importanti per il corretto ripiegamento delle proteine).
- Proteine della risposta a proteine mal ripiegate (Unfolded Protein Response – UPR), un meccanismo di controllo qualità cellulare.
- Proteine chaperone, che aiutano altre proteine ad assumere la loro forma corretta.
Molte di queste proteine erano già note per essere associate al cancro al seno, suggerendo che H6PD potesse essere coinvolto in questi percorsi critici.
Riflettori puntati su AGR2
Tra tutti i candidati, uno ha attirato particolarmente la nostra attenzione: la proteina gradiente anteriore 2 (AGR2). Perché? Primo, perché ha ottenuto un punteggio molto alto nel nostro screening BioID, indicando una forte vicinanza a H6PD. Secondo, perché AGR2 è già nota per essere associata al cancro al seno e alla sua progressione. Poteva essere lei il partner misterioso che cercavamo?
Per approfondire, abbiamo analizzato grandi database di dati genomici e proteomici (TCGA e CPTAC) da pazienti con cancro al seno. Abbiamo scoperto che nei tessuti tumorali dove sia H6PD che AGR2 erano altamente espressi, erano arricchite diverse vie metaboliche e cellulari cruciali per l’aggressività del cancro, tra cui:
- Glicolisi (il modo principale in cui le cellule tumorali ottengono energia rapidamente).
- Metabolismo degli acidi grassi (importante per la costruzione delle membrane cellulari e come fonte energetica).
- Ipossia (risposta a bassi livelli di ossigeno, comune nei tumori).
- Angiogenesi (formazione di nuovi vasi sanguigni per nutrire il tumore).
- Transizione epitelio-mesenchimale (EMT, un processo che permette alle cellule tumorali di diventare mobili e invadere altri tessuti).
Il quadro si faceva sempre più interessante: H6PD e AGR2 sembravano collaborare in processi chiave per la malignità.

La prova del nove: la Co-immunoprecipitazione
BioID è fantastico per trovare vicini, ma non garantisce un contatto fisico diretto. Per confermare che H6PD e AGR2 si “toccano” davvero, abbiamo usato un’altra tecnica chiamata Co-immunoprecipitazione (Co-IP). Abbiamo usato un “amo” molecolare (un anticorpo specifico per H6PD) per pescare H6PD dalle cellule MCF7 (un’altra linea di cancro al seno che esprime naturalmente sia H6PD che AGR2). Se AGR2 fosse stata legata a H6PD, sarebbe stata pescata insieme a lei.
Ebbene sì! Abbiamo trovato AGR2 nei campioni immunoprecipitati con l’anticorpo anti-H6PD, ma non nei controlli. Questa è stata la conferma che H6PD e AGR2 interagiscono fisicamente all’interno delle cellule. Abbiamo anche confermato l’interazione con altre due proteine identificate da BioID, MOGS e CANX, ma non con CALR, suggerendo che quest’ultima fosse probabilmente una vicina “indiretta”.
Cosa fa questa interazione? AGR2 potenzia H6PD!
Ok, interagiscono. Ma cosa significa a livello funzionale? Abbiamo condotto esperimenti per capirlo.
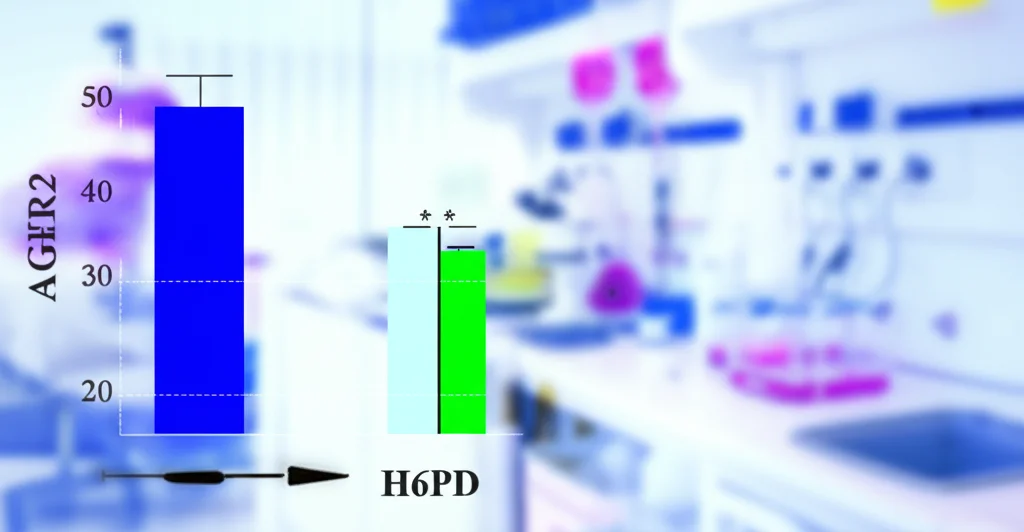
In cellule HEK-293 (una linea cellulare facile da manipolare), abbiamo espresso H6PD da sola o insieme ad AGR2. Abbiamo scoperto che la presenza di AGR2 non cambiava molto la quantità di proteina H6PD, ma ne aumentava significativamente l’attività enzimatica! In pratica, AGR2 sembrava rendere H6PD più efficiente nel produrre NADPH.
Poi abbiamo fatto l’esperimento opposto nelle cellule MCF7: abbiamo “silenziato” il gene AGR2 usando siRNA, riducendone l’espressione. Sorprendentemente, questo ha portato a un aumento dei livelli della proteina H6PD, ma a una diminuzione della sua attività specifica (attività per quantità di proteina). Questo risultato, apparentemente controintuitivo, suggerisce che AGR2 non solo potenzia l’attività di H6PD, ma potrebbe anche essere coinvolta nella regolazione della quantità di enzima *attivo* rispetto a quello totale. Forse AGR2 aiuta H6PD a raggiungere la sua forma più performante o la stabilizza in uno stato attivo.
Conclusioni e prospettive future
Quindi, cosa abbiamo imparato? Abbiamo usato con successo la tecnica BioID per esplorare l’interactoma di H6PD nel reticolo endoplasmatico delle cellule di cancro al seno, un ambiente cellulare complesso da studiare. Abbiamo identificato AGR2 come un nuovo partner di interazione fisica per H6PD. E, cosa ancora più eccitante, abbiamo scoperto che AGR2 non è un partner passivo: regola i livelli di H6PD e ne potenzia l’attività enzimatica.
Questa interazione potrebbe essere molto importante per il cancro al seno. L’aumento dell’attività di H6PD, stimolato da AGR2, potrebbe fornire più NADPH per alimentare vie metaboliche (come la sintesi di acidi grassi o la glicolisi) che favoriscono la crescita e la sopravvivenza delle cellule tumorali, contribuendo a un fenotipo più aggressivo. Potrebbe anche influenzare l’equilibrio redox e la risposta allo stress nel reticolo endoplasmatico, altri aspetti cruciali per le cellule cancerose.
Naturalmente, c’è ancora molto da scoprire. Dobbiamo capire esattamente come AGR2 potenzia l’attività di H6PD (magari aiutandola a ripiegarsi correttamente o modificandola chimicamente?) e quali sono le conseguenze precise di questa interazione potenziata sulle diverse vie metaboliche e di segnalazione nel cancro al seno.
Questo studio non solo svela una nuova affascinante interazione molecolare, ma apre anche nuove strade per la ricerca. Comprendere a fondo il dialogo tra H6PD e AGR2 potrebbe, in futuro, aiutarci a identificare nuovi bersagli terapeutici per contrastare l’aggressività del cancro al seno. La caccia ai segreti delle cellule tumorali continua!
Fonte: Springer