Decifrare USP30: Come abbiamo hackerato una proteina per svelare i segreti dell’inibizione specifica (e perché è importante per il Parkinson)
Ciao a tutti! Oggi voglio raccontarvi una storia affascinante che arriva direttamente dal cuore della ricerca biomedica, un viaggio nel mondo microscopico delle proteine e delle malattie neurodegenerative. Parleremo di come, con un pizzico di ingegneria “proteica” e tanta curiosità, siamo riusciti a svelare i meccanismi molecolari dietro l’inibizione di un enzima cruciale, chiamato USP30, che gioca un ruolo chiave nella regolazione della “pulizia” delle nostre centrali energetiche cellulari, i mitocondri. E perché tutto questo è tremendamente importante per malattie come il Parkinson.
USP30: Il guardiano (un po’ troppo zelante) dei mitocondri
Immaginate i mitocondri come le batterie delle nostre cellule. Quando si danneggiano, devono essere smaltiti correttamente per evitare problemi. Questo processo di pulizia selettiva si chiama mitofagia. Qui entra in gioco USP30, un enzima della famiglia delle deubiquitinasi (DUB). Cosa fa? In pratica, rimuove dei piccoli “tag” molecolari chiamati ubiquitina dai mitocondri. Questi tag sono segnali che dicono “hey, questo mitocondrio è da buttare!”. USP30, togliendo questi segnali, frena la mitofagia.
Ora, in alcune malattie come il Parkinson, specialmente nelle forme legate a mutazioni nei geni PINK1 e parkin, la mitofagia non funziona a dovere. I mitocondri danneggiati si accumulano, specialmente nei neuroni dopaminergici (quelli che muoiono nel Parkinson), causando danni progressivi. L’idea, quindi, è semplice e potente: se riuscissimo a inibire USP30, potremmo “allentare il freno” sulla mitofagia, permettendo alle cellule di ripulirsi meglio dai mitocondri difettosi e proteggendo così i neuroni. Non a caso, ci sono già farmaci inibitori di USP30 in fase di sperimentazione clinica!
La sfida: Capire come funzionano gli inibitori
Sembra facile, no? Blocchiamo USP30 e via. Peccato che, fino ad ora, nessuno sapesse esattamente come questi farmaci inibitori riuscissero a legarsi a USP30 in modo così specifico ed efficace. Perché è importante saperlo? Perché conoscere la struttura tridimensionale del complesso farmaco-proteina è fondamentale per:
- Progettare inibitori ancora migliori, più potenti e specifici.
- Evitare effetti collaterali indesiderati, assicurandosi che il farmaco colpisca solo USP30 e non altre proteine simili (ce ne sono più di 50 nella famiglia USP!).
Il problema è che USP30 è una proteina notoriamente “difficile”: è molto flessibile e poco incline a formare cristalli, un passaggio necessario per determinarne la struttura ai raggi X. Le poche strutture ottenute in passato richiedevano complessi stratagemmi.
L’idea geniale: L’ingegneria delle proteine chimera
Qui entra in gioco la nostra strategia. Ci siamo detti: se USP30 non vuole cristallizzare, perché non renderla un po’ più “collaborativa”? Abbiamo pensato di creare delle versioni “chimera” di USP30. Cosa significa? Abbiamo preso il “cuore” funzionale di USP30 e abbiamo sostituito alcune sue parti periferiche e più flessibili (in particolare la regione delle “dita” o “fingers”) con le regioni corrispondenti di altre proteine della stessa famiglia (USP7, USP14, USP35) che invece sono note per essere più stabili e cristallizzare bene. È un po’ come costruire un modellino LEGO usando pezzi provenienti da set diversi per ottenere una struttura più solida!
Dopo vari tentativi, progettando e testando ben 15 diverse chimere (grazie anche a modelli predittivi come AlphaFold2), abbiamo trovato la candidata perfetta: USP30ch3. Questa chimera, che incorporava pezzi di USP14 e USP35, non solo era più stabile della proteina originale, ma manteneva la sua attività enzimatica e, cosa cruciale, veniva ancora inibita efficacemente dai farmaci noti! Era la nostra chiave per aprire la porta.

Finalmente la struttura: Svelato il legame segreto!
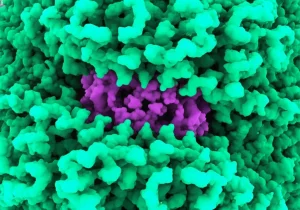
Armati della nostra super-chimera USP30ch3 e di un inibitore specifico (NK036, una versione leggermente modificata e più solubile del composto 39 già in trial clinici), siamo finalmente riusciti a ottenere cristalli di alta qualità. Analizzandoli ai raggi X, abbiamo svelato la struttura tridimensionale del complesso USP30-inibitore con una risoluzione notevole.
E qui la sorpresa! Abbiamo scoperto come l’inibitore si lega a USP30:
- Occupa il sito chiave: Una parte dell’inibitore (l’anello fluorobenzoilico) si infila proprio nella tasca dove normalmente si lega un pezzetto cruciale dell’ubiquitina (l’amminoacido Leucina 73 o Leu73). Questo spiega come l’inibitore blocchi l’attività dell’enzima: impedisce fisicamente all’ubiquitina di legarsi! È un meccanismo di inibizione competitiva.
- Induce una tasca nascosta (criptica): Ma la cosa più affascinante è che un’altra parte dell’inibitore (il gruppo benzensolfonammidico con il tert-butile) si estende in una regione della proteina, nel sottodominio “thumb”, che normalmente non è una tasca! L’inibitore stesso induce un cambiamento conformazionale sorprendente in un’ansa flessibile della proteina, chiamata “switching loop”. Questa ansa cambia forma, si ripiega addirittura formando una piccola elica (mai vista prima in USP30!), e crea una tasca criptica perfetta per accogliere l’inibitore. Questo spiega l’altissima affinità e la specificità del legame, quasi “pseudo-covalente” per quanto è stabile.
Il segreto della specificità: Due amminoacidi unici
Ma perché questo inibitore colpisce quasi esclusivamente USP30 e non le altre decine di proteine USP simili? Analizzando la struttura, abbiamo identificato due “guardiani” molecolari: due amminoacidi nel sito di legame, Leu328 e Phe453, che sono presenti in USP30 ma sono diversi nella stragrande maggioranza delle altre USP (dove di solito troviamo una Fenilalanina e una Tirosina, rispettivamente). Questi due residui unici creano la forma perfetta per accogliere l’inibitore e sono cruciali per la sua specificità. Se provassimo a mettere l’inibitore in un’altra proteina USP, questi residui diversi creerebbero degli “intralci” sterici o elettronici, impedendo un legame efficace.
Per confermare questa ipotesi, abbiamo creato delle versioni mutate di USP30 in cui abbiamo sostituito Phe453 con la Tirosina “canonica”. Risultato? La proteina mutata era molto, molto meno sensibile all’inibitore (la potenza si riduceva di oltre 100 volte!). Bingo! Avevamo trovato le chiavi molecolari della specificità.

Dalla provetta alla cellula: La conferma finale
Ovviamente, era fondamentale verificare che tutto questo avesse senso anche nel contesto biologico reale, cioè dentro le cellule. Abbiamo condotto esperimenti su cellule umane (linee cellulari HeLa e HEK293):
- Abbiamo confermato che l’inibitore NK036 (e il suo parente stretto, il composto 39) aumentava effettivamente l’ubiquitinazione delle proteine mitocondriali target di USP30, segno che l’enzima era inibito.
- Abbiamo dimostrato che, mentre l’inibitore bloccava l’attività di USP30 normale nelle cellule, la versione mutata USP30-F453Y era resistente all’inibitore, proprio come previsto dalla nostra struttura.
- Abbiamo testato altre mutazioni nei residui coinvolti nella formazione della tasca criptica (come Phe157, Ala162, Ile154) e anche queste rendevano USP30 resistente all’inibitore nelle cellule.
Questi esperimenti hanno validato pienamente il meccanismo d’azione e di specificità che avevamo scoperto grazie alla nostra struttura cristallina.
Un quadro più ampio: Verso nuovi farmaci DUB-specifici
Questa ricerca non si limita a spiegare come funziona un particolare inibitore per USP30. Ci offre molto di più:
- Una strategia generalizzabile: L’approccio dell’ingegneria chimera potrebbe essere usato per studiare e cristallizzare altre proteine DUB “difficili”, accelerando la scoperta di farmaci per diverse malattie.
- Un modello per l’inibizione specifica: Abbiamo identificato un principio generale. Molti inibitori di DUB (anche di famiglie diverse come USP e UCH) sembrano “ancorarsi” alla tasca che riconosce la Leu73 dell’ubiquitina (un vero e proprio “hotspot” di legame). La specificità deriva poi da come diverse “estensioni” della molecola inibitrice si adattano ad altre regioni uniche della proteina target, come la nostra tasca criptica in USP30. Il nostro inibitore è particolare perché ha una geometria “tripartita”, occupando ben tre aree distinte, il che spiega la sua eccezionale potenza e specificità.
- Nuove speranze per le malattie neurodegenerative: Comprendere a livello atomico come inibire specificamente USP30 apre la strada alla progettazione razionale di farmaci ancora migliori per il Parkinson e potenzialmente altre condizioni legate alla disfunzione mitocondriale, come alcune malattie renali.

Insomma, partendo da una proteina sfuggente e da un mistero molecolare, siamo riusciti, grazie all’ingegneria proteica, a svelare un meccanismo di inibizione affascinante e complesso. È la dimostrazione di come la ricerca di base, quella che indaga i meccanismi fondamentali della vita a livello molecolare, sia essenziale per sviluppare nuove terapie mirate e più efficaci contro malattie devastanti. La strada è ancora lunga, ma ogni pezzetto di conoscenza che aggiungiamo ci avvicina all’obiettivo.
Fonte: Springer Nature