Citoscheletro Sotto la Lente: Come Abbiamo Svelato i Geni Legati alle Malattie dell’Invecchiamento
Ciao a tutti! Oggi voglio portarvi con me in un viaggio affascinante nel cuore delle nostre cellule, alla scoperta di una struttura tanto fondamentale quanto complessa: il citoscheletro. Immaginatelo come l’impalcatura dinamica delle nostre cellule, una rete intricata di filamenti proteici che non solo dà forma e sostegno, ma partecipa attivamente a una miriade di funzioni vitali.
Per decenni, noi ricercatori abbiamo capito che la natura dinamica del citoscheletro è strettamente legata a eventi di segnalazione che regolano l’attività cellulare, controllando persino processi come l’invecchiamento e le malattie neurodegenerative. Ma come esattamente? E quali geni specifici del citoscheletro sono coinvolti nelle malattie che spesso accompagnano l’avanzare dell’età?
La Sfida: Decifrare il Legame tra Citoscheletro e Malattie Legate all’Età
L’invecchiamento è un processo naturale, ma porta con sé un aumento del rischio per diverse patologie croniche. Pensate a malattie come l’Alzheimer (AD), le malattie cardiovascolari (come la Cardiomiopatia Ipertrofica – HCM, la Malattia Coronarica – CAD, e la Cardiomiopatia Dilatativa Idiopatica – IDCM), il Diabete di Tipo 2 (T2DM). Spesso, superati i 60 anni, ci si ritrova a combattere con più di una di queste condizioni contemporaneamente.
Nonostante l’invecchiamento sia il fattore di rischio numero uno, c’è ancora tanto da scoprire per identificare bersagli terapeutici che possano promuovere un invecchiamento sano e longevo. Qui entra in gioco il citoscheletro. Questa rete, composta principalmente da microfilamenti (actina), filamenti intermedi e microtubuli, è essenziale per:
- Mantenere la forma e l’integrità cellulare
- Generare forze per il movimento cellulare
- Organizzare spazialmente il contenuto della cellula
- Gestire il trasporto intracellulare e processi come la fagocitosi
È logico pensare, quindi, che alterazioni nella sua dinamica o organizzazione possano portare a malattie, dal cancro alla neurodegenerazione. Eppure, nonostante il suo ruolo cruciale, specialmente nei neuroni, sappiamo ancora poco sulla sua partecipazione specifica nell’invecchiamento fisiologico e nelle malattie neurodegenerative.
Studi precedenti avevano già messo in luce alcuni legami. Ad esempio, difetti nel citoscheletro causano miopatie; in pazienti con IDCM si è vista un’alterata espressione di proteine sarcomeriche e citoscheletriche; nell’HCM sono state identificate mutazioni in geni che codificano per proteine del sarcomero o associate ad esso; varianti genetiche in geni che regolano l’assemblaggio del citoscheletro sono state trovate in pazienti con CAD; e nell’Alzheimer, difetti nei microtubuli sembrano causare problemi nel trasporto assonale, contribuendo forse alla perdita di memoria. Anche nel Diabete di Tipo 2, si è notata un’alterazione nell’espressione di proteine legate alla struttura del citoscheletro.
Nonostante questi indizi, restano molte domande aperte sul ruolo preciso della disgregazione del citoscheletro nella patologia di queste malattie. Ecco perché abbiamo deciso di affrontare questa sfida con un approccio innovativo.

Il Nostro Approccio: Machine Learning e Analisi Differenziale per Smascherare i Geni Chiave
Nel nostro studio, abbiamo messo a punto un framework computazionale integrato. Cosa significa? Abbiamo combinato la potenza dei modelli di machine learning (ML) con l’analisi dell’espressione genica differenziale (DEA) per indagare i cambiamenti trascrizionali (cioè, quanto vengono “accesi” o “spenti”) dei geni del citoscheletro e dei loro regolatori in cinque specifiche malattie legate all’età: HCM, CAD, AD, IDCM e T2DM.
L’obiettivo era ambizioso: identificare potenziali biomarcatori basati proprio sui geni del citoscheletro. Per farlo, abbiamo prima recuperato un elenco di ben 2304 geni associati al citoscheletro dal Gene Ontology Browser. Poi, per ciascuna delle cinque malattie, abbiamo “addestrato” diversi algoritmi di machine learning utilizzando i dati di espressione genica di questi geni, provenienti da dataset pubblici (principalmente dal Gene Expression Omnibus – GEO).
Abbiamo testato cinque algoritmi diversi: Alberi Decisionali (DTs), Random Forest (RF), K-Nearest Neighbors (k-NN), Gaussian Naive Bayes (GNB) e, soprattutto, le Support Vector Machines (SVM). E indovinate un po’? L’algoritmo SVM si è dimostrato il più accurato nel classificare correttamente i campioni dei pazienti rispetto a quelli di controllo per tutte e cinque le malattie! Le SVM sono particolarmente adatte per i dati di espressione genica perché gestiscono bene grandi set di dati e identificano pattern complessi.
Ma non ci siamo fermati qui. Avere tanti geni a disposizione non è sempre un vantaggio. Volevamo identificare il set *minimo* ma *più informativo* di geni per ciascuna malattia. Per questo, abbiamo usato una tecnica chiamata Recursive Feature Elimination (RFE), sempre in coppia con il nostro campione, l’SVM. L’RFE è come un setaccio intelligente: rimuove ricorsivamente i geni meno importanti e valuta le prestazioni del modello con i geni rimanenti, fino a trovare il gruppo ottimale.
In parallelo, abbiamo condotto un’analisi di espressione differenziale (DEA) per vedere quali geni del citoscheletro fossero significativamente sovra- o sotto-espressi nei pazienti rispetto ai controlli sani.
I Risultati: Geni del Citoscheletro Come Potenziali Spie delle Malattie
Il momento della verità è arrivato quando abbiamo incrociato i risultati: quali geni erano stati selezionati sia dall’RFE (per la loro capacità predittiva) sia dalla DEA (per la loro significativa alterazione nell’espressione)? Questo incrocio ci ha permesso di focalizzarci sui candidati più promettenti.
Ecco cosa abbiamo scoperto:
- HCM (Cardiomiopatia Ipertrofica): 4 geni chiave (ARPC3, CDC42EP4, LRRC49, MYH6). Tutti sotto-espressi tranne LRRC49. MYH6, in particolare, ha mostrato una capacità discriminatoria eccezionale.
- CAD (Malattia Coronarica): 5 geni (CSNK1A1, AKAP5, TOPORS, ACTBL2, FNTA). Tutti sotto-espressi tranne ACTBL2.
- AD (Alzheimer): Ben 23 geni comuni tra RFE e DEA! Tra i più performanti (con AUC > 0.75 nella validazione esterna) c’erano ENC1, NEFM, ITPKB, PCP4, CALB1. Tutti sotto-espressi tranne ITPKB, che ha raggiunto un AUC di 1.0, indicando una fortissima capacità di distinguere i pazienti AD dai controlli.
- IDCM (Cardiomiopatia Dilatativa Idiopatica): 2 geni (MNS1, MYOT). MNS1 sovra-espresso, MYOT sotto-espresso. Entrambi molto performanti nella validazione.
- T2DM (Diabete di Tipo 2): 1 gene (ALDOB), risultato sovra-espresso e con ottima capacità discriminatoria.
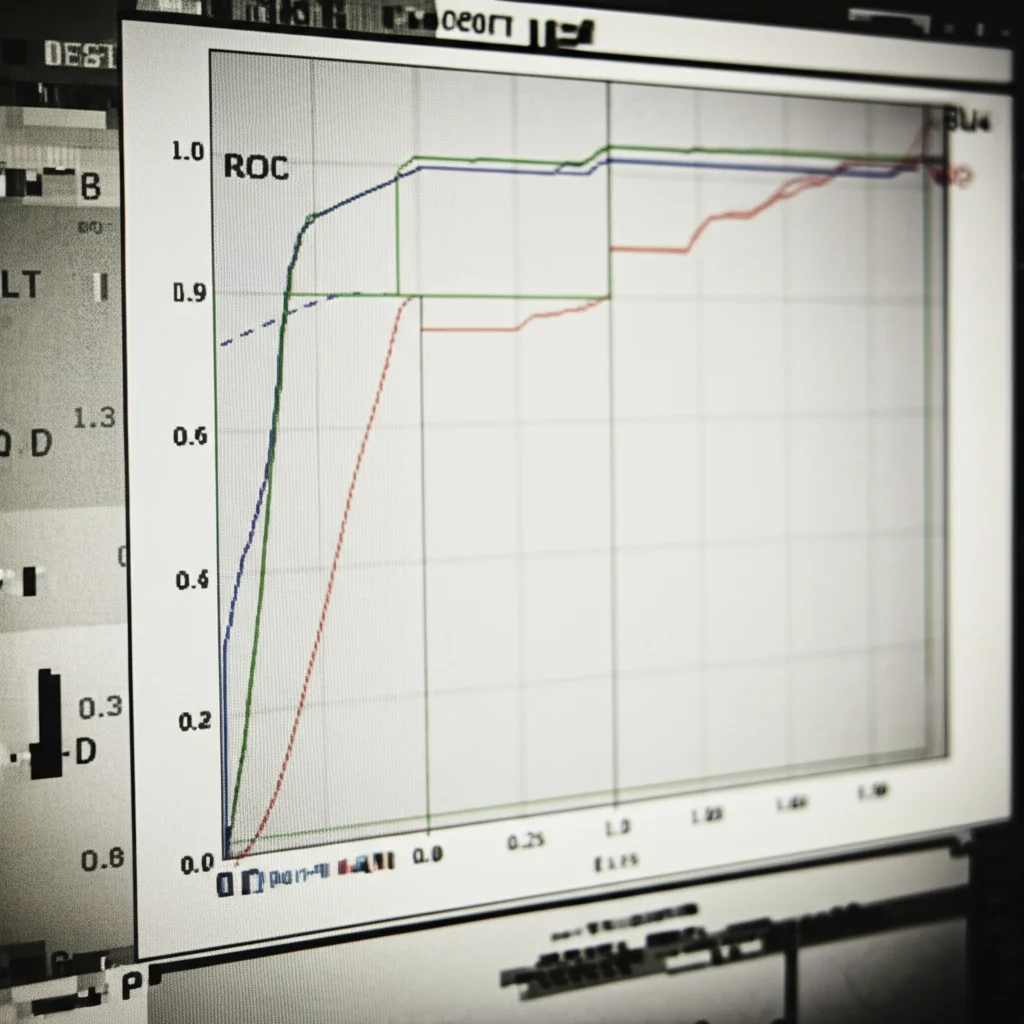
Per essere sicuri che questi geni non fossero solo un “caso” legato ai dati iniziali, abbiamo validato le loro performance usando dataset esterni, indipendenti da quelli usati per costruire i modelli. Abbiamo utilizzato l’analisi ROC (Receiver Operating Characteristic) e calcolato l’area sotto la curva (AUC), un valore che misura quanto bene un gene riesce a distinguere tra pazienti e controlli (un AUC di 1 è perfetto, 0.5 è casuale). I risultati della validazione hanno ampiamente confermato la potenza discriminatoria della maggior parte dei geni identificati!
Discussione: Implicazioni e Prospettive Future
Cosa ci dicono questi risultati? Prima di tutto, confermano in modo robusto l’ipotesi che la disregolazione trascrizionale dei geni del citoscheletro sia fortemente associata a diverse malattie legate all’età. Non si tratta solo di geni strutturali, ma anche di regolatori che influenzano la dinamica di questa complessa rete cellulare.
I geni che abbiamo identificato – come MYH6 per l’HCM, ITPKB per l’AD, ALDOB per il T2DM, MYOT per l’IDCM, o ACTBL2 e AKAP5 per la CAD – hanno tutti legami biologici plausibili con le rispettive patologie, come discusso in letteratura (ruoli nella contrazione cardiaca, funzione sinaptica, metabolismo del glucosio, ecc.). Questo rafforza la validità del nostro approccio computazionale.
L’integrazione del machine learning (in particolare RFE-SVM) con l’analisi di espressione differenziale si è rivelata una strategia potente per “distillare” un piccolo numero di geni altamente informativi da un grande pool iniziale. Questo è fondamentale perché sviluppare strumenti diagnostici o prognostici basati su centinaia di geni è impraticabile. Identificare un set ristretto di potenziali biomarcatori apre invece strade concrete per:
- Diagnosi precoce
- Monitoraggio della progressione della malattia
- Identificazione di nuovi bersagli terapeutici
Certo, il nostro è uno studio computazionale. Il prossimo passo fondamentale sarà la validazione sperimentale in laboratorio (“wet lab”) di questi risultati. Bisognerà confermare il ruolo di questi geni a livello proteico e funzionale nei modelli cellulari e animali delle malattie studiate.
Tuttavia, questo lavoro fornisce una visione d’insieme preziosa e punta i riflettori su specifici attori molecolari – i geni del citoscheletro – che potrebbero essere cruciali nella lotta contro le malattie legate all’invecchiamento. Mantenere l’integrità e la dinamica del citoscheletro potrebbe diventare una nuova frontiera terapeutica per promuovere un invecchiamento più sano.
Il nostro viaggio nel cuore della cellula ci ha mostrato come, combinando biologia e intelligenza artificiale, possiamo iniziare a decifrare alcuni dei misteri più complessi della salute umana. E questo, per me, è l’aspetto più affascinante della ricerca scientifica!
Fonte: Springer