Cancro al Seno: l’IA Decifra HER2 dalle Immagini Standard? La Nostra Scommessa!
Ciao a tutti! Oggi voglio parlarvi di qualcosa che mi appassiona tantissimo e che potrebbe davvero cambiare le carte in tavola nella lotta contro il cancro al seno. Immaginate se potessimo rendere la diagnosi di un fattore cruciale come l’HER2 più semplice, veloce ed economica, usando strumenti che già abbiamo a disposizione. Sembra fantascienza? Forse non del tutto!
La Sfida della Diagnosi HER2
Partiamo dalle basi. Il recettore HER2 (Human Epidermal growth factor Receptor 2) è una proteina super importante nel contesto del cancro al seno. Sapere se un tumore è “HER2-positivo” o meno è fondamentale perché guida le scelte terapeutiche. Circa il 15-20% dei tumori al seno sono HER2-positivi e possono beneficiare enormemente di terapie mirate anti-HER2. Quindi, capite bene quanto sia critica un’identificazione accurata.
Attualmente, per capire lo stato di HER2, ci affidiamo principalmente all’immunoistochimica (IHC) e, in casi dubbi (quelli classificati come 2+), alla FISH (Fluorescence In Situ Hybridization). Questi metodi sono il “gold standard”, ma hanno i loro contro:
- Variabilità: L’interpretazione dei risultati può variare a seconda dell’esperienza del patologo o della qualità delle attrezzature. Questo è un problema serio, specialmente con la nuova categoria “HER2-low” che richiede ancora più precisione.
- Costi e Tempi: Sono esami che richiedono tempo e risorse, non proprio una passeggiata.
Ecco perché da tempo si cerca un modo più snello ed efficiente. E se la risposta fosse nascosta… nelle immagini più comuni?
L’Idea: Sfruttare l’Ematossilina-Eosina (HE) con l’IA
Qui entra in gioco la nostra idea un po’ folle, ma affascinante. E se potessimo “leggere” lo stato di HER2 direttamente dai vetrini colorati con Ematossilina ed Eosina (HE)? Questi sono i vetrini standard, quelli che si usano per la prima analisi morfologica di un tessuto. L’ipotesi di partenza è che le differenze a livello molecolare (come l’espressione di HER2) spesso si traducono in cambiamenti visibili nella forma e nella struttura delle cellule (la morfologia, appunto), anche se non sono evidenziate da colorazioni specifiche come l’IHC.
Negli ultimi anni, l’intelligenza artificiale (IA) e il deep learning hanno fatto passi da gigante nell’analisi delle immagini mediche, specialmente delle “Whole Slide Images” (WSI), che sono le scansioni digitali ad altissima risoluzione dei vetrini istologici. Alcuni studi hanno già provato a usare l’IA sui vetrini HE per predire caratteristiche molecolari, ma spesso si sono scontrati con alcuni limiti:
- Variabilità tra gruppi: Hanno trascurato le differenze *all’interno* dei gruppi HER2-positivo e HER2-negativo (la cosiddetta variabilità intra-classe).
- Validazione limitata: Spesso mancava una validazione su dati provenienti da ospedali e laboratori diversi (multi-coorte), fondamentale per assicurarsi che un modello funzioni davvero ovunque e non solo con i dati su cui è stato allenato.
Il Nostro Studio Multi-Coorte: Mettiamo alla Prova l’IA
Ed eccoci al cuore del nostro lavoro. Abbiamo deciso di affrontare queste sfide raccogliendo un bel po’ di dati: 578 immagini WSI colorate con HE, provenienti da ben cinque coorti diverse. Tre di queste erano set di dati pubblici (così chiunque può verificare i nostri risultati) e due erano privati, provenienti da ospedali con cui collaboriamo. Questa varietà era essenziale per testare la robustezza del nostro approccio.
Abbiamo messo a confronto due principali strategie di intelligenza artificiale:
- Aggregazione basata su sotto-classificatori (con Transfer Learning): L’idea qui è insegnare a un modello (usando una tecnica potentissima chiamata “Transfer Learning”, che permette di trasferire conoscenza da un compito all’altro) a riconoscere piccole porzioni (patch) dell’immagine indicative dello stato HER2. Poi, si aggregano le previsioni di tutte le patch per ottenere un punteggio per l’intera immagine. Abbiamo chiamato il nostro modello specifico TL-PA (Transfer-Learning-based Probabilistic Aggregation).
- Multi-Instance Learning (MIL): Un approccio diverso, molto popolare in patologia computazionale, dove l’immagine intera è vista come un “sacco” di piccole istanze (le patch) e il modello impara a predire lo stato generale basandosi su queste, spesso usando meccanismi di “attenzione” per focalizzarsi sulle patch più rilevanti. Abbiamo testato varianti di MIL, alcune usando anche il Transfer Learning (TL-CLAM, TL-SlideGraph+).
Abbiamo allenato i modelli su una coorte (Cohort A) e poi li abbiamo scatenati sulle altre quattro per vedere come se la cavavano (validazione). Abbiamo usato l’AUC (Area Under the Curve), una metrica standard per valutare quanto bene un modello distingue tra due classi (in questo caso, HER2+ vs HER2-).
I Risultati: TL-PA, il Nostro Asso nella Manica!
Ebbene, i risultati sono stati davvero incoraggianti! Il nostro modello TL-PA ha nettamente superato i modelli basati su MIL. Ha mostrato una performance solida e consistente su tutte le coorti di validazione, ottenendo AUC intorno a 0.75-0.77. Questo significa che è stato bravo a distinguere i casi HER2-positivi da quelli negativi, e lo ha fatto in modo affidabile su dati provenienti da fonti diverse. Un’ottima notizia per la generalizzabilità!
Abbiamo anche cercato di capire se i punteggi dati dal nostro modello avessero un senso rispetto alla classificazione più dettagliata che fanno i patologi (0, 1+, 2+ con FISH-, 2+ con FISH+, 3+) e rispetto alla risposta alla terapia neoadiuvante (NAT), cioè la terapia fatta prima dell’intervento chirurgico.
- Correlazione con lo stato HER2 IHC: Abbiamo trovato una correlazione moderata (coefficiente di Spearman intorno a 0.37). Non perfetta, ma indica che il modello sta cogliendo qualcosa di biologicamente rilevante legato ai livelli di HER2.
- Correlazione con la risposta alla terapia (NAT): In una coorte specifica di pazienti HER2-positivi sottoposti a NAT, il nostro modello ha ottenuto un AUC di 0.77 nel predire chi non avrebbe avuto una risposta completa (non-pCR). Inoltre, i punteggi del modello mostravano una correlazione moderata (coefficiente di Spearman 0.45) con i gradi RCB (Residual Cancer Burden), un altro indicatore di quanta malattia rimane dopo la terapia. Più alto il punteggio del modello, peggiore tendeva ad essere la risposta alla terapia.

Analizzando le “mappe di calore” generate dal modello (che mostrano su quali aree dell’immagine si concentra), abbiamo notato un pattern interessante: l’IA sembrava dare più importanza alle aree centrali delle lesioni maligne, con una probabilità che diminuiva man mano che ci si spostava verso la periferia del tumore. Questo approccio, focalizzato sull’intensità dell’espressione nelle aree tumorali piuttosto che sulla semplice quantità di aree sospette, è più simile a come ragiona un patologo.
Non è Tutto Oro Quello che Luccica: Le Sfide Rimaste
Siamo entusiasti, ma dobbiamo essere onesti: non abbiamo ancora la bacchetta magica. Sebbene il TL-PA sia bravo nella classificazione binaria (positivo/negativo), ha mostrato delle difficoltà nel catturare le sfumature, la famosa variabilità intra-classe. Non è riuscito a distinguere chiaramente i diversi livelli di espressione HER2 (0, 1+, 2+, 3+) in modo netto e ordinato come farebbe un patologo esperto.
In pratica, i punteggi per i diversi livelli all’interno della categoria “positiva” o “negativa” si sovrapponevano parecchio. Questo indica che, sebbene il modello colga il segnale generale, i dettagli più fini gli sfuggono ancora. Probabilmente, questo è dovuto al fatto che abbiamo allenato il modello solo con etichette binarie (positivo/negativo) a livello di intera immagine. Queste etichette sono un po’ “rumorose”, perché non tutte le cellule in un vetrino positivo esprimono HER2 allo stesso modo, e viceversa.
Inoltre, abbiamo notato una leggera “deriva” dei punteggi tra coorti diverse, il che renderebbe difficile stabilire delle soglie universali per una classificazione più granulare.
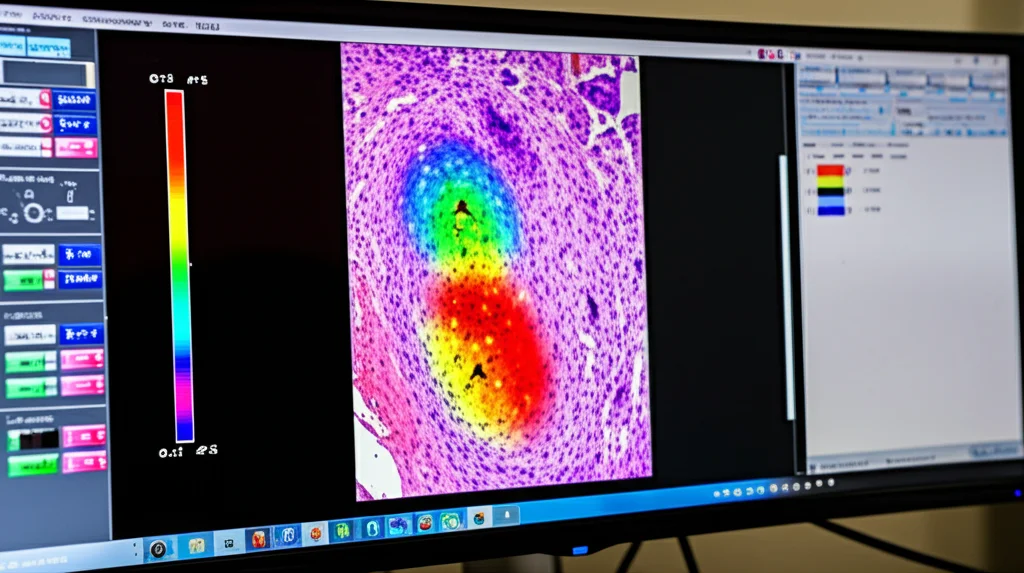
Cosa Ci Riserva il Futuro?
Il nostro studio dimostra che l’idea di usare l’IA sui vetrini HE standard per lo scoring HER2 è fattibile e promettente, soprattutto grazie all’uso del transfer learning e a un’intelligente aggregazione delle informazioni. Il modello TL-PA ha mostrato una buona capacità di generalizzare su dati diversi, il che è un passo avanti importante.
Tuttavia, per rendere questi strumenti davvero affidabili e utili nella pratica clinica quotidiana, dobbiamo fare di più. La strada da percorrere è quella di integrare annotazioni più dettagliate durante l’allenamento dei modelli. Immaginate di poter “dire” all’IA esattamente quali aree corrispondono a un’espressione 1+, quali a 3+, quali sono tessuto normale, ecc. Questo aiuterebbe l’IA a imparare a “ragionare” in modo più simile a un patologo e a cogliere quelle sfumature che ora le sfuggono.
Potrebbe essere utile anche integrare le immagini IHC per guidare l’attenzione del modello o esplorare tecniche come il “federated learning” per gestire meglio le differenze tra i dati provenienti da laboratori diversi senza dover condividere i dati sensibili.
Insomma, la ricerca continua! Siamo convinti che l’intelligenza artificiale abbia un potenziale enorme per supportare i patologi, rendendo le diagnosi più rapide, consistenti e accessibili. Il nostro lavoro è un tassello in questo puzzle affascinante, e non vediamo l’ora di vedere cosa riusciremo a costruire in futuro. Rimanete sintonizzati!

Fonte: Springer