Il Genoma 3D dei Tumori: Un Incredibile Viaggio nel Cuore della Cellula Cancerosa!
Amici appassionati di scienza, preparatevi per un’avventura affascinante! Oggi vi porto con me in un viaggio tridimensionale, non nello spazio cosmico, ma all’interno del nucleo delle nostre cellule, e più precisamente, nel caotico mondo delle cellule tumorali. Avete presente il DNA? Quel lunghissimo filamento che contiene tutte le istruzioni per farci funzionare? Bene, immaginate che in ogni cellula ce ne siano circa due metri! Come fa a stare in un nucleo che è grande appena una decina di micrometri (un centesimo di millimetro)? Semplice, o meglio, incredibilmente complesso: si piega, si avvolge, si organizza in una struttura tridimensionale mozzafiato. E questa architettura 3D non è casuale, anzi, è fondamentale per regolare l’espressione dei geni.
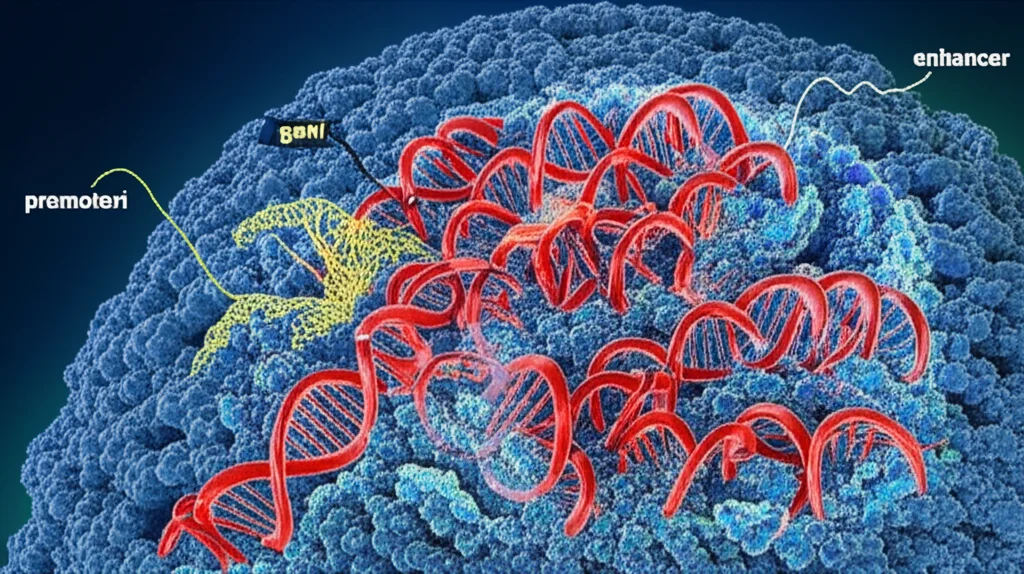
Un Paesaggio Complesso: Compartimenti, TAD e Loop
Pensate al genoma come a una metropoli intricata. Ci sono i “quartieri” principali, chiamati compartimenti A e B. Il compartimento A è come il centro città, pieno di attività, dove i geni sono più attivi (eucromatina). Il compartimento B, invece, è più simile a una periferia tranquilla, con geni meno attivi (eterocromatina). All’interno di questi quartieri, troviamo i “distretti”, noti come domini di associazione topologica (TAD). Immaginateli come isolati urbani: le interazioni avvengono preferenzialmente all’interno di un TAD, mentre sono più rare tra TAD diversi. E poi, ci sono le “strade” che collegano punti specifici: i loop enhancer-promotore (E-P). Gli enhancer sono come degli interruttori a distanza che possono accendere o spegnere i geni (i promotori sono l’interruttore principale vicino al gene), anche se si trovano a decine o centinaia di migliaia di basi di distanza! È un sistema di regolazione finissimo e cruciale.
Cosa Succede nel Cancro? Il Caos Organizzato
Ora, cosa c’entra tutto questo con il cancro? Beh, moltissimo! Le cellule tumorali sono maestre nel sovvertire le regole. Le alterazioni nell’espressione genica, nella metilazione del DNA e nell’accessibilità della cromatina sono all’ordine del giorno nei tumori. Ma capire come gli elementi regolatori a distanza (come gli enhancer) influenzino i loro geni bersaglio è una sfida enorme, un po’ come cercare di capire chi controlla cosa in una città impazzita. Studi precedenti avevano già suggerito che modifiche nella topologia del cromosoma potessero “ricablare” gli enhancer nel cancro, ma mancava una visione d’insieme sistematica dell’architettura 3D dei genomi tumorali primari. E fidatevi, studiare le cellule tumorali direttamente dai pazienti è molto diverso che studiare linee cellulari coltivate in laboratorio per decenni!
Per gettare luce su questo affascinante paesaggio, abbiamo (parlo come se fossi parte del team di ricerca, per farvi sentire più coinvolti!) analizzato l’architettura del genoma tridimensionale e il “connettoma” degli enhancer (cioè come gli enhancer sono collegati ai geni) di 69 campioni tumorali, rappresentanti ben 15 tipi di cancro umano primario, grazie ai dati raccolti dal progetto The Cancer Genome Atlas (TCGA). Per farlo, abbiamo usato una tecnica super potente chiamata HiChIP, che ci permette di vedere contemporaneamente l’attività degli enhancer (marcati da una modifica istonica specifica, H3K27ac) e le loro interazioni con i geni bersaglio.
Archetipi di Utilizzo degli Enhancer e il Ruolo del Microambiente
Cosa abbiamo scoperto? Innanzitutto, abbiamo identificato tre “archetipi” principali di come gli enhancer vengono utilizzati per oltre 100 oncogeni (i geni che, se alterati, possono promuovere il cancro):
- Utilizzo statico: alcuni oncogeni usano sempre gli stessi enhancer, un po’ come avere un interruttore fisso. Un esempio è NRAS.
- Guadagno selettivo: in alcuni tipi di cancro, un oncogene specifico può acquisire nuove connessioni con enhancer. Pensate a EGFR nel glioblastoma.
- Ricablaggio dinamico: altri oncogeni, come il famoso MYC, mostrano schemi di contatti con enhancer altamente variabili e dinamici a seconda del tipo di cancro.
È incredibile vedere come lo stesso oncogene possa essere regolato in modi così diversi! Per esempio, per MYC, in alcuni tumori come quello del colon (COAD), l’attività degli enhancer si concentra a monte del gene, mentre in altri, come il carcinoma epatocellulare (LIHC), si concentra a valle. E queste differenze riflettono spesso l’attività degli enhancer nei tessuti sani di origine.
Ma non è tutto. I tumori non sono solo un ammasso di cellule cancerose; sono ecosistemi complessi, il cosiddetto microambiente tumorale (TME), che include cellule immunitarie, fibroblasti e vasi sanguigni. Grazie all’integrazione dei nostri dati HiChIP con dati di accessibilità della cromatina a singola cellula (scATAC-seq), siamo riusciti a “deconvolvere” il segnale e a capire quali interazioni enhancer-promotore fossero specifiche delle cellule tumorali e quali delle altre cellule nel TME. Ad esempio, abbiamo identificato un’interazione enhancer-promotore per il gene CD274 (che codifica per PD-L1, un importante bersaglio dell’immunoterapia) specifica delle cellule mieloidi (un tipo di cellula immunitaria) nel tumore del polmone. Questo ci dice che l’espressione di PD-L1, e quindi la capacità del tumore di sfuggire al sistema immunitario, può dipendere fortemente dalle cellule immunitarie presenti nel TME!
Varianti Strutturali e DNA Extracromosomico: Quando il Genoma si Sconvolge
I genomi tumorali sono spesso un vero e proprio campo di battaglia, pieni di varianti strutturali (SV): delezioni, duplicazioni, inversioni, traslocazioni. Queste SV possono stravolgere l’organizzazione 3D del genoma, creando nuove interazioni. Immaginate di spostare interi quartieri di una città o di costruire autostrade dove prima non c’erano! Abbiamo visto che le traslocazioni, in particolare, tendono a promuovere un ampio ricablaggio degli enhancer.
E poi c’è un attore particolarmente nefasto: il DNA extracromosomico (ecDNA). Si tratta di piccole molecole circolari di DNA, da 100 kilobasi a 5 megabasi, che si trovano al di fuori dei cromosomi. L’ecDNA può contenere oncogeni amplificati a dismisura e la sua presenza è spesso associata a una prognosi infausta. Abbiamo scoperto che l’ecDNA promuove un ricablaggio degli enhancer ancora più esteso rispetto ad altri meccanismi di amplificazione focale. È come se questi “mini-cromosomi” fluttuanti potessero creare connessioni selvagge, potenziando l’espressione degli oncogeni che trasportano. Ad esempio, abbiamo osservato questo fenomeno per oncogeni come ERBB2 e EGFR.
Mutazioni Non Codificanti: Piccoli Cambi, Grandi Effetti
Non solo le grandi SV, ma anche le singole mutazioni puntiformi non codificanti (cioè quelle che non cadono direttamente dentro un gene, ma in regioni regolatorie come gli enhancer) possono avere un impatto. Abbiamo identificato mutazioni somatiche (acquisite durante la vita, non ereditate) in elementi regolatori attivi che sembrano promuovere l’attività dell’enhancer. Per esempio, una variante nel promotore dell’oncogene MECOM in un tumore dello stomaco mostrava una frequenza allelica molto più alta nei dati HiChIP (che arricchiscono per regioni attive) rispetto al sequenziamento dell’intero genoma (WGS), suggerendo un’attività regolatoria potenziata. Un altro caso affascinante è una variante in un enhancer legato a FGFR1 nel cancro della vescica: questa singola mutazione creava un nuovo sito di legame per un fattore di trascrizione (TFCP2L1), aumentando drasticamente l’interazione enhancer-promotore e l’espressione di FGFR1, senza che ci fossero variazioni nel numero di copie del gene.
Copie Geniche vs. Attività degli Enhancer: Chi Guida l’Oncogene?
Una domanda chiave è: l’iperespressione di un oncogene è dovuta principalmente a un aumento del numero delle sue copie (amplificazione genica) o a un potenziamento dell’attività dei suoi enhancer? Abbiamo usato la regressione lineare multipla per capirlo e, sorpresa (o forse no!), per la maggioranza degli oncogeni, l’attività degli enhancer e il loro ricablaggio spiegano meglio l’aumento dell’espressione dell’mRNA rispetto al semplice aumento del numero di copie del gene. Certo, per alcuni oncogeni, come KRAS, l’amplificazione del numero di copie è il meccanismo dominante, ma per molti altri, come MET, è il “software” regolatorio (gli enhancer) a fare la differenza, più che l’ “hardware” (il numero di copie).
Implicazioni e Sguardo al Futuro
Questo studio ci fornisce una prima, ma incredibilmente ricca, mappa dell’architettura 3D del genoma e del panorama degli enhancer in 15 tipi di cancro umano. Abbiamo visto che, mentre l’organizzazione generale in compartimenti e TAD è largamente conservata, sono i loop enhancer-promotore a variare sostanzialmente e a distinguere i diversi tipi di cancro. È come se l’impianto urbanistico di base delle città (i TAD) rimanesse simile, ma le connessioni specifiche e il traffico (i loop E-P) cambiassero radicalmente.
Comprendere come le alterazioni nella regolazione genica impattino il cancro apre nuove prospettive. Identificare mutazioni non codificanti funzionali o capire i meccanismi di ricablaggio degli enhancer potrebbe, in futuro, guidare la profilazione clinica per identificare pazienti ad alto rischio o candidati a terapie mirate. Pensateci: se sappiamo che un particolare “interruttore” è rotto e tiene un oncogene sempre acceso, forse un giorno potremo sviluppare farmaci per riparare quell’interruttore o per bloccare quella specifica connessione.
Inoltre, la capacità di distinguere le interazioni specifiche delle cellule maligne da quelle delle cellule del microambiente tumorale è cruciale. Abbiamo visto che le cellule mieloidi contribuiscono molto all’espressione di checkpoint immunitari come PD-L1. D’altro canto, i loop E-P specifici delle cellule maligne si intersecano spesso con varianti genetiche (SNP) che predispongono al cancro, suggerendo meccanismi autonomi della cellula tumorale per questi alleli di rischio.
Il viaggio nell’architettura 3D del genoma tumorale è appena iniziato, ma è già chiaro che questo “paesaggio” nascosto gioca un ruolo fondamentale nell’eziologia e, speriamo, un giorno anche nella terapia del cancro. Così come la scoperta degli oncogeni mutati ha portato a terapie mirate, la comprensione dei circuiti regolatori tridimensionali del genoma potrebbe spianare la strada a strategie terapeutiche completamente nuove. Non è affascinante?
Fonte: Springer