Svelati i Segreti del Triptolide: Come i Geni WRKY Dirigono l’Orchestra nella Pianta Miracolosa *Tripterygium wilfordii*
Ciao a tutti gli appassionati di scienza e scoperte! Oggi voglio portarvi con me in un viaggio affascinante nel cuore di una pianta dalle proprietà straordinarie, la *Tripterygium wilfordii*. Magari il nome non vi dice molto, ma in Cina è conosciuta come “Lei Gong Teng”, il “Rampicante del Dio del Tuono”, un nome che la dice lunga sulla sua potenza! Questa pianta è usata da secoli nella medicina tradizionale per trattare reumatismi e problemi della pelle, ma nasconde un tesoro ancora più prezioso: il triptolide.
Cos’è il Triptolide e Perché è Così Speciale?
Scoperto nel 1972, il triptolide è un composto chimico dalla struttura complessa (un diterpenoide triepossido, per i più tecnici) che ha mostrato incredibili proprietà anti-infiammatorie e anti-tumorali. Pensate che è considerato uno dei prodotti naturali più potenti mai scoperti in questo campo, con un’efficacia da 100 a 200 volte superiore agli estratti grezzi della pianta! La ricerca moderna sta esplorando il suo potenziale contro una miriade di malattie: artrite reumatoide, lupus, asma, rigetto nei trapianti e persino diversi tipi di cancro, in particolare quello pancreatico.
Ma c’è un problema, un grosso problema: la pianta ne produce pochissimo! Parliamo di quantità infinitesimali (6-16 nanogrammi per grammo di pianta). Questo rende difficile studiarlo a fondo e, soprattutto, utilizzarlo su larga scala per sviluppare nuovi farmaci. Ecco perché capire come la pianta produce il triptolide è fondamentale. Se riusciamo a svelare i meccanismi genetici che ne regolano la sintesi, potremmo trovare modi per aumentarne la produzione, sia nella pianta stessa che attraverso sistemi biotecnologici.
Entrano in Scena i Fattori di Trascrizione WRKY
Qui entra in gioco il mio campo di ricerca preferito: la genetica e la regolazione dell’espressione genica. In particolare, ci siamo concentrati su una grande famiglia di “interruttori” genetici chiamati fattori di trascrizione WRKY. Queste proteine sono come dei direttori d’orchestra all’interno della cellula vegetale: si legano a specifiche sequenze del DNA e decidono quali geni devono essere “accesi” o “spenti”. Sono cruciali per la crescita della pianta, la sua risposta agli stress (come attacchi di patogeni o siccità) e, cosa che ci interessa di più, per la regolazione del metabolismo secondario, cioè la produzione di quelle molecole complesse come il triptolide.
Sappiamo da studi su altre piante che i WRKY sono coinvolti nella produzione di terpenoidi (la classe di molecole a cui appartiene il triptolide). Ad esempio, in altre specie, attivare certi geni WRKY aumenta la produzione di composti simili. Ma nel caso specifico del triptolide in *T. wilfordii*, il ruolo dei WRKY era ancora un mistero. Fino ad ora!
La Mappa del Tesoro Genetico: Identificare i WRKY in *T. wilfordii*
Il primo passo è stato scandagliare l’intero genoma di *T. wilfordii*, che per fortuna è stato recentemente sequenziato. Utilizzando strumenti bioinformatici specifici (come il programma ITAK), siamo andati a caccia di tutti i geni che presentassero la firma caratteristica dei WRKY, un dominio proteico specifico (chiamato PF03106). E ne abbiamo trovati ben 95! Li abbiamo chiamati *TwWRKY* (Tw sta per *Tripterygium wilfordii*).
Questi 95 geni non sono distribuiti a caso sui cromosomi della pianta, ma si trovano su 22 dei 23 cromosomi (tranne il cromosoma 5), con alcuni cromosomi (come il 4, 10, 13, 18 e 21) che ne ospitano di più, specialmente il 18 che ne ha ben 11. Analizzando le sequenze delle proteine codificate da questi geni, abbiamo visto che sono piuttosto “conservate”, cioè simili a quelle di altre piante. La maggior parte presenta la sequenza amminoacidica “WRKYGQK” nel punto chiave, con una sola eccezione che ha “WRKYGKK”. Questa piccola variazione potrebbe influenzare il modo in cui la proteina si lega al DNA.

Per capire meglio le relazioni tra questi 95 geni *TwWRKY*, abbiamo costruito un albero filogenetico, una sorta di albero genealogico molecolare, confrontandoli anche con i geni WRKY di un’altra pianta modello, l’Arabidopsis thaliana. Questo ci ha permesso di dividerli in tre gruppi principali (Gruppo I, II e III), come avviene anche in altre specie. Il Gruppo II è il più numeroso (56 geni, quasi il 60%) ed è ulteriormente suddiviso in 5 sottogruppi (II-a, II-b, II-c, II-d, II-e). Questa classificazione non è solo un esercizio accademico: geni nello stesso gruppo o sottogruppo spesso hanno funzioni simili, quindi ci aiuta a ipotizzare quali *TwWRKY* potrebbero essere coinvolti in processi specifici, come la biosintesi del triptolide. Abbiamo anche analizzato i “motivi” conservati, brevi sequenze proteiche ricorrenti, scoprendo che alcuni motivi sono specifici di certi gruppi, suggerendo funzioni specializzate.
L’Evoluzione dei WRKY: Duplicazioni e Relazioni
Come si è evoluta questa grande famiglia di geni? Un meccanismo chiave nell’evoluzione dei genomi è la duplicazione genica. A volte, per errori durante la replicazione del DNA, intere porzioni di cromosoma o singoli geni vengono duplicati. Questi geni “extra” possono poi evolvere nuove funzioni. Analizzando il genoma di *T. wilfordii*, abbiamo trovato prove di numerose duplicazioni segmentali (98 coppie di geni *TwWRKY* derivanti da duplicazioni di ampi blocchi cromosomici) e anche 5 casi di duplicazioni tandem (geni duplicati uno accanto all’altro). Questo suggerisce che la duplicazione ha giocato un ruolo fondamentale nell’espansione e nella diversificazione della famiglia *TwWRKY* in questa pianta.
Abbiamo anche confrontato la posizione dei geni *TwWRKY* con quella dei geni WRKY in altre cinque specie vegetali, sia dicotiledoni (come pioppo, Arabidopsis, salvia, vite) che monocotiledoni (mais), alcune più vicine evolutivamente a *T. wilfordii*, altre più lontane. Abbiamo trovato molte corrispondenze (regioni di colinearità), indicando un’origine ancestrale comune per molti di questi geni e confermando l’importanza delle duplicazioni su larga scala (come la duplicazione dell’intero genoma, WGD) nell’evoluzione di questa famiglia.
Dove e Quando si Esprimono i *TwWRKY*? Indizi sulla Funzione
Identificare i geni è solo il primo passo. Per capire cosa fanno, dobbiamo vedere dove e quando vengono attivati (“espressi”) nella pianta. Sappiamo che il triptolide si accumula principalmente nella corteccia delle radici. Quindi, se un gene *TwWRKY* è coinvolto nella sua produzione, ci aspetteremmo di trovarlo particolarmente attivo proprio lì.
Abbiamo analizzato dati di trascrittomica (che misurano l’espressione di tutti i geni) provenienti da diverse parti della pianta: foglie, corteccia della radice, floema della radice, xilema della radice, corteccia del fusto, fusto senza corteccia e fiori. I risultati sono stati illuminanti! Quasi tutti i 95 geni *TwWRKY* erano espressi, ma con livelli molto diversi a seconda del tessuto. Ben 20 *TwWRKY* mostravano la loro massima espressione proprio nella corteccia della radice, in perfetta coerenza con l’accumulo di triptolide! Altri erano più attivi nella corteccia del fusto o nel fusto senza corteccia. Questo ci dà un forte indizio su quali geni potrebbero essere i regolatori che stiamo cercando.

La Rete di Correlazione: Collegare Geni e Metaboliti
Per stringere ulteriormente il cerchio, abbiamo fatto un passo avanti. Abbiamo preso i dati di espressione dei geni *TwWRKY* nei diversi tessuti e li abbiamo messi in relazione con i livelli di 32 composti chimici trovati nelle radici, che si pensa siano intermedi o prodotti secondari della via biosintetica del triptolide (incluso il triptolide stesso e un suo precursore, il triptofenolide). Abbiamo calcolato i coefficienti di correlazione di Pearson: in pratica, abbiamo cercato quali geni *TwWRKY* avessero un andamento di espressione simile all’andamento dell’accumulo di questi composti nei vari tessuti.
Abbiamo costruito una “rete di regolazione gene-metabolita”. Immaginate una mappa con i geni e i composti come nodi, e delle linee che li collegano se la loro correlazione è forte (coefficiente > 0.7). Sono emersi 32 geni *TwWRKY* fortemente correlati con i composti legati al triptolide. Analizzando a quali gruppi filogenetici appartenevano questi 32 candidati, abbiamo notato una predominanza del Gruppo II-c (8 geni), seguito dal Gruppo I e II-b (6 geni ciascuno). Questo suggerisce che i membri del Gruppo II-c potrebbero essere particolarmente importanti per la regolazione del triptolide.
Due geni in particolare sono balzati all’occhio: TW06G00089.1 e TW23G00056.1. Questi due mostravano correlazioni positive molto forti con quasi tutti i 32 composti, inclusi triptolide e triptofenolide (correlazioni superiori a 0.76!). Sembrano davvero dei candidati promettenti come regolatori chiave.
L’Effetto del Metil Jasmonato (MeJA): Stimolare la Produzione
C’è un altro modo per studiare la regolazione: usare degli “elicitori”, sostanze che stimolano la pianta a produrre metaboliti secondari, spesso come meccanismo di difesa. Il metil jasmonato (MeJA) è un ormone vegetale noto per indurre la produzione di terpenoidi e anche per influenzare l’espressione dei geni WRKY in molte piante.
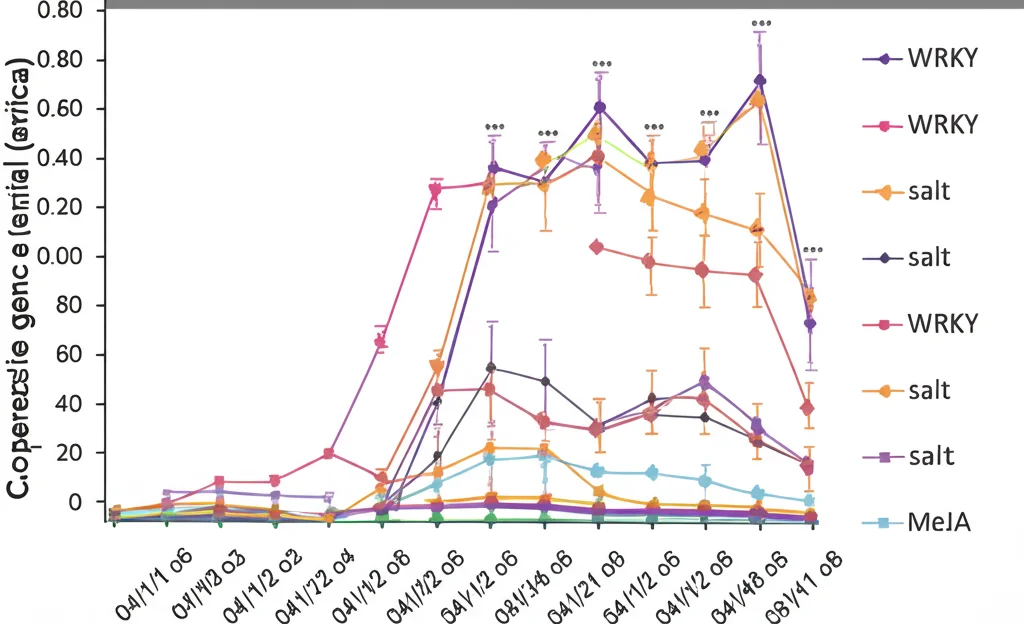
Abbiamo quindi analizzato dati di trascrittomica provenienti da colture di cellule di *T. wilfordii* in sospensione, trattate con MeJA per diversi periodi di tempo (0, 4, 12, 24 e 48 ore). Sapevamo da studi precedenti che dopo 48 ore di trattamento con MeJA, i livelli di triptolide e triptofenolide aumentano significativamente. Ci aspettavamo quindi che i geni WRKY che regolano questa via rispondessero al MeJA ancora prima.
E così è stato! Abbiamo monitorato l’espressione dei nostri 32 candidati *TwWRKY* dopo l’induzione con MeJA. I risultati?
- 11 geni sono stati significativamente sovraregolati (la loro espressione è aumentata) dopo il trattamento. Tra questi, cinque (TW03G01107.1, TW04G00647.1, TW13G00001.1, TW17G00083.1 e il nostro già noto TW23G00056.1) sembrano i candidati più probabili come regolatori positivi della biosintesi del triptolide.
- 13 geni sono stati invece sottoregolati (la loro espressione è diminuita). Alcuni di questi (come TW02G00847.1, TW06G00252.1, TW07G00767.1, TW12G00751.1 e anche TW04G00076.1, che avevamo ipotizzato potesse avere un ruolo negativo basandoci sulla filogenesi) potrebbero agire come regolatori negativi.
Dentro la Cellula: Dove Agisce TW23G00056.1?
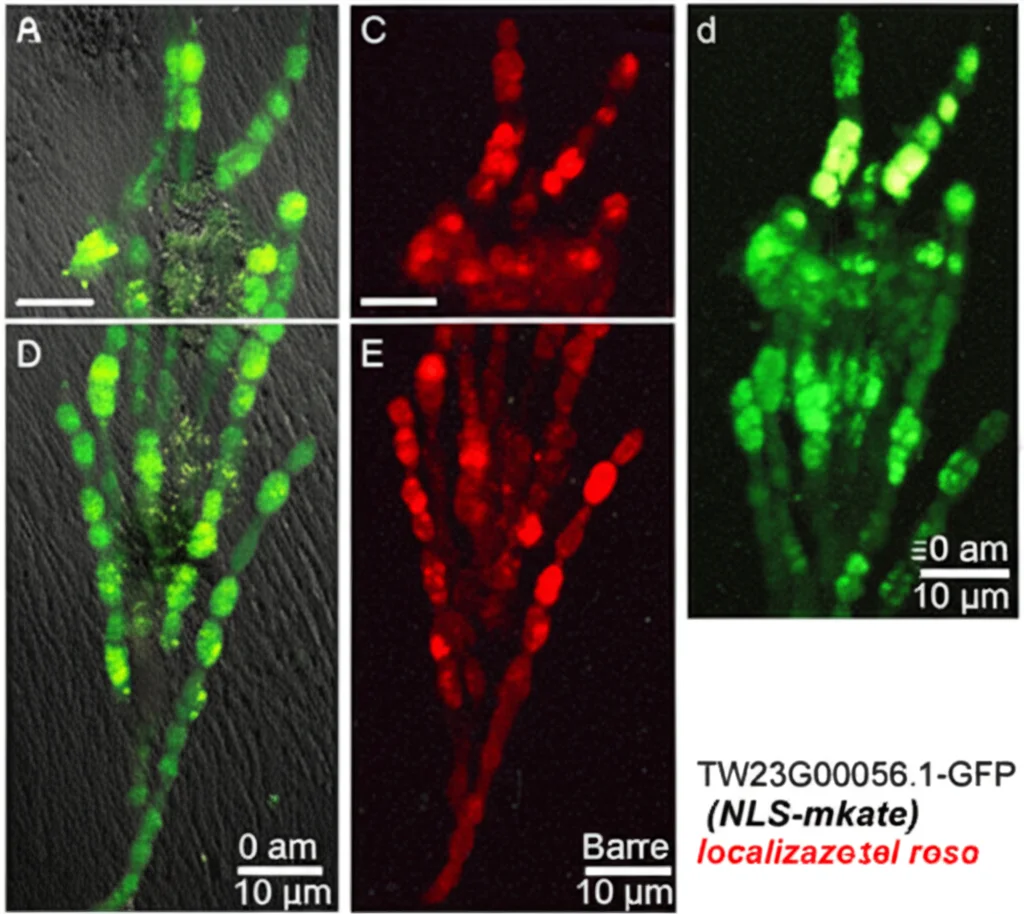
Avevamo identificato *TW23G00056.1* come un candidato super interessante: forte correlazione con il triptolide e i suoi precursori, e sovraregolato dal MeJA. Ma dove svolge il suo lavoro questo fattore di trascrizione all’interno della cellula? I fattori di trascrizione di solito agiscono nel nucleo, dove si trova il DNA da regolare.
Per verificarlo, abbiamo condotto un esperimento di localizzazione subcellulare. Abbiamo preso la sequenza codificante di *TW23G00056.1* e l’abbiamo fusa con un gene che produce una proteina fluorescente verde (GFP). Abbiamo poi inserito questa costruzione genetica in protoplasti di Arabidopsis (cellule vegetali senza parete cellulare). Come controllo, abbiamo usato solo la GFP (che si distribuisce in tutta la cellula) e un marcatore specifico per il nucleo (NLS-mkate, fluorescente rosso). Osservando le cellule al microscopio a fluorescenza, abbiamo visto chiaramente che la fluorescenza verde della proteina di fusione TW23G00056.1-GFP si sovrapponeva perfettamente alla fluorescenza rossa del marcatore nucleare. Bingo! TW23G00056.1 agisce proprio nel nucleo, confermando la sua probabile funzione di regolatore trascrizionale dei geni coinvolti nella biosintesi del triptolide, che sono sparsi su diversi cromosomi ma vengono orchestrati proprio dal nucleo.
Conclusioni e Prospettive Future: Verso Più Triptolide?
Insomma, questo studio ci ha permesso di fare un bel passo avanti nella comprensione di come la *Tripterygium wilfordii* produce il prezioso triptolide. Abbiamo:
- Identificato l’intera famiglia dei geni *TwWRKY* (95 membri).
- Analizzato la loro evoluzione e classificazione.
- Studiato la loro espressione nei diversi tessuti e in risposta allo stimolo con MeJA.
- Costruito reti di correlazione per collegare l’espressione dei *TwWRKY* all’accumulo di metaboliti.
- Identificato un gruppo di 32 candidati regolatori, con alcuni particolarmente promettenti come *TW23G00056.1*.
- Confermato che almeno uno di questi candidati chiave agisce nel nucleo.
Certo, decifrare completamente il meccanismo regolatorio della biosintesi del triptolide è ancora una sfida complessa. Ma i geni candidati che abbiamo identificato forniscono una base solida per ricerche future. Il prossimo passo sarà verificare sperimentalmente la funzione di questi geni, ad esempio modificandone l’espressione nella pianta o in colture cellulari per vedere se questo aumenta effettivamente la produzione di triptolide.
L’obiettivo finale? Sfruttare queste conoscenze per migliorare geneticamente la *T. wilfordii* o per ingegnerizzare microrganismi in grado di produrre triptolide in quantità maggiori e in modo più sostenibile. Sarebbe un passo enorme per rendere questo potente composto più accessibile per lo sviluppo di nuove terapie contro malattie gravi. La strada è ancora lunga, ma aver identificato questi “direttori d’orchestra” genetici è un inizio davvero promettente!
Fonte: Springer