Flavonoidi dall’Erythrina: La Natura Potrebbe Avere la Chiave Contro SARS-CoV-2?
Ragazzi, la ricerca non si ferma mai, vero? Soprattutto quando si parla di trovare nuove armi contro bestiacce come il SARS-CoV-2, il virus che ha messo sottosopra le nostre vite con la pandemia di COVID-19. Da scienziato (o almeno, appassionato che si cala nel ruolo!), vi racconto di un’avventura affascinante nel mondo della biochimica computazionale, una sorta di caccia al tesoro digitale alla ricerca di molecole promettenti. E indovinate dove siamo andati a cercare? Nel regno vegetale, specificamente nel genere Erythrina, piante tropicali e subtropicali note per essere una miniera di flavonoidi.
Perché proprio l’RdRp? Il tallone d’Achille del Virus
Il SARS-CoV-2, per replicarsi e diffondersi nel nostro corpo, ha bisogno di copiare il suo materiale genetico (l’RNA). Per farlo, usa un enzima chiave chiamato RNA-dependent RNA polymerase (RdRp). Immaginate il virus come una fotocopiatrice impazzita che sforna copie di sé stessa: ecco, l’RdRp è il motore di quella fotocopiatrice. Bloccare l’RdRp significa inceppare la macchina e fermare la replicazione virale. Non a caso, farmaci già noti come il Remdesivir (approvato dall’OMS per certi casi di COVID-19) agiscono proprio su questo bersaglio. La nostra idea? Trovare composti naturali, magari più efficaci o con meno effetti collaterali, che facciano lo stesso lavoro.
La Nostra Strategia: Setacciare la Natura con il Computer
Abbiamo preso in esame ben 473 flavonoidi isolati dalle piante del genere Erythrina. Ma come capire se potevano funzionare contro l’RdRp senza passare anni in laboratorio? Qui entra in gioco la potenza della simulazione computazionale!
- Virtual Screening (Docking Molecolare): È come organizzare un gigantesco “speed date” digitale. Abbiamo preso la struttura 3D dell’enzima RdRp (ottenuta da database pubblici come il Protein Data Bank) e, usando software specifici (AutoDockTools, Raccoon), abbiamo simulato come ciascuno dei 473 flavonoidi si “incastrasse” nel sito attivo dell’enzima. L’obiettivo era misurare l'”affinità di legame” (binding energy): più è forte (valore più negativo), meglio è.
- Filtro di Lipinski e ADMET: Non basta che una molecola si leghi bene! Deve anche avere caratteristiche da potenziale farmaco (“drug-likeness”). Abbiamo usato la Regola dei Cinque di Lipinski per verificare parametri come peso molecolare, solubilità e capacità di formare legami idrogeno. Poi, con strumenti come SwissADME e pkCSM, abbiamo predetto l’ADMET (Assorbimento, Distribuzione, Metabolismo, Escrezione, Tossicità): come verrebbe assorbita dal corpo, dove andrebbe, come verrebbe trasformata ed eliminata, e se potrebbe essere tossica.
- Simulazioni di Dinamica Molecolare (MD): Per i candidati più promettenti, siamo andati oltre. Le simulazioni MD (usando software come Amber) ci permettono di vedere come la molecola e l’enzima interagiscono e si muovono nel tempo, a livello atomico, in un ambiente che simula quello del nostro corpo (acqua, sali, temperatura fisiologica). È come girare un film microscopico per capire la stabilità e la dinamica del legame.
Risultati Elettrizzanti: Oltre 100 Candidati Promettenti!
Il primo round, il virtual screening, è stato un successo! Ben 128 flavonoidi hanno mostrato un’energia di legame con l’RdRp migliore di quella del Remdesivir. Tra questi, composti come l’abyssinoside A e B hanno mostrato i punteggi migliori in assoluto. Tuttavia, quando siamo passati al filtro di Lipinski, questi “campioni” hanno mostrato qualche problema (peso molecolare troppo alto, troppi donatori/accettori di legami idrogeno), suggerendo che potrebbero avere difficoltà a diventare farmaci orali efficaci. Ma non ci siamo persi d’animo!

La Star dello Studio: Buteina (119)
Continuando a setacciare i dati ADMET tra i 128 candidati, una molecola ha iniziato a brillare più delle altre: la Buteina (composto 119). Questo flavonoide (un calcone, per la precisione) non solo aveva una buona energia di legame, ma rispettava la Regola di Lipinski e, soprattutto, mostrava un profilo ADMET davvero interessante:
- Buon assorbimento intestinale previsto.
- Non sembrava essere un substrato problematico per le proteine di efflusso (P-glicoproteina) che possono ridurre l’assorbimento dei farmaci.
- Profilo metabolico gestibile (previsto come inibitore solo del CYP1A2, che potrebbe avere anche effetti chemoprotettivi).
- Bassa tossicità prevista: Non mutageno (test AMES negativo), non epatotossico, non cardiotossico (nessun blocco previsto dei canali hERG I o II). Questo è un punto cruciale! Un dato supportato anche da studi pregressi su ratti.
- Buona biodisponibilità potenziale (bassa clearance totale prevista).
Insomma, la Buteina sembrava avere tutte le carte in regola per essere un candidato farmaco sicuro ed efficace.
Un Meccanismo d’Azione Sorprendente
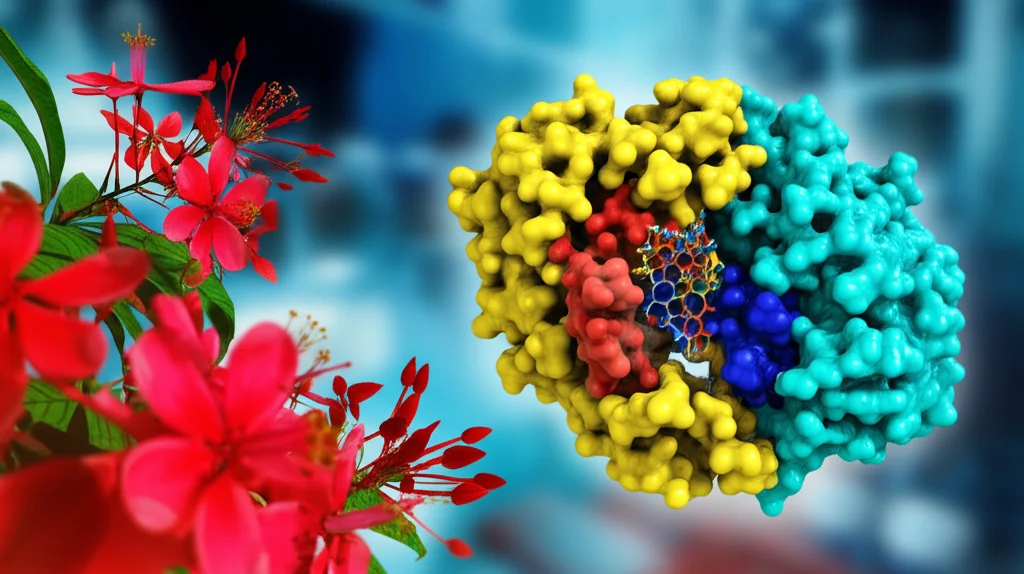
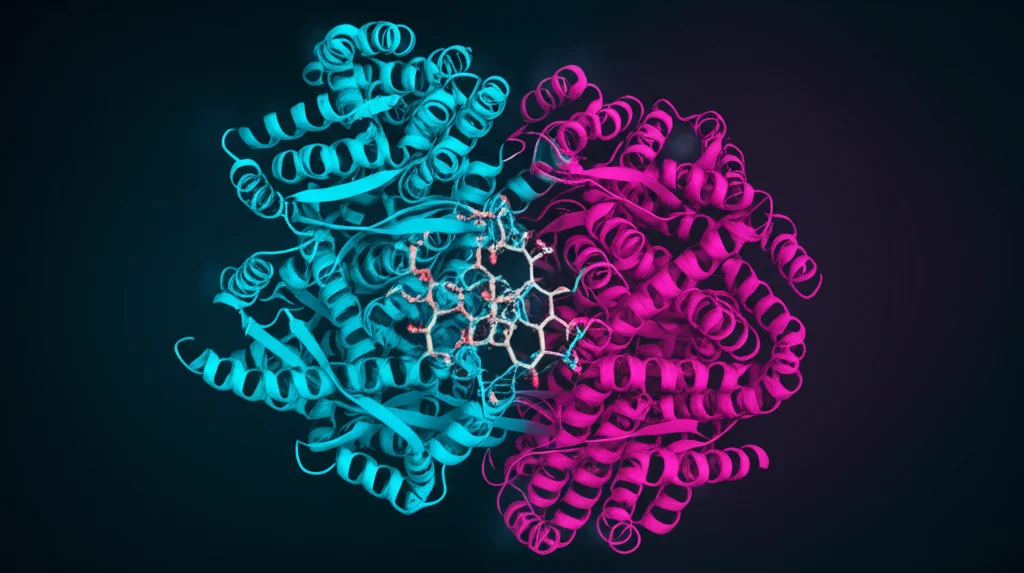
Ma come agisce la Buteina? Le simulazioni di docking e, soprattutto, quelle di dinamica molecolare (MD) per 250 nanosecondi ci hanno riservato una sorpresa. A differenza del Remdesivir, che si lega in un sito specifico dell’RdRp, la Buteina si è posizionata in un punto diverso, interagendo non solo con gli amminoacidi dell’enzima (nsp12, in particolare nel “palmo” o palm subdomain, una regione cruciale per la catalisi), ma anche direttamente con le basi dell’RNA stampo e dell’RNA primer (i filamenti che l’enzima sta copiando).
Immaginate che l’RdRp stia facendo scorrere i filamenti di RNA per copiarli: la Buteina sembra infilarsi proprio lì in mezzo, stabilizzandosi con legami idrogeno e interazioni idrofobiche sia con l’enzima che con l’RNA (in particolare con le basi U12, A13, A14 dello stampo e U18, U19, U20 del primer). Questo meccanismo “multi-sfaccettato” potrebbe letteralmente bloccare l’allungamento della catena di RNA, fermando la replicazione virale in modo efficace.
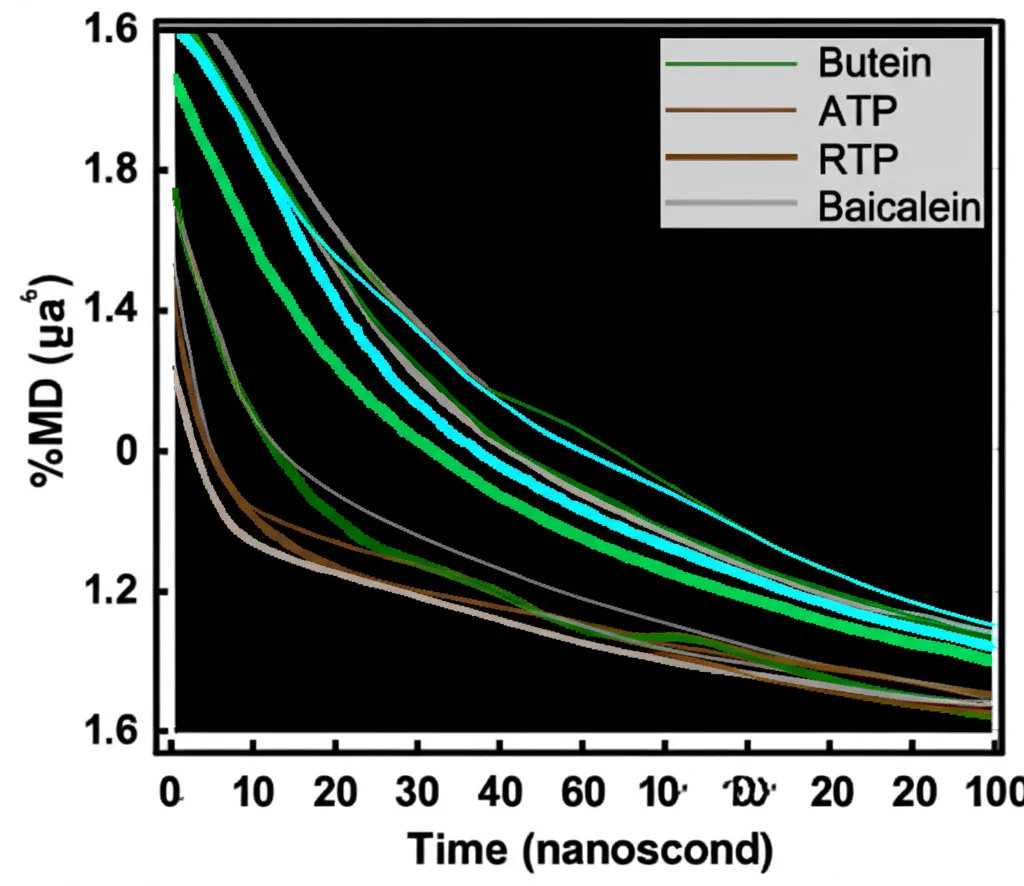
Le simulazioni MD hanno confermato la stabilità di questo legame. L’analisi RMSD (Root Mean Square Deviation), che misura quanto la molecola si sposta dalla sua posizione media, ha mostrato che la Buteina rimaneva molto più stabile nel sito di legame rispetto ad altri composti testati (come l’ATP, il substrato naturale dell’enzima, e la baicaleina, un altro flavonoide studiato). Anche l’enzima stesso (RdRp) mostrava una stabilità conformazionale comparabile a quando legato a inibitori noti, suggerendo un’interazione favorevole.
Energia di Legame e Residui Chiave
Abbiamo anche calcolato l’energia libera di legame con il metodo MMGBSA (Molecular Mechanics Generalized Born Surface Area). I risultati? La Buteina si lega all’RdRp con un’affinità significativamente maggiore rispetto all’ATP (il “cibo” naturale dell’enzima) e alla baicaleina. Sebbene l’affinità fosse inferiore a quella del Remdesivir trifosfato (la forma attiva del farmaco), è comunque un risultato notevole per una molecola naturale scoperta tramite screening.
L’analisi per residuo ha mostrato che i contributi energetici più forti al legame della Buteina provenivano proprio dalle interazioni con i nucleotidi dell’RNA primer (A19, U20, U18) e template (A13, A14, U12), confermando l’importanza di questo meccanismo d’azione unico. Anche alcuni residui dell’enzima nel “palm subdomain” (come Thr591, Ile589) contribuivano significativamente, principalmente tramite forze di van der Waals.
Cosa Significa Tutto Questo? Prospettive Future
Questo studio, seppur computazionale, ci apre una finestra entusiasmante. Abbiamo identificato un flavonoide naturale, la Buteina, derivato dalle piante di Erythrina, come un potenziale inibitore dell’RdRp di SARS-CoV-2. La cosa più interessante è il suo potenziale meccanismo d’azione, che coinvolge l’interazione diretta con l’RNA virale all’interno del sito attivo dell’enzima, e il suo promettente profilo di sicurezza previsto.
Certo, la strada è ancora lunga. Questi risultati in silico sono una base solida, ma ora servono urgentemente studi sperimentali in vitro per confermare l’attività inibitoria della Buteina sull’RdRp e sulla replicazione del virus in cellule. Se i risultati saranno positivi, si potrà poi passare a studi preclinici più approfonditi per valutarne la farmacocinetica reale, la farmacodinamica e la sicurezza, con la speranza di poter sviluppare un nuovo agente antivirale di origine naturale.
La natura continua a sorprenderci con il suo incredibile arsenale chimico. Chissà che la chiave per combattere future pandemie non si nasconda proprio in una pianta come l’Erythrina. Noi continuiamo a cercare!
Fonte: Springer