IBD: Sveliamo i Segreti della Natura con il Computer per Nuove Cure Multi-Bersaglio!
Ciao a tutti! Oggi voglio portarvi in un viaggio affascinante nel mondo della ricerca, un posto dove la tecnologia più avanzata incontra la saggezza antica della natura per combattere malattie complesse come le Malattie Infiammatorie Croniche Intestinali, o IBD (dall’inglese Inflammatory Bowel Disease). Parliamo di condizioni come la colite ulcerosa (UC) e il morbo di Crohn (CD), disturbi cronici che colpiscono l’intestino e che, purtroppo, stanno diventando sempre più comuni in tutto il mondo.
Capire l’IBD: Un Nemico Complesso
Le cause esatte dell’IBD sono ancora un po’ un mistero, ma sappiamo che c’è un mix intricato di fattori genetici, ambientali e immunologici. I sintomi? Spesso debilitanti: dolore addominale, diarrea, perdita di peso… un vero calvario per chi ne soffre. Alla base di tutto, troviamo spesso uno squilibrio del microbiota intestinale (la famosa disbiosi), un’infiammazione persistente e un danneggiamento della barriera epiteliale, quel rivestimento protettivo fondamentale del nostro intestino.
Immaginate questa barriera come un muro di mattoni ben sigillato. Nell’IBD, questo muro si indebolisce, perde le sue “giunzioni strette” e diventa permeabile. Questo permette ai batteri e ad altre sostanze normalmente confinate nell’intestino di entrare in contatto eccessivo con le cellule del nostro sistema immunitario. Il risultato? Una reazione immunitaria esagerata, con il rilascio di un fiume di molecole infiammatorie (citochine come IL-12, IL-23, IL-6, IL-17, IFN-γ). Questa infiammazione, a sua volta, danneggia ulteriormente la barriera, creando un circolo vizioso che rende la malattia cronica. Ecco perché, per trattare efficacemente l’IBD, non basta spegnere l’infiammazione: è cruciale anche riparare e rigenerare la barriera epiteliale.
L’Approccio Multi-Target: Più Frecce al Nostro Arco
Tradizionalmente, molti farmaci cercano di colpire un singolo “bersaglio” molecolare coinvolto nella malattia. Ma per una condizione complessa come l’IBD, con tanti meccanismi in gioco, questo approccio può avere dei limiti. Può portare a resistenza ai farmaci o richiedere dosi elevate che causano effetti collaterali.
Ecco dove entra in gioco la strategia multi-target. L’idea è semplice ma potente: sviluppare farmaci che possano agire contemporaneamente su più vie molecolari chiave. Nel nostro caso, vogliamo colpire sia l’infiammazione sia i meccanismi di riparazione della barriera. Pensiamo che questo possa portare a terapie più efficaci e magari con meno effetti collaterali.
I Nostri Bersagli: PDE4B, PHD1 e PHD2
Nel nostro studio, ci siamo concentrati su tre proteine “bersaglio” molto interessanti:
- PDE4B (Fosfodiesterasi 4B): Questo enzima gioca un ruolo chiave nell’infiammazione. In pratica, degrada una molecola chiamata cAMP, che normalmente aiuta a tenere a bada l’infiammazione. Inibendo PDE4B, aumentiamo i livelli di cAMP, il che porta a una riduzione della produzione di citochine infiammatorie e a un aumento di quelle anti-infiammatorie. Farmaci che inibiscono PDE4 (come Apremilast, approvato dalla FDA e testato anche per l’IBD) hanno già dimostrato di essere promettenti. Abbiamo scelto specificamente la forma B (PDE4B) perché sembra avere meno effetti collaterali rispetto ad altre forme come la PDE4D.
- PHD1 e PHD2 (Prolyl Hydroxylase Domain enzymes 1 e 2): Questi enzimi sono coinvolti nella regolazione della risposta cellulare all’ipossia (carenza di ossigeno) e, cosa cruciale per noi, nella sopravvivenza delle cellule epiteliali e nell’integrità della barriera. Normalmente, PHD1 e PHD2 “marcano” una proteina chiamata HIF-α (Hypoxia-Inducible Factor-alpha) per la distruzione. Se noi inibiamo PHD1 e PHD2, i livelli di HIF-α aumentano. E questo è un bene! HIF-α promuove l’espressione di fattori protettivi come le mucine e le β-defensine, che rafforzano la barriera epiteliale. Inoltre, inibire PHD1 sembra ridurre l’apoptosi (morte programmata) delle cellule epiteliali e l’infiammazione, mentre inibire PHD2 riduce anch’esso fattori infiammatori. Farmaci inibitori di PHD sono già in fase di studio per varie malattie, inclusa l’IBD (come AKB-4924).
Finora, questi bersagli erano stati studiati per lo più separatamente. La nostra scommessa è stata quella di cercare molecole capaci di colpirli tutti e tre.
La Natura Incontra il Computer: La Caccia ai Composti Bioattivi
Da secoli l’umanità si affida alle piante per curarsi. Oggi, con la crescente consapevolezza degli effetti collaterali dei farmaci di sintesi, c’è un ritorno di interesse verso i rimedi naturali. Il mercato globale della medicina erboristica è in piena espansione! Pensate alla curcumina (dalla curcuma) per le sue proprietà anti-infiammatorie, o al finocchio come aiuto digestivo.
Ma come trovare le molecole giuste in questo vasto arsenale naturale? Setacciare migliaia di composti in laboratorio richiederebbe tempi e costi proibitivi. Ed è qui che la tecnologia ci viene in aiuto con i metodi in silico, ovvero le simulazioni al computer. Questi strumenti ci permettono di accelerare drasticamente il processo di scoperta di nuovi farmaci.
Nel nostro lavoro, abbiamo iniziato con un database di ben 2299 composti bioattivi di origine vegetale. Usando potenti software e algoritmi, abbiamo fatto una sorta di “screening virtuale”.
Il Nostro Viaggio Computazionale: Dal Database ai Candidati Promettenti
Il nostro processo è stato meticoloso:
1. Selezione e Preparazione: Abbiamo ottenuto le strutture tridimensionali delle nostre proteine target (PDE4B, PHD1, PHD2) dal Protein Data Bank (PDB). Abbiamo preparato queste strutture e i 2299 composti vegetali usando software specifici (AutoDock Tools, OpenBabel).
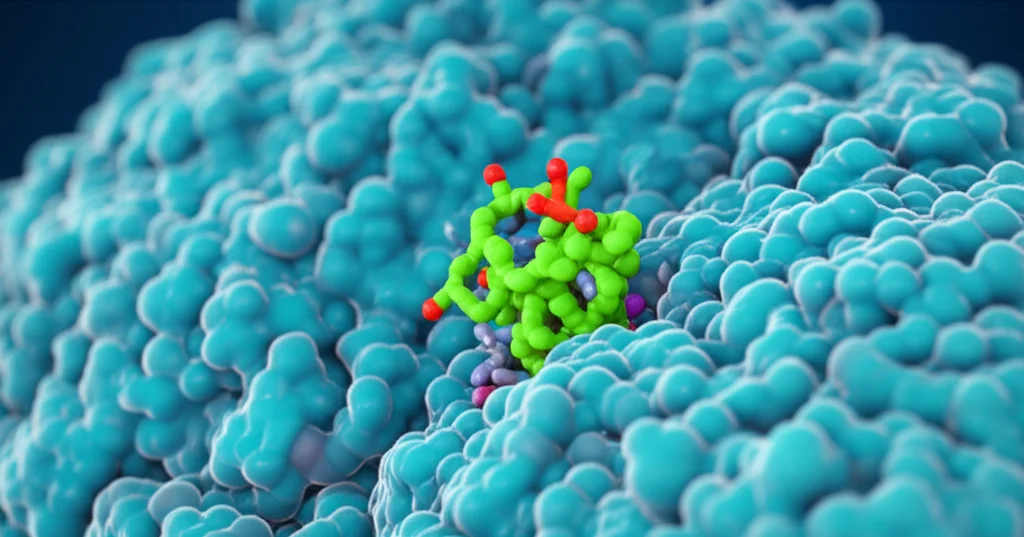
2. Docking Molecolare: Abbiamo simulato come ciascuno dei 2299 composti si “incastra” (lega) nel sito attivo di ciascuna delle tre proteine target, usando AutoDock Vina. Questo ci ha dato un punteggio di affinità (quanto bene si legano).
3. Filtro ADMET e Regola di Lipinski: Non basta che un composto si leghi bene; deve anche poter diventare un farmaco! Abbiamo quindi valutato le proprietà farmacocinetiche (Assorbimento, Distribuzione, Metabolismo, Escrezione – ADME) e la potenziale Tossicità (T) usando piattaforme online come OSIRIS Property Explorer e pkCSM. Abbiamo verificato se rispettavano la “Regola dei Cinque” di Lipinski, un insieme di criteri che predicono se un composto ha buone probabilità di essere assorbito per via orale.
4. Selezione dei Finalisti: Basandoci sui punteggi di affinità e sulle proprietà ADMET, abbiamo selezionato i cinque composti più promettenti: Cassiamin C, Ginkgetin, Hinokiflavone, Sciadopitysin e Sojagol. Questi composti hanno mostrato buona assorbibilità intestinale prevista, bassa probabilità di attraversare la barriera emato-encefalica (il che riduce il rischio di effetti sul sistema nervoso centrale) e nessun segnale preoccupante di tossicità o mutagenicità nei test predittivi.
5. Simulazioni di Dinamica Molecolare (MD): Per capire meglio come questi cinque composti interagiscono con le proteine target in condizioni più realistiche (simulando l’ambiente fisiologico), abbiamo eseguito simulazioni di dinamica molecolare per 150 nanosecondi usando GROMACS. Abbiamo analizzato parametri come RMSD (deviazione quadratica media, per la stabilità generale), RMSF (fluttuazione quadratica media, per la flessibilità locale), Raggio di Girazione (Rg, per la compattezza) e RDF (funzione di distribuzione radiale, per vedere come i composti si posizionano nel sito attivo). I risultati hanno confermato che i complessi proteina-ligando erano stabili durante la simulazione.
6. Calcolo dell’Energia Libera di Legame (MM-PBSA): Infine, abbiamo usato il metodo MM-PBSA per calcolare l’energia libera di legame (ΔGbind), una misura più accurata della forza dell’interazione tra il composto e la proteina. Abbiamo confrontato i nostri cinque candidati con i ligandi di riferimento (Apremilast per PDE4B e Vadadustat per PHD1/2).
I Risultati: Composti Naturali Battono i Riferimenti?
I risultati sono stati davvero incoraggianti!
- Energia di Legame: Quasi tutti i nostri composti hanno mostrato energie di legame (ΔGbind) migliori (più negative, quindi più forti) rispetto ai ligandi di riferimento per tutte e tre le proteine target. L’unica eccezione è stata Sojagol, che si è legato a PDE4B con un’energia leggermente meno favorevole di Apremilast, ma ha comunque superato Vadadustat per PHD1 e PHD2.
- Il Campione Multi-Target: Ginkgetin: Tra i cinque, Ginkgetin è emerso come un candidato particolarmente brillante. Ha mostrato l’energia di legame più forte in assoluto con PDE4B (-231.24 kJ/mol, contro -75.28 di Apremilast!) e ottime energie anche con PHD1 (-135.99 kJ/mol) e PHD2 (-132.04 kJ/mol), superando Vadadustat in entrambi i casi (-84.90 e -63.83 kJ/mol rispettivamente).
- Altri Forti Contendenti: Anche Hinokiflavone e Sciadopitysin hanno mostrato energie di legame molto forti con tutti e tre i target, spesso superando ampiamente i riferimenti. Cassiamin C ha anch’esso performato bene, superando i riferimenti su tutti i fronti.
- Interazioni Chiave: Analizzando le interazioni molecolari (legami idrogeno, interazioni idrofobiche, ponti salini, stacking π-π), abbiamo visto che questi composti formano legami robusti e specifici all’interno dei siti attivi delle proteine, spiegando la loro forte affinità.
Questi composti naturali, già noti in letteratura per altre proprietà benefiche (ad esempio, Ginkgetin è noto per le sue attività anti-infiammatorie, anti-cancro e neuroprotettive; Hinokiflavone per quelle anti-proliferative), sembrano avere le carte in regola per agire sui meccanismi chiave dell’IBD.
Guardando al Futuro: Dalla Simulazione alla Terapia
Certo, questi sono risultati ottenuti al computer. Il prossimo passo fondamentale è la validazione sperimentale. Bisognerà testare questi composti in vitro (su cellule in laboratorio, ad esempio cellule immunitarie attivate) e poi in vivo (su modelli animali di IBD, come il modello di colite indotta da DSS nei topi). Se questi test confermeranno l’efficacia e la sicurezza, si potrà pensare a studi clinici sull’uomo.
Siamo ottimisti. Crediamo che questo approccio multi-target, che sfrutta la potenza dei composti naturali identificati grazie a sofisticate strategie computazionali, possa davvero aprire la strada a nuove e più efficaci terapie per l’IBD. È un esempio perfetto di come la tecnologia moderna possa aiutarci a riscoprire e valorizzare i tesori nascosti nella farmacia della natura. La strada è ancora lunga, ma i primi passi sono molto promettenti!
Fonte: Springer







