Editing Genetico di Precisione: La Doppia Inibizione che Rivoluziona il Prime Editing!
Amici scienziati e appassionati di genetica, preparatevi perché oggi vi racconto una storia che ha del rivoluzionario! Parliamo di prime editing, una specie di “correttore di bozze” super sofisticato per il nostro DNA. Immaginate di poter entrare nel codice genetico e correggere piccoli errori, inserire pezzettini mancanti o togliere quelli di troppo con una precisione da chirurgo. Fantastico, vero? Il prime editing ci promette proprio questo, superando in precisione molti altri metodi di editing genetico, come la riparazione diretta dall’omologia (HDR).
Il “Ma” della Situazione: Efficienza vs. Precisione
Però, c’è un “ma”, come in tutte le belle storie. Sebbene il prime editing sia già di per sé più preciso di altri sistemi, a volte può comunque generare qualche “effetto collaterale”, come piccole inserzioni o delezioni non volute (le famigerate indels) o modifiche imprecise. Questo problema si accentua soprattutto quando cerchiamo di spingere sull’acceleratore dell’efficienza. Sistemi come il PE3 e il PE5, che usano un secondo “taglio” guida (gRNA nickante), o i sistemi con doppio pegRNA (come TwinPE, HOPE, Bi-PE), e quelli che usano una nucleasi completamente attiva (come il PEn), sono sì più potenti nel modificare il DNA, ma rischiano di compromettere la pulizia del lavoro. È un po’ come chiedere a un pittore di fare un ritratto dettagliatissimo in metà tempo: magari finisce prima, ma qualche sbavatura potrebbe scappare.
La Genialata: Bloccare i Riparatori Pasticcioni
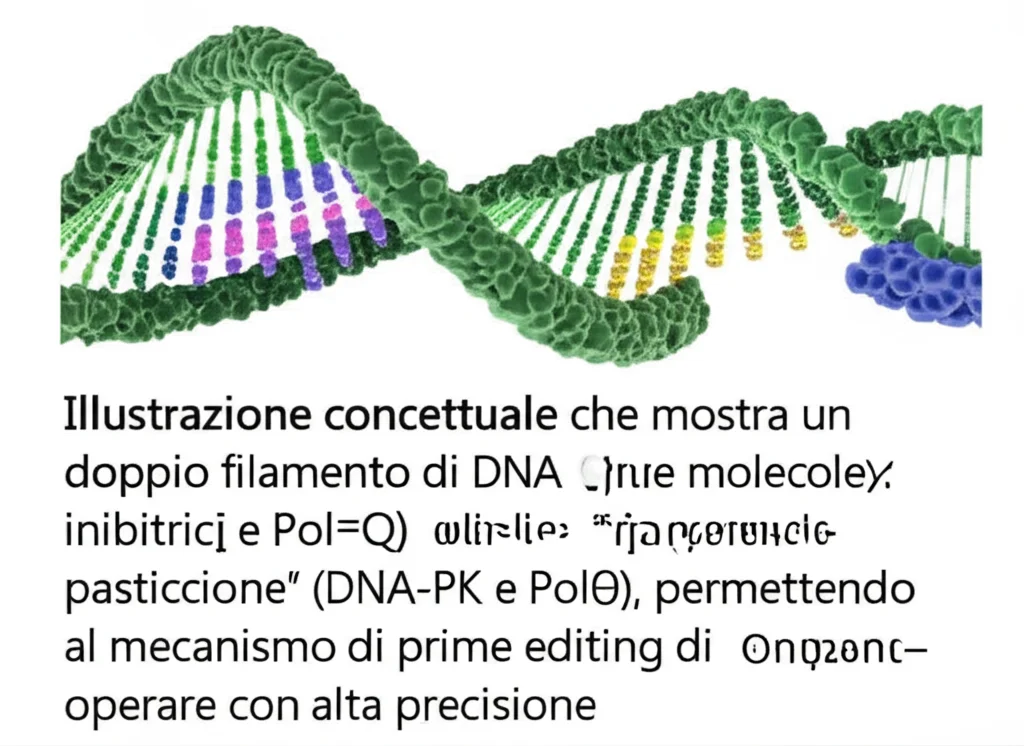
E qui, signore e signori, entra in gioco la genialata che vi voglio raccontare. Un gruppo di scienziati ha pensato: “E se potessimo ‘istruire’ la cellula su come riparare il DNA nel modo giusto, evitando che faccia pasticci?” Detto, fatto! Hanno scoperto che inibendo farmacologicamente due proteine chiave coinvolte nei percorsi di riparazione del DNA più “inclini all’errore”, chiamate DNA-PK e Polϴ (Pol Theta), la precisione di un’ampia gamma di sistemi di prime editing fa un balzo da gigante!
Pensate a DNA-PK e Polϴ come a due operai un po’ frettolosi che, quando c’è un danno al DNA (come i tagli indotti dal prime editing), intervengono per ripararlo, ma a volte lo fanno in modo un po’ approssimativo, causando appunto le indels. Bloccando temporaneamente questi due “operai pasticcioni” con delle piccole molecole (inibitori), si dà più spazio e tempo ai meccanismi di riparazione precisi, quelli che vogliamo noi!
PEn Sotto la Lente: Il Test Definitivo
Per mettere alla prova questa idea, i ricercatori hanno iniziato con il sistema PEn. Perché proprio PEn? Perché è uno dei più efficienti, ma anche uno di quelli che tende a generare più “sottoprodotti” indesiderati, a causa della sua nucleasi Cas9 completamente attiva che crea rotture del doppio filamento di DNA (DSB). Queste rotture attivano meccanismi di riparazione mutagenici come il NHEJ (Non-Homologous End Joining), mediato da DNA-PK, e l’alt-EJ (Alternative End-Joining), in cui Polϴ gioca un ruolo chiave.
Hanno quindi trattato cellule HEK293T con un inibitore specifico per DNA-PK (chiamato AZD7648, o DNA-PKi) e due diversi inibitori per Polϴ (PolQi1, che blocca il dominio polimerasico, e PolQi2, che blocca il dominio elicasico). L’obiettivo era installare una mutazione puntiforme nel gene KCNA1.
I risultati? Clamorosi!
- Il solo PEn era efficiente ma impreciso.
- PEn + DNA-PKi (1iPEn) migliorava la precisione ma non eliminava del tutto le indels, suggerendo che l’alt-EJ (mediato da Polϴ) prendeva il sopravvento.
- Ma la combinazione di DNA-PKi + PolQi1 (chiamata “2iPEn“) o DNA-PKi + PolQi1 + PolQi2 (“2+iPEn“) ha drasticamente ridotto le indels e migliorato la precisione a livelli stellari, mantenendo un’efficienza oltre 5 volte superiore rispetto al sistema PE5 (un altro sistema di prime editing)!
In pratica, con questa “doppia inibizione”, si è ottenuta una purezza di editing quasi completa. Le analisi hanno mostrato che l’inibizione di DNA-PK riduceva l’inserzione errata del “flap” di omologia dal pegRNA, mentre l’inibizione di Polϴ diminuiva le piccole delezioni attorno al sito di taglio. E la cosa bella è che questi trattamenti non sembravano influenzare la proliferazione cellulare.
Un Successo su Tutta la Linea: Da PEn agli Altri Sistemi
Ma la cosa non si ferma a PEn! I ricercatori hanno testato questa strategia “2+iPEn” su un pannello di bersagli genetici e tipi di modifiche (mutazioni puntiformi, inserzioni, delezioni) sia in cellule HEK293T che HeLa. Ebbene, 2+iPEn ha costantemente ridotto le indels, portando la precisione a livelli paragonabili a quelli del sistema PE5 (noto per la sua alta precisione ma minore efficienza rispetto a PEn), pur mantenendo l’alta efficienza di PEn. Parliamo di un aumento di precisione rispetto a PEn “nudo e crudo” di 4.3 volte in cellule HEK293T e addirittura 9.8 volte in cellule HeLa!
Meno Errori, Anche Fuori Bersaglio!
Un altro grattacapo dell’editing genetico, incluso PEn quando si usano pegRNA un po’ “promiscui”, sono le modifiche off-target, cioè quando l’editor taglia e cuce nel posto sbagliato del genoma. Sorprendentemente, la strategia 2+iPEn non solo ha migliorato la precisione sul bersaglio (on-target), ma ha anche ridotto drasticamente gli eventi off-target! Rispetto al PEn da solo, 2+iPEn ha diminuito l’editing off-target in media di ben 27.9 volte! Questo è un enorme passo avanti per la sicurezza di queste tecnologie.
PE3, PE5 e PE7: Tutti Beneficiano della Doppia Inibizione
Ok, PEn è sistemato. Ma che dire degli altri sistemi “potenziati” come PE3 e PE5, che usano un secondo nick per aumentare l’efficienza ma rischiano più indels? I ricercatori hanno testato l’approccio con inibitori (chiamandolo genericamente “2iPE” o “2+iPE” quando applicato a questi sistemi) anche su di loro.
Hanno provato a inserire 11 paia di basi nel gene PCSK9. Come previsto, PE2 e PE4 (versioni più “soft”) erano precisi ma poco efficienti. PE3 e PE5, invece, erano più efficienti ma meno precisi. L’aggiunta degli inibitori, specialmente nella combinazione 2+iPE, ha portato la precisione di PE3 e PE5 fino al 98% e 96% rispettivamente, mantenendo alta l’efficienza! Le delezioni indesiderate che spesso si vedevano con PE3 sparivano quasi completamente.
Questo effetto benefico è stato confermato su 12 diversi pegRNA, 4 loci genomici e vari tipi di modifiche. La purezza delle modifiche con PE3/PE5 è passata da circa il 60% a oltre il 93% in cellule HEK293T, e da circa il 50% a oltre il 90% in cellule HeLa, grazie alla strategia 2+iPE.
Anche il più recente sistema PE7, che co-esprime la proteina La per stabilizzare i pegRNA e aumentare l’efficienza, ha mostrato gli stessi benefici: quando usato con un ngRNA aggiuntivo per massimizzare l’efficienza, produceva più indels, ma queste venivano significativamente ridotte dalla co-inibizione di DNA-PK e Polϴ.
Applicabilità Vasta: Diverse Cellule, Stesso Successo
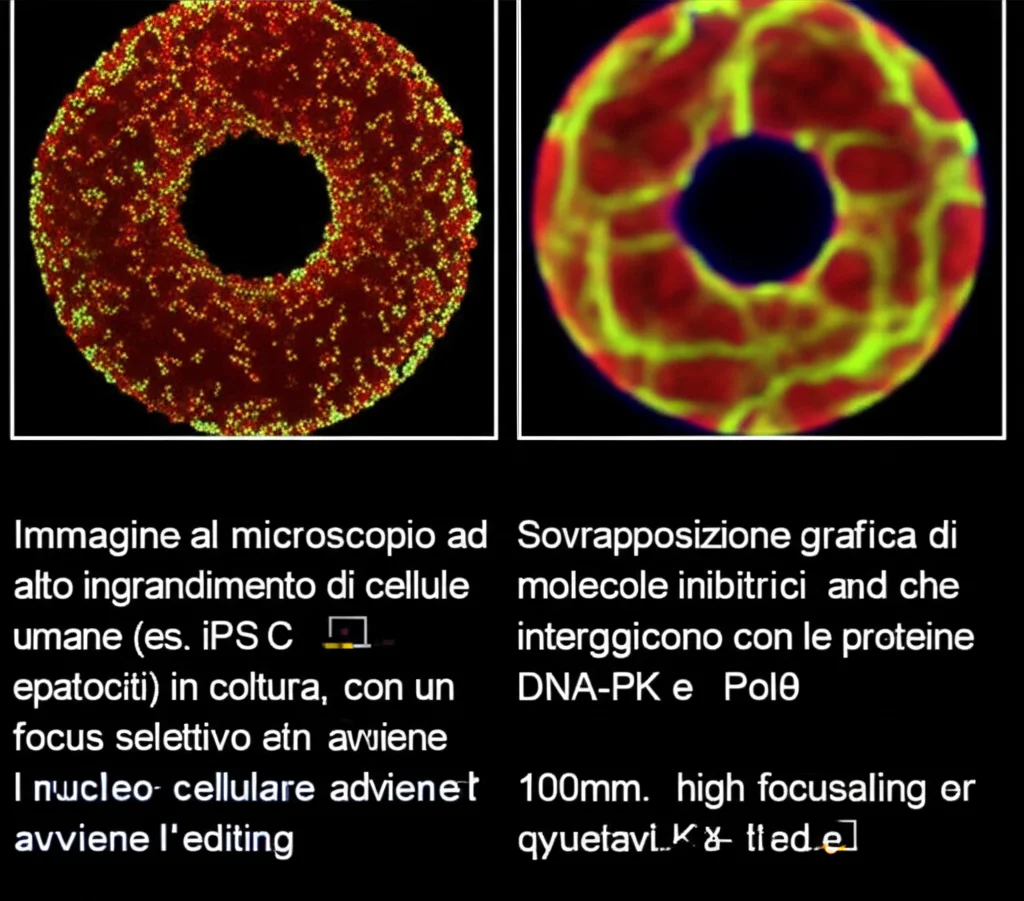
Per dimostrare che non si trattava di un colpo di fortuna limitato a un paio di linee cellulari, la strategia 2iPE e 2iPEn è stata testata su un’ampia gamma di tipi cellulari: linee cellulari come HepG2, Huh7, K562, ma anche cellule staminali pluripotenti indotte umane (iPSC) e persino epatociti umani primari (PHH). In generale, la strategia “2i” ha ridotto la formazione di indels in tutti i tipi cellulari testati (tranne nei PHH dove la frequenza basale di indels era già bassissima). Questi dati sottolineano l’ampia utilità di questo approccio.
Anche i Sistemi a Doppio pegRNA Ringraziano
E non è finita! I ricercatori si sono chiesti se questa doppia inibizione potesse migliorare anche la precisione dei sistemi di prime editing che usano due pegRNA contemporaneamente, come TwinPE, HOPE e Bi-PE. Questi sistemi sono fantastici per sostituire sequenze genomiche più lunghe o per introdurre delezioni ampie o mutazioni multiple, ma proprio per la loro natura “doppio taglio”, possono generare indels indesiderate.
Hanno provato con TwinPE a inserire siti di attacco per l’integrasi Bxb1 (attP da 50bp e attB da 38bp) in tre diversi loci genomici. TwinPE era efficiente (fino al 70%), ma introduceva fino al 9% di indels. L’aggiunta degli inibitori di DNA-PK e Polϴ (nella forma 2+i-TwinPE) ha ridotto le indels a meno dell’1%, mantenendo tassi simili di sostituzione precisa!
Risultati simili sono stati ottenuti con HOPE e Bi-PE: la co-inibizione ha ridotto sostanzialmente le indels mantenendo l’efficienza dell’editing preciso.
Infine, hanno testato anche una versione di TwinPE basata su nucleasi (TwinPEn). Come previsto, TwinPEn generava molte più indels rispetto alla versione basata su nickasi. Tuttavia, anche qui, l’aggiunta degli inibitori ha ridotto significativamente queste indels e aumentato i risultati di editing preciso.
Cosa Significa Tutto Questo?
In sostanza, abbiamo una nuova freccia al nostro arco per rendere il prime editing ancora più affidabile e preciso. Questa strategia, che chiamo affettuosamente “2iPE”, usa un cocktail di piccole molecole per “calmare” i meccanismi di riparazione del DNA più inclini all’errore (NHEJ e alt-EJ), permettendo al prime editing di fare il suo lavoro in modo pulito.
È interessante notare che, a differenza di quanto osservato con la riparazione HDR (dove gli stessi inibitori possono aumentare l’efficienza dell’integrazione del DNA), nel prime editing l’effetto principale è sulla riduzione dei sottoprodotti, senza generalmente aumentare l’efficienza della modifica precisa in sé. Questo suggerisce che i meccanismi alla base del prime editing e dell’HDR, pur essendo entrambi dipendenti da una riparazione basata sull’omologia (specialmente per PEn), hanno delle differenze cruciali, forse legate alle caratteristiche dei “template” di riparazione o alla dipendenza dal ciclo cellulare (l’HDR è principalmente ristretto alle fasi G2/S, mentre il prime editing sembra più indipendente).
La bellezza di questo approccio “2iPE” è la sua ampia applicabilità a diversi sistemi di prime editing (PEn, PE3, PE5, PE7, TwinPE, HOPE, Bi-PE) e la sua capacità di migliorare la purezza degli esiti di editing mantenendo alta l’efficienza. Inoltre, la riduzione degli off-target con PEn è un bonus non da poco!
Questo lavoro apre la strada a un utilizzo ancora più sicuro ed efficace del prime editing, sia per la ricerca di base, per modellare malattie, sia, in prospettiva, per future applicazioni terapeutiche. Insomma, un bel passo avanti per il mondo dell’ingegneria genetica di precisione!
Fonte: Springer







