TAC 3D Multifase: La Mia Scommessa per Smascherare il Cancro al Fegato!
Amici della scienza e della medicina, oggi voglio parlarvi di un argomento che mi sta particolarmente a cuore e che, ne sono convinto, rappresenta una svolta importante nella lotta contro un nemico subdolo: il cancro primario al fegato. Sapete, questa patologia è un vero problema di salute globale, con numeri da capogiro sia in termini di nuovi casi ogni anno (circa 905.677!) sia, purtroppo, di decessi (oltre 830.000), piazzandosi al terzo posto tra le cause di morte per cancro. Dati che fanno riflettere, vero?
Ecco perché una diagnosi precoce e accurata, insieme a una classificazione precisa dei suoi sottotipi, non è solo importante, è fondamentale. Pensateci: poter scegliere la terapia giusta al momento giusto può fare un’enorme differenza per l’esito del trattamento e per la qualità di vita dei pazienti. E qui entra in gioco una tecnologia che considero una vera alleata: la tomografia computerizzata con mezzo di contrasto (CECT). Questo strumento è noto per la sua alta sensibilità e specificità nel diagnosticare il cancro al fegato.
Il Nemico Insidioso: Il Cancro Primario al Fegato
Prima di addentrarci nei dettagli tecnici, capiamo meglio chi abbiamo di fronte. Il cancro primario al fegato non è un’entità unica. Esistono diversi sottotipi, i principali sono:
- Carcinoma Epatocellulare (HCC): il più comune, spesso legato a cirrosi o epatiti croniche.
- Colangiocarcinoma Intraepatico (ICC): che origina dai dotti biliari all’interno del fegato.
- Carcinoma Epatocolangiocellulare Combinato (cHCC-CCA): una forma più rara che presenta caratteristiche di entrambi.
Ognuno di questi “cattivi” richiede un approccio terapeutico su misura. Da qui, l’importanza di identificarli correttamente e il prima possibile. Una diagnosi precisa è la base per pianificare trattamenti efficaci e migliorare la prognosi.
La TAC con Mezzo di Contrasto (CECT): Un Alleato Prezioso
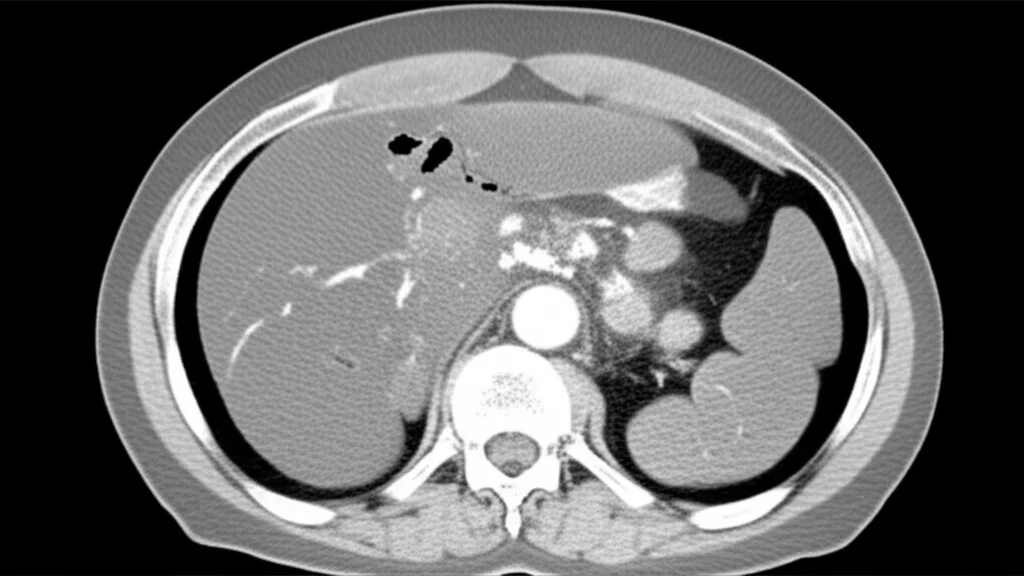
La CECT è una tecnica di imaging potentissima. Immaginate di poter “fotografare” il fegato in momenti diversi dopo l’iniezione di un mezzo di contrasto. Questo ci permette di osservare come il sangue fluisce attraverso l’organo e, soprattutto, attraverso eventuali lesioni. Tipicamente, una scansione CECT del fegato comprende quattro fasi chiave:
- Fase Pre-contrasto (o basale): un’immagine “nuda” del fegato, prima del contrasto.
- Fase Arteriosa: acquisita poco dopo l’iniezione, quando il contrasto è principalmente nelle arterie. È cruciale per i tumori ipervascolarizzati come l’HCC, che si “accendono” brillantemente.
- Fase Venosa (o portale): quando il contrasto si distribuisce nelle vene portali e nel parenchima epatico.
- Fase Tardiva (o di equilibrio): acquisita più tardi, utile per vedere il “washout” del contrasto da alcune lesioni o l’accumulo in altre.
Queste fasi sono essenziali perché diversi tipi di tumore mostrano comportamenti distinti in ciascuna di esse, fornendo indizi diagnostici preziosissimi. La CECT ci aiuta a valutare la vascolarizzazione del tumore, un fattore importante per capirne l’aggressività e pianificare i trattamenti.
Il Problema dei Dati: Una Sfida per la Ricerca
Con i progressi dell’intelligenza artificiale (IA) e del deep learning, abbiamo fatto passi da gigante nella classificazione delle immagini, nel rilevamento di oggetti e nella segmentazione. E, naturalmente, questi modelli avanzati sono stati applicati con successo anche al cancro al fegato. Gli studi dimostrano che utilizzare immagini CECT multifase migliora l’identificazione dei sottotipi di cancro primario al fegato. Il che sottolinea quanto siano importanti dati CECT completi per costruire modelli diagnostici di alta qualità.
Qui, però, casca l’asino. I dataset pubblici di scansioni CECT per il cancro al fegato sono limitati. Spesso si concentrano su un singolo tipo di cancro o, peggio ancora, non includono tutte le fasi della scansione. Pensate al dataset LiTS (Liver Tumor Segmentation Benchmark): include solo alcuni sottotipi e non offre una copertura completa delle fasi. Altri dataset, come SLIVER07 o 3D-IRCADb e CHAOS, hanno troppo pochi campioni per allenare modelli di classificazione o segmentazione robusti. Queste limitazioni, diciamocelo chiaramente, frenano i progressi.

È un po’ come voler insegnare a un bambino a riconoscere tutti gli animali avendogli mostrato solo foto di cani e gatti, e magari solo di profilo! Come può imparare a distinguere una tigre da un leone, o a riconoscerli da diverse angolazioni?
La Nostra Risposta: Un Dataset 3D CECT Rivoluzionario
Ed è qui che entra in gioco il nostro lavoro, la mia scommessa. Abbiamo ipotizzato che l’uso di immagini CECT 3D con fasi di scansione complete potesse davvero aiutare a sviluppare e validare modelli diagnostici e di segmentazione per il cancro primario al fegato. Così, ci siamo rimboccati le maniche e abbiamo creato un nuovo dataset CECT con aree epatiche e lesionali annotate.
Il nostro dataset include 278 casi di cancro al fegato, comprendenti carcinoma epatocellulare (HCC), colangiocarcinoma intraepatico (ICC) e carcinoma epatocolangiocellulare combinato (cHCC-CCA). Non solo: per aiutare i ricercatori a distinguere i tessuti tumorali da quelli normali, abbiamo incluso anche scansioni CECT di 83 soggetti senza cancro al fegato come gruppo di controllo. In totale, parliamo di oltre 50.000 “fette” (slice) di immagini con lesioni cancerose annotate. Un vero e proprio tesoro!
Dentro il Dataset: Cosa Contiene di Speciale?
Qualcuno potrebbe pensare che 278 pazienti con cancro al fegato siano pochi per addestrare modelli complessi. Ma la forza del nostro dataset risiede nella sua completezza e nella cura dei dettagli. Ogni paziente ha immagini CECT 3D complete che coprono tutte e quattro le fasi di cui parlavamo prima. E le oltre 50.560 slice annotate con lesioni sono state etichettate meticolosamente da medici esperti. Questo lo rende una risorsa sostanziale e di grande valore.
Le potenziali applicazioni cliniche sono enormi: dalla diagnosi precoce, identificando sottili cambiamenti nella struttura del fegato attraverso le diverse fasi CECT, alla classificazione dei sottotipi di cancro (HCC, ICC, cHCC-CCA), che potrebbe portare a piani di trattamento più accurati e personalizzati. Utilizzando questo dataset, i ricercatori possono contribuire a migliorare i modelli predittivi.
Certo, siamo consapevoli che si tratta di un dataset monocentrico, il che potrebbe richiedere dati aggiuntivi per lo sviluppo e la validazione di alcuni modelli. Tuttavia, crediamo fermamente che la sua diffusione pubblica sia preziosa. Può essere utilizzato come parte di dataset più ampi provenienti da altre fonti o come set di validazione esterno per altri ricercatori.
Come Abbiamo Costruito Questa Risorsa?
Il processo è stato lungo e meticoloso. Abbiamo raccolto retrospettivamente ed elaborato scansioni multifase. Questo ha comportato:
- Selezione dei campioni: Abbiamo incluso pazienti con diagnosi primaria di HCC, ICC o cHCC-CCA, maggiori di 18 anni, con conferma istologica o citologica. Abbiamo escluso tumori non primari, pazienti già trattati, o con altre patologie epatiche o sistemiche gravi che potessero confondere il quadro.
- Estrazione dei dati CECT: I pazienti, provenienti dal Dipartimento di Radiologia dell’Ospedale Popolare del Distretto di Chongqing Yubei tra gennaio 2015 e dicembre 2022, avevano subito una CECT multifase completa entro due settimane prima dell’intervento chirurgico. Abbiamo utilizzato scanner TC multi-detettore Canon e Philips, con protocolli specifici per le fasi arteriosa, venosa e tardiva.
- Conversione da DICOM a NIFTI: Le immagini, originariamente in formato DICOM per singola slice, sono state unite per creare immagini CECT 3D e salvate in formato NIFTI utilizzando il pacchetto SimpleITK.
- Regolazione di finestra e livello: Per migliorare la chiarezza dell’anatomia epatica e delle aree lesionali, abbiamo regolato la finestra di visualizzazione (livello 0, larghezza 400), “clippando” l’intensità dei pixel nell’intervallo (-200, 200) con NumPy.
- Selezione delle slice epatiche: I medici hanno identificato e registrato gli indici di tutte le slice contenenti la regione epatica, scartando le altre.
- Annotazione: Qui viene il bello! Per la regione epatica, abbiamo usato un modello pre-addestrato (LiverMask) come base, poi affinato dai medici con ITK-SNAP. Per le lesioni, due medici hanno eseguito annotazioni indipendenti su ciascuna immagine CECT 3D, esaminando simultaneamente tutte le fasi e considerando le caratteristiche di enhancement, morfologiche e interne (necrosi, emorragia, calcificazioni). Ogni divergenza è stata discussa e risolta congiuntamente fino al pieno accordo. Immaginate il lavoro certosino!
- De-identificazione: Per proteggere la privacy, abbiamo rimosso tutte le informazioni potenzialmente identificabili (PHI) dagli header dei file NIFTI, in conformità con l’HIPAA.
- Validazione: Per garantire qualità e coerenza.

La figura 1 del paper originale (che qui non posso mostrarvi, ma immaginate un diagramma di flusso) riassume splendidamente tutto questo processo, dalla selezione del campione al dataset finale etichettato.
Garanzia di Qualità: Validazione e De-identificazione
La validazione è stata un passaggio cruciale. Abbiamo verificato l’integrità dei dati (presenza di tutte e quattro le fasi, nessuna corruzione), la coerenza nella conversione da DICOM a NIFTI (mantenimento della risoluzione e delle proprietà spaziali), e la corrispondenza del numero di slice. Per le annotazioni, abbiamo calcolato il coefficiente di Dice per l’accordo inter-osservatore tra i due medici: la media è stata di 0.7586 (mediana 0.7474), un buon risultato! Le discrepanze sono state risolte per creare maschere di consenso. Abbiamo anche valutato l’accordo volumetrico sui tumori, con una differenza media di volume di 55.78 cm³.
La de-identificazione, come accennato, è stata rigorosa, controllando campioni casuali di file NIFTI per assicurarci che nessuna informazione protetta fosse rimasta.
Un Tesoro a Disposizione di Tutti: Accesso e Utilizzo
E la notizia più bella è che questo dataset è pubblicamente accessibile! Lo abbiamo depositato sulla piattaforma Science Databank con licenza Creative Commons Attribution 4.0 International (CC BY 4.0). Questo significa che chiunque può scaricarlo, usarlo e ridistribuirlo, anche per scopi commerciali, a patto di citare il lavoro originale. Un grande passo per la scienza aperta!
Il dataset contiene immagini CECT 3D in formato NIFTI per ogni fase (Basale, Arteriosa, Venosa, Tardiva), insieme ai file NIFTI delle maschere del fegato e delle lesioni. C’è anche un file CSV (“patient_data.csv”) con i metadati: ID paziente (anonimizzati da P0001 a P0361), età, sesso, tipo di cancro, fasi di imaging e percorsi ai file. Le dimensioni delle scansioni sono ni,j × 512 × 512, dove n (il numero di slice) varia. Per facilitare l’analisi, abbiamo fornito codice Python su GitHub (repository: ljwa2323/PLC_CECT) con un file demo (demo.ipynb) che mostra come caricare il dataset.
Parlando di numeri, per i pazienti con cancro al fegato (94 HCC, 99 ICC, 85 cHCC-CCA), l’età mediana era di 59 anni, con il 67.3% di maschi. In totale 1.112 immagini CECT 3D, con 50.560 slice con lesioni. Ogni paziente oncologico aveva una mediana di 124 slice, di cui 37 con lesioni. Il volume mediano delle lesioni era 75.37 cm³. Per il gruppo di controllo (83 individui), l’età mediana era 41 anni, con l’81.9% di femmine, e 332 immagini CECT 3D.
Prospettive Future: L’Impatto sulla Diagnosi e la Cura
Credo fermamente che questo dataset possa offrire un supporto prezioso per lo sviluppo e la validazione di modelli per la classificazione e la segmentazione del cancro primario al fegato. L’obiettivo finale? Migliorare la diagnosi precoce, personalizzare i trattamenti e, in definitiva, dare nuove speranze ai pazienti.
Questo studio ha ricevuto l’approvazione del Comitato Etico dell’Ospedale Popolare del Distretto di Chongqing Yubei (Approvazione N. 2022SA11), che ha anche concesso l’esonero dal consenso informato data la natura retrospettiva dello studio.
Spero che questa “immersione” nel mondo dell’imaging del cancro al fegato vi abbia affascinato quanto affascina me. La strada è ancora lunga, ma ogni dataset di qualità, ogni nuova intuizione, ci avvicina un po’ di più a sconfiggere questo terribile avversario.

Fonte: Springer Nature