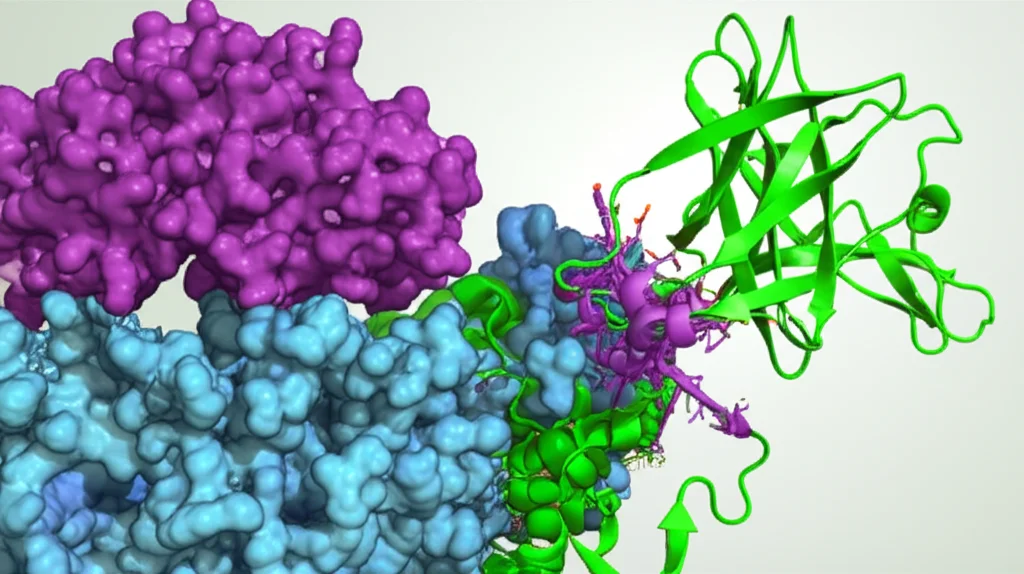
Chaperone Molecolari: Svelato l’Intrigante Segreto del Loro “Abbraccio” alle Proteine Appena Nate!
Amici scienziati (e non)! Preparatevi per un viaggio affascinante nel mondo microscopico delle nostre cellule, o meglio, in quello dei batteri, i nostri piccoli e instancabili coinquilini planetari. Oggi vi racconto una storia che mi ha letteralmente entusiasmato, una di quelle scoperte che ti fanno dire: “Ma allora è così che funziona!”. Parliamo di proteine, i mattoncini della vita, e di come riescono a prendere la loro forma corretta appena escono dalla “fabbrica” cellulare, il ribosoma. Un processo tutt’altro che scontato, ve lo assicuro!
Gli Angeli Custodi delle Proteine: Gli Chaperone
Immaginate una proteina come un lunghissimo filo di perle (gli amminoacidi) che, appena prodotto, deve ripiegarsi in una struttura tridimensionale precisissima per poter svolgere il suo lavoro. Se sbaglia forma, sono guai seri: può diventare inutile o, peggio, tossica. Qui entrano in gioco i nostri eroi: gli chaperone molecolari. Sono delle proteine specializzate che aiutano le altre proteine “neonate” (o nascenti, come diciamo noi scienziati) a non fare pasticci, a ripiegarsi correttamente e a non aggregarsi tra loro. Un po’ come delle tate molecolari super efficienti.
In particolare, nel batterio Escherichia coli, che è un po’ il nostro modello di studio preferito per queste cose, ci sono due chaperone principali che lavorano durante la sintesi proteica (un processo che chiamiamo co-traduzionale): il Trigger Factor (TF) e il sistema DnaK. Per anni, l’idea dominante è stata che questi chaperone agissero un po’ come degli “scudi”, legandosi principalmente alle catene proteiche ancora completamente spiegate, come se fossero dei fili aggrovigliati, per evitare che si appiccicassero a caso.
Una Nuova Luce sulla Danza Chaperone-Proteina
Ma la scienza è bella perché è sempre in evoluzione, e quello che abbiamo scoperto di recente getta una luce nuova e, per certi versi, più elegante su questo meccanismo. La domanda che ci siamo posti è stata: ma come fanno questi chaperone a essere così generali, interagendo con una marea di proteine diverse, e allo stesso tempo così specifici da aiutarle nel modo giusto? C’è sotto una regola universale?
Per capirci qualcosa, abbiamo messo in campo un arsenale di tecniche sofisticate. Abbiamo usato il Selective Ribosome Profiling (SeRP), una specie di “GPS molecolare” che ci dice quando e dove, lungo la catena nascente, gli chaperone si legano. Poi, siamo passati a esperimenti su singole molecole usando delle “pinzette ottiche” combinate con la fluorescenza, che ci hanno permesso di “vedere” e “manipolare” una proteina mentre si forma e interagisce con TF e DnaK. Infine, abbiamo usato la potenza dei computer e di un software pazzesco chiamato AlphaFold per fare delle predizioni.
E qui viene il bello! Contrariamente a quanto si pensava, abbiamo visto che TF e DnaK non preferiscono le catene proteiche completamente spiegate. Anzi! Si legano in modo molto più stabile a conformazioni parzialmente ripiegate della proteina nascente. È come se aspettassero che la proteina inizi a prendere una vaga forma, per poi intervenire.
I “Residui Insoddisfatti”: La Chiave di Volta
Abbiamo identificato un principio generale che sembra governare questo legame: i cosiddetti “residui insoddisfatti“. Cosa sono? Immaginate che la proteina, mentre emerge dal ribosoma, inizi a formare delle piccole strutture tridimensionali (domini). Alcuni amminoacidi (i residui) in queste strutture vorrebbero fare dei legami specifici con altri amminoacidi per completare il ripiegamento del dominio, ma questi “partner” non sono ancora stati sintetizzati, sono ancora “in coda” nel ribosoma. Questi residui, che hanno iniziato a formare una struttura ma non possono completarla, li abbiamo chiamati “insoddisfatti”.
Ecco, sembra proprio che TF e DnaK riconoscano e si leghino a questi agglomerati di residui insoddisfatti esposti sulle strutture parzialmente ripiegate. Quando poi la traduzione prosegue e i “partner” mancanti emergono, il dominio si completa, i residui diventano “soddisfatti” e lo chaperone si stacca. Geniale, no?
Questo principio ci ha permesso addirittura di predire, usando solo la sequenza della proteina, dove TF e DnaK si legheranno lungo quasi tutto il proteoma (l’insieme delle proteine) di E. coli, e le nostre predizioni combaciavano alla grande con i dati sperimentali! È una cosa notevole, considerando la complessità enorme di questi processi.
Un Lavoro di Squadra Ben Orchestrato
Ma non è finita qui. Abbiamo anche scoperto che TF e DnaK hanno ruoli un po’ diversi, ma sinergici. Il Trigger Factor (TF) sembra agire per primo, vicino all’uscita del tunnel ribosomiale. Si concentra soprattutto sul guidare il ripiegamento all’interno di un singolo dominio proteico. Una volta che il dominio è più o meno a posto, TF si stacca.
A questo punto, o anche in parallelo su proteine più lunghe, entra in gioco DnaK. Lui sembra più specializzato nell’evitare contatti prematuri e sbagliati tra domini diversi di una proteina. Immaginate una proteina fatta da più “palline” (i domini) collegate da fili. DnaK fa in modo che queste palline non si appiccichino nel modo sbagliato mentre si stanno ancora formando o mentre aspettano che altre palline vengano sintetizzate. DnaK, a differenza di TF, non ha bisogno di essere attaccato al ribosoma e può quindi seguire la proteina anche quando si allontana.
E se uno dei due manca? Il sistema è robusto! Se togliamo TF (con una mutazione genetica, ovviamente), DnaK e un altro chaperone, GroEL, si fanno avanti e si legano prima e a più proteine, cercando di compensare. È come se ci fosse un programma di “triage” adattivo. Curiosamente, se manca DnaK, TF non sembra cambiare molto il suo comportamento.
Chaperone al Lavoro Anche sulle Proteine di Membrana
Un’altra cosa interessante è che DnaK non si occupa solo delle proteine che stanno nel citoplasma. Abbiamo visto che dà una mano importante anche alle proteine che si inseriscono nella membrana cellulare (IMPs). Molte di queste hanno grosse porzioni che sporgono nel citoplasma, e DnaK si lega proprio quando queste porzioni emergono, aiutandole a ripiegarsi correttamente. Pensate alle chinasi istidiniche, dei recettori di membrana: DnaK si lega a quasi tutte mentre si formano!
GroEL, invece, sembra avere un ruolo diverso con le IMPs, forse più di “salvataggio” per quelle che non riescono a inserirsi bene nella membrana, per evitare che formino aggregati dannosi.
Una Semplice Regola per un Mondo Complesso
Quindi, cosa ci portiamo a casa da tutta questa storia? Che il legame degli chaperone co-traduzionali, almeno per TF e DnaK in E. coli, sembra seguire una regola di base sorprendentemente semplice: riconoscere e legarsi a questi “residui insoddisfatti” presenti su strutture proteiche parzialmente formate. Questo contrasta con l’idea che si leghino preferenzialmente a catene completamente spiegate.
Questa “semplicità di fondo” è affascinante, perché ci permette di fare predizioni accurate su un processo incredibilmente dinamico e complesso che coinvolge migliaia di proteine diverse. È come aver trovato una chiave di lettura universale per un linguaggio molecolare intricato.
Certo, il nostro modello è una semplificazione, non cattura ogni singola sfumatura. Ad esempio, non spiega perché TF leghi meno i domini più lontani dall’inizio della proteina in molecole molto grandi (probabilmente per ingombro sterico). Ma il suo scopo era proprio capire se delle regole generali, senza informazioni specifiche sulla proteina (se non la sua sequenza), potessero avere un valore predittivo su larga scala. E la risposta sembra essere un sonoro sì!
L’idea dei residui insoddisfatti potrebbe essere rilevante anche oltre il ripiegamento co-traduzionale. Chissà, forse ci aiuterà a capire meglio come funzionano altri chaperone, come le cellule gestiscono lo stress proteico, o persino i meccanismi alla base di alcune malattie legate al misfolding proteico. La ricerca, come sempre, continua, e ogni risposta apre la porta a nuove, eccitanti domande!
Fonte: Springer Nature