Carcinoma Anaplastico della Tiroide: A Caccia di Biomarcatori con la Bioinformatica!
Amici della scienza, oggi vi porto con me in un viaggio affascinante nel mondo della ricerca oncologica, un campo dove ogni scoperta può accendere una nuova speranza. Parleremo di una forma di cancro particolarmente ostica, il carcinoma anaplastico della tiroide (ATC), e di come la bioinformatica, una sorta di “investigazione digitale” nel nostro DNA, ci stia aiutando a capirlo meglio e, speriamo, a combatterlo più efficacemente.
L’ATC è un vero osso duro. Immaginatelo come il “cattivo” più temibile tra i tumori della tiroide: raro, sì, ma incredibilmente aggressivo e responsabile di una percentuale sproporzionata di decessi legati a questa ghiandola. Spesso, quando viene diagnosticato, è già in fase avanzata, rendendo le cure molto complesse. Ma perché è così aggressivo? La risposta, come spesso accade in biologia, è scritta nei geni.
L’Indagine Bioinformatica: Mettere Sotto la Lente i Geni
Per scovare i segreti molecolari dell’ATC, un team di ricercatori ha intrapreso un’analisi bioinformatica integrativa. Cosa significa? In pratica, hanno raccolto e analizzato dati provenienti da cinque diversi studi (chiamati “microarray datasets”) disponibili pubblicamente nel database GEO. Questi studi confrontavano i profili di espressione genica – cioè quali geni sono “accesi” o “spenti” e quanto – tra tessuti di ATC e tessuti tiroidei sani.
Pensate a GEO2R, uno degli strumenti usati, come a un sofisticatissimo comparatore che evidenzia le differenze. Per essere considerati “differenzialmente espressi” (DEGs), i geni dovevano mostrare una variazione significativa (un log2 fold change di almeno 1) e questa differenza doveva essere statisticamente robusta (p-value < 0.05). Per non perdersi in un mare di dati, hanno poi usato un diagramma di Venn (sì, proprio quelli che si studiano a scuola!) per trovare i geni che erano costantemente "sballati" in tutti e cinque i set di dati. Un po' come cercare l'identikit di un sospettato confrontando diverse testimonianze!
Una volta identificati questi geni “colpevoli”, bisognava capire cosa facessero. Qui entrano in gioco strumenti come DAVID e Metascape, che aiutano a interpretare le funzioni dei geni, raggruppandoli in base ai processi biologici, ai componenti cellulari e alle funzioni molecolari a cui partecipano (le famose Gene Ontology o GO) e ai percorsi metabolici o di segnalazione (KEGG pathways).
Infine, per vedere come questi geni interagiscono tra loro – perché nel nostro corpo nulla lavora mai da solo – hanno costruito una rete di interazione proteina-proteina (PPI) con STRING e analizzata con Cytoscape e il suo plugin MCODE. Immaginate una ragnatela complessa dove ogni filo è un’interazione: MCODE aiuta a trovare i “nodi” più importanti, i cosiddetti geni hub.
I Risultati: Cosa Abbiamo Scoperto?
L’analisi ha fatto emergere ben 7532 geni differenzialmente espressi (DEGs): 3509 erano “iperattivi” (upregolati) nelle cellule tumorali rispetto a quelle sane, mentre 4023 erano “silenziati” (downregolati). È un bel po’ di scompiglio genetico!
Concentrandosi sui geni che mostravano un comportamento anomalo in tutti e cinque i dataset, ne sono stati individuati 44: 10 costantemente upregolati e 34 costantemente downregolati.
E cosa fanno questi geni? Beh, quelli upregolati sono risultati essere coinvolti principalmente nella divisione cellulare e nel controllo del fuso mitotico. Non sorprende, vero? Il cancro è, per definizione, una crescita cellulare incontrollata. Questi geni sembrano spingere l’acceleratore sulla proliferazione.
I geni downregolati, invece, erano per lo più legati alla produzione di ormoni tiroidei e allo sviluppo della ghiandola tiroidea. Questo suggerisce che le cellule tumorali perdono le loro funzioni specializzate, un classico segno di dedifferenziazione tumorale, che spesso si associa a maggiore aggressività.
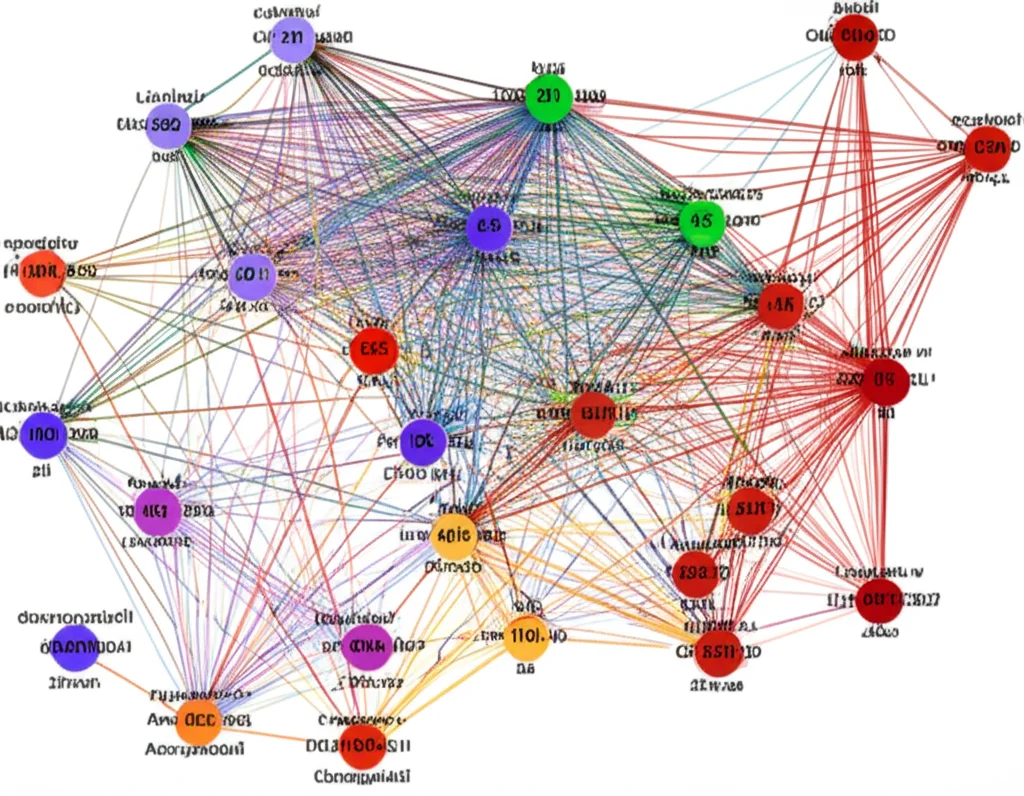
I “Sei Grandi”: I Geni Hub Sotto i Riflettori
Dall’analisi della rete di interazioni, sono emersi sei geni particolarmente “centrali”, dei veri e propri registi occulti di questa attività cellulare anomala. Eccoli qui, i nostri principali sospettati:
- TPX2: Un fattore cruciale per l’assemblaggio del fuso mitotico, quella struttura che permette alle cellule di dividersi correttamente. È già noto per essere coinvolto in vari tipi di cancro, e la sua iperattività nell’ATC potrebbe essere uno dei motori della proliferazione selvaggia.
- MAD2L1: Un guardiano del checkpoint del fuso mitotico. Se non funziona bene, la cellula può dividersi in modo errato, portando a instabilità genomica, un altro marchio di fabbrica del cancro.
- CDC20: Un componente regolatorio del complesso che promuove l’anafase (APC), essenziale per la progressione del ciclo cellulare. La sua sovraespressione è comune in molti tumori ed è già stato confermato essere alto nell’ATC. Un candidato forte per terapie mirate!
- CDKN3: Un inibitore delle chinasi ciclina-dipendenti, che dovrebbe frenare il ciclo cellulare. Paradossalmente, in alcuni tumori, inclusi quelli tiroidei aggressivi, la sua espressione anomala sembra contribuire alla deregolamentazione del ciclo.
- CENPF: Una proteina associata ai centromeri, importante per la corretta segregazione dei cromosomi durante la divisione. Alti livelli di CENPF sono stati correlati a una prognosi peggiore nell’ATC, suggerendo un ruolo nell’aggressività del tumore.
- NEK2: Una chinasi coinvolta nella regolazione mitotica e nella separazione dei centrosomi. La sua sovraespressione è documentata in molti cancri ed è associata a instabilità cromosomica e prognosi infausta. Potrebbe anche contribuire alla resistenza ai farmaci.
Questi sei geni sono tutti implicati in vie cruciali per la divisione cellulare e l’integrità cromosomica. Non è un caso che siano al centro della rete: la loro disregolazione sembra essere un evento chiave nella patogenesi dell’ATC.
Perché Questa Ricerca è Importante?
Questo studio, integrando dati da diverse fonti, ci offre una visione più robusta e affidabile del panorama molecolare dell’ATC. Identificare questi geni “chiave” e le vie biologiche alterate non è solo un esercizio accademico. Apre la strada a diverse possibilità concrete:
- Biomarcatori Diagnostici Precoci: Alcuni di questi geni potrebbero diventare dei segnali d’allarme per identificare l’ATC nelle sue fasi iniziali, quando è più trattabile.
- Nuovi Bersagli Terapeutici: Conoscere i meccanismi molecolari che guidano il tumore ci permette di pensare a farmaci “intelligenti” che vadano a colpire specificamente questi bersagli, magari con meno effetti collaterali rispetto alle terapie tradizionali.
- Medicina di Precisione: In futuro, potremmo analizzare il profilo genetico del tumore di ogni singolo paziente per scegliere la terapia più adatta.
Certo, ci sono delle limitazioni. Questa è un’analisi in silico, cioè fatta al computer. I risultati, per quanto promettenti, necessitano di conferme sperimentali in laboratorio, usando linee cellulari di ATC e modelli animali. Inoltre, i dati provenivano da campioni tumorali “bulk”, cioè un miscuglio di cellule diverse, il che potrebbe mascherare alcune specificità. Studi futuri con analisi a singola cellula potrebbero dare dettagli ancora più fini.
Verso il Futuro
In conclusione, questa dettagliata analisi bioinformatica ha acceso un faro su 7532 geni con espressione alterata nel carcinoma anaplastico della tiroide, e in particolare ha messo in evidenza sei “geni hub” – TPX2, MAD2L1, CDC20, CDKN3, CENPF e NEK2 – come potenziali protagonisti nella progressione di questa malattia aggressiva. Sono coinvolti in processi fondamentali come la mitosi e la regolazione del ciclo cellulare, il che li rende candidati promettenti sia per la diagnosi precoce sia come bersagli per nuove strategie terapeutiche.
Il bello della scienza è che ogni risposta apre nuove domande. Ora la palla passa ai laboratori, per verificare queste scoperte e, si spera, tradurle in strumenti concreti per migliorare la vita dei pazienti affetti da ATC. La strada è ancora lunga, ma ogni passo avanti, anche quello compiuto analizzando righe di codice e dati genetici, ci avvicina all’obiettivo.
Fonte: Springer