Biopsie ad Ago nel Glioblastoma: Una Finestra Rivoluzionaria sul Tumore Cerebrale
Ciao a tutti! Oggi voglio parlarvi di qualcosa che sta davvero cambiando il modo in cui guardiamo a uno dei tumori cerebrali più aggressivi: il glioblastoma (GBM). Come sapete, o come forse immaginate, combattere il GBM è una sfida enorme. Le terapie attuali, purtroppo, spesso non bastano e la prognosi rimane severa. Una delle difficoltà più grandi è capire cosa succede *dentro* il tumore mentre un paziente è sotto trattamento. Dopo l’intervento chirurgico iniziale, ottenere altri campioni di tessuto è complicato e rischioso, lasciandoci un po’ “al buio” sull’evoluzione della malattia e sulla risposta alle cure.
Ma se vi dicessi che uno strumento diagnostico di routine, la biopsia stereotassica ad ago, potrebbe diventare la nostra “lente d’ingrandimento” super potente per spiare il tumore da vicino? Esatto, quella procedura che i neurochirurghi usano da decenni per prelevare un piccolo campione di tessuto e fare diagnosi, oggi si rivela una miniera d’oro di informazioni!
Oltre la Diagnosi: Nascono le Biopsie “Investigative”
Qui entra in gioco un concetto affascinante: le biopsie investigative. L’idea è semplice ma rivoluzionaria: usare quelle stesse biopsie ad ago, non solo per confermare la diagnosi, ma per raccogliere una quantità incredibile di dati dettagliati sul tumore e sul suo microambiente (il famoso Tumor Microenvironment, o TME). Pensateci: potremmo monitorare come le cellule tumorali e quelle immunitarie reagiscono alle terapie, quasi in tempo reale!
Fino a poco tempo fa, si pensava che i campioncini prelevati con l’ago fossero troppo piccoli per analisi complesse. E invece, la ricerca ha fatto passi da gigante! Nel nostro campo, abbiamo dimostrato che è assolutamente fattibile ottenere dati profondi e multimodali da questi piccoli prelievi. E quando dico multimodali, intendo un vero e proprio check-up completo del tumore:
- Sequenziamento dell’RNA a singola cellula (scRNA-seq): Per vedere cosa fa ogni singola cellula nel campione.
- Trascrittomica spaziale: Per capire dove si trovano le diverse cellule e come interagiscono nello spazio.
- Metabolomica spaziale: Per mappare le molecole del metabolismo e capire l’attività “energetica” del tumore.
- Proteomica e Fosfoproteomica: Per analizzare le proteine e le loro modifiche, che ci dicono quali “interruttori” molecolari sono accesi o spenti.
- Analisi del clonotipo dei linfociti T: Per studiare le cellule immunitarie che combattono (o dovrebbero combattere) il tumore.
- Immunopeptidomica MHC di Classe I: Per scoprire quali “bandierine” (antigeni) il tumore espone sulla sua superficie, potenziali bersagli per il sistema immunitario.
Tutto questo da un prelievo standard! Incredibile, vero?
Affrontare le Sfide Tecniche: Ottimizzare per il “Piccolo”
Ovviamente, non è stato un percorso in discesa. Lavorare con quantità minime di tessuto (<50 mg, pensate!) richiede protocolli super ottimizzati. Prendiamo lo scRNA-seq: ottenere abbastanza cellule vitali da un core bioptico era considerato quasi impossibile. Gli studi precedenti riportavano poche centinaia di cellule per biopsia. Noi, però, abbiamo messo a punto delle tecniche, soprattutto di dissociazione manuale del tessuto, che ci hanno permesso di ottenere centinaia di migliaia di cellule vitali da un paio di core! Un salto quantico che apre le porte ad analisi molto più dettagliate.
Abbiamo anche confrontato diverse strategie: dissociazione manuale vs automatica (spoiler: vince il “fatto a mano”!), uso di enzimi vs solo metodi meccanici, analisi su cellule fresche vs nuclei congelati. Per esempio, abbiamo visto che analizzare cellule fresche (scRNA) ci dà un quadro più completo dell’espressione genica nelle cellule immunitarie mieloidi rispetto all’analisi di nuclei congelati (snRNA), che invece perdeva alcuni geni chiave. Ogni dettaglio è fondamentale per ottenere dati di alta qualità.

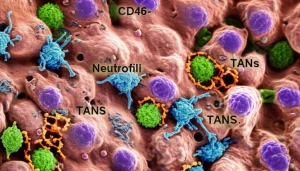
Un’altra sfida è l’eterogeneità del glioblastoma. Sappiamo che questo tumore non è un blocco unico, ma un mosaico di aree diverse (più cellulari, infiltrative, necrotiche…). Quindi, un singolo prelievo è rappresentativo? Per rispondere, abbiamo sviluppato un sistema ingegnoso: prelevando più core dalla stessa micro-regione (ruotando l’ago) e da regioni diverse, e registrando le coordinate 3D esatte di ogni prelievo, possiamo mappare tutto sulla risonanza magnetica del paziente. Poi, usando analisi avanzate (come la valutazione delle alterazioni del numero di copie del DNA – CNV – e l’analisi computerizzata delle immagini istologiche HeE), abbiamo creato un metodo per quantificare la variabilità *dentro* una stessa regione e *tra* regioni diverse. Questo ci aiuta a capire quanto è omogeneo il campione e a identificare eventuali “outlier” dovuti, ad esempio, a una maggiore necrosi.
Dalla Mappa alla Funzione: Integrare i Dati è la Chiave
La vera potenza di questo approccio sta nell’integrazione dei dati multimodali. Avere tante informazioni diverse dallo stesso campione ci permette di collegare i puntini e avere un quadro molto più completo. Per esempio:
- Abbiamo confrontato i dati di metabolomica spaziale con quelli di scRNA-seq, trovando una buona correlazione nei percorsi metabolici attivi nelle cellule immunitarie (come il metabolismo del glutammato o dell’acido linoleico). È come vedere la stessa cosa da due angolazioni diverse!
- Abbiamo usato l’immunopeptidomica per vedere quali antigeni erano presentati dal tumore e poi abbiamo cercato i geni corrispondenti nei dati scRNA. Risultato? L’espressione genica non è un buon predittore di quali peptidi finiscono davvero “in vetrina” sulla cellula. Questo sottolinea l’importanza di misurare direttamente i peptidi!
- Forse l’esempio più affascinante è la validazione delle interazioni cellula-cellula. Algoritmi come Cellchat, basati su scRNA-seq, predicono quali cellule potrebbero comunicare tra loro tramite coppie ligando-recettore (L-R). Noi abbiamo “filtrato” queste predizioni usando i dati di fosfoproteomica (che ci dicono quali recettori sono effettivamente attivi) e abbiamo identificato una coppia L-R chiave: Osteopontina (SPP1) e CD44. Poi, siamo andati a “vedere” questa interazione direttamente nel tessuto con tecniche di imaging spaziale (CyCIF), confermando che le cellule mieloidi esprimevano SPP1 e interagivano con cellule T e tumorali che esprimevano CD44, proprio come previsto! È la prova che integrare dati diversi ci porta a scoperte più solide.

Non Solo Dati: Creare Modelli Paziente-Specifici
E non è finita qui! Un altro aspetto incredibile è che questi piccoli campioni bioptici possono essere usati anche per creare modelli tumorali paziente-specifici, come i Patient-Derived Xenografts (PDX). Si tratta di impiantare le cellule tumorali del paziente in topi immunocompromessi per far crescere un “avatar” del tumore originale. Questo è fondamentale per testare nuovi farmaci in un contesto che mima la malattia del singolo paziente.
Abbiamo analizzato retrospettivamente i dati di un trial clinico e abbiamo scoperto che era possibile generare PDX da biopsie ad ago con un tasso di successo notevole (circa il 42%!). Sorprendentemente, il successo non dipendeva tanto dalla percentuale di tumore nel campione, quanto dal numero totale di cellule vitali che riuscivamo a isolare. Siamo riusciti a creare modelli anche da campioni con solo il 10% di cellule tumorali o con una certa quantità di necrosi. Questo apre la strada a una medicina davvero personalizzata, dove possiamo studiare il tumore specifico di un paziente e testare terapie su misura sul suo “avatar” tumorale, il tutto partendo da una semplice biopsia.

Verso il Futuro: Monitoraggio Longitudinale e Nuove Terapie
Qual è il messaggio finale? Le biopsie stereotassiche ad ago, considerate a lungo solo strumenti diagnostici, sono in realtà potentissime piattaforme per la ricerca e, potenzialmente, per la clinica. La capacità di generare dati multimodali così ricchi da campioni minimamente invasivi apre scenari entusiasmanti:
- Monitoraggio della risposta alla terapia: Immaginate di poter fare biopsie investigative a diversi timepoint durante il trattamento per vedere esattamente come il tumore e il sistema immunitario stanno reagendo. Questo potrebbe guidare decisioni terapeutiche personalizzate.
- Sviluppo di nuovi farmaci: Capire nel dettaglio le interazioni cellulari e i meccanismi di resistenza nel TME è cruciale per disegnare terapie più efficaci, magari combinando approcci diversi.
- Biomarcatori: Avere dati così dettagliati dal tessuto ci aiuterà a cercare corrispondenze nel sangue o nel liquido cerebrospinale, per sviluppare biomarcatori meno invasivi per monitorare la malattia.
- Oltre il cancro: Queste tecniche potrebbero essere utili anche per studiare altre malattie neurologiche, come quelle neuroinfiammatorie.
Questo lavoro è solo l’inizio. Abbiamo dimostrato che è fattibile, che i dati sono di alta qualità e rilevanti. Ora la sfida è applicare questa piattaforma su larga scala. Infatti, è già in corso un trial clinico (NCT03152318) che prevede campionamenti longitudinali in pazienti con GBM, proprio per sfruttare appieno il potenziale delle biopsie investigative.
Insomma, quel piccolo ago da biopsia si sta rivelando uno strumento incredibilmente potente. Non è solo un modo per diagnosticare, ma una vera e propria finestra spalancata sul complesso e affascinante mondo del microambiente tumorale del glioblastoma. E io sono convinto che da questa finestra arriveranno molte delle risposte che cerchiamo per combattere questa terribile malattia.
Fonte: Springer