Apprendimento Contrastivo e Mixture of Experts: L’AI che Decifra le Banche Dati Biologiche
Un Tuffo nel Complesso Mondo della Scienza Biomedica
Avete mai pensato a quanto sia incredibilmente vasto e complesso il mondo della ricerca scientifica, specialmente in campo biomedico? Ogni giorno vengono pubblicati migliaia di studi, creando un oceano di informazioni in cui è facile perdersi. Come possiamo navigare efficacemente in questa marea di dati? Qui entra in gioco l’intelligenza artificiale, e in particolare una tecnica chiamata embedding vettoriale. In parole povere, si tratta di trasformare documenti complessi, come articoli scientifici, in “punti” matematici (vettori) all’interno di uno spazio multidimensionale. Più due documenti sono simili nel contenuto, più i loro punti saranno vicini in questo spazio. Fantastico, no? Permette ricerche super veloci e analisi di similarità impensabili fino a poco tempo fa.
La Sfida: Quando l’AI Standard Non Basta
Ma c’è un “ma”. I modelli AI più diffusi, anche quelli potentissimi come i transformer (alla base di meraviglie come ChatGPT o BERT), a volte faticano un po’ quando si tratta di letteratura scientifica molto specifica. Immaginate di dover distinguere tra due studi che parlano di meccanismi molecolari leggermente diversi all’interno della stessa malattia: una sfumatura sottile per un umano esperto, ma una sfida notevole per un’AI generica. Questi modelli possono generare rappresentazioni vettoriali non ottimali, poco “precise”, rendendo difficile distinguere ricerche altamente correlate o appartenenti a nicchie specifiche. E con la crescente dipendenza da sistemi di ricerca potenziati dall’AI (come la Retrieval Augmented Generation – RAG), avere vettori davvero descrittivi e precisi è diventato cruciale.
La Nostra Ricetta Segreta: Apprendimento Contrastivo e “Esperti” AI
Allora, come abbiamo affrontato questa sfida? Abbiamo messo a punto un approccio che combina due potenti tecniche del mondo AI: l’Apprendimento Contrastivo (Contrastive Learning) e la Mixture of Experts (MoE).
L’Apprendimento Contrastivo è un po’ come insegnare a un bambino a riconoscere le differenze e le somiglianze. Abbiamo “allenato” i modelli AI (partendo da basi solide come BERT) mostrandogli coppie di abstract di articoli scientifici. Come metrica di similarità abbiamo usato le co-citazioni: se due articoli vengono citati insieme da un terzo articolo, è molto probabile che trattino argomenti simili o correlati. Quindi, abbiamo insegnato al modello: “questi due abstract (co-citati) sono simili, mentre questi altri due (presi a caso) probabilmente non lo sono”. Questo aiuta l’AI a cogliere le sfumature specifiche di diversi domini biomedici.
E qui arriva la parte ancora più intrigante: la Mixture of Experts (MoE). Immaginate di avere non un solo “cervello” AI, ma un team di specialisti! Abbiamo preso un modello pre-allenato (nello specifico, il performante ModernBERT) e, invece di avere una sola sezione che elabora le informazioni (il Multi-Layer Perceptron o MLP), ne abbiamo create diverse copie identiche, ognuna destinata a diventare un “esperto” in un dominio specifico. Nel nostro studio, abbiamo creato esperti per malattie cardiovascolari (CVD), broncopneumopatia cronica ostruttiva (COPD), malattie parassitarie, malattie autoimmuni e tumori della pelle.
La genialata? Abbiamo usato dei “token speciali” (come [CVD], [COPD]) all’inizio di ogni abstract per dire all’AI: “Ehi, questo testo appartiene a questo dominio, quindi mandalo al suo esperto dedicato!”. Questo sistema di “instradamento forzato”, basato su questi token speciali, fa sì che ogni esperto si specializzi nel suo campo e, cosa importantissima, mantiene la velocità di elaborazione identica a quella di un modello standard quando si processano dati dello stesso dominio! Niente rallentamenti, solo più intelligenza specifica.
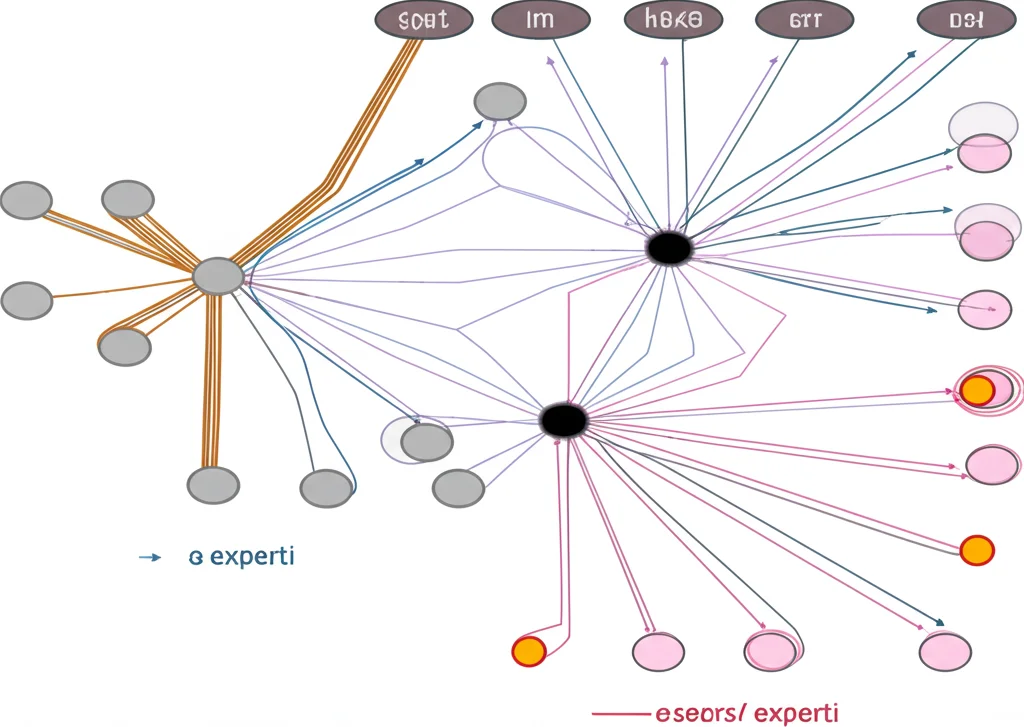
Alla Prova dei Fatti: L’Esperimento Biomedico
Per vedere se la nostra idea funzionava, abbiamo messo insieme un bel dataset. Abbiamo usato PubMed Central per raccogliere abstract di articoli scientifici pubblicati tra il 2010 e il 2022, appartenenti ai cinque domini biomedici scelti:
- Malattie Cardiovascolari (CVD)
- Broncopneumopatia Cronica Ostruttiva (COPD)
- Malattie Parassitarie
- Malattie Autoimmuni
- Tumori della Pelle
Abbiamo identificato le coppie co-citate (i nostri esempi “positivi”) e creato coppie “negative” mettendo insieme abstract non co-citati. Abbiamo quindi addestrato diversi modelli: il nostro modello MoE con 5 esperti (chiamato MoEall), un modello standard ma addestrato su tutti i domini (SEall), modelli standard addestrati su un solo dominio (es. SEcvd), e li abbiamo confrontati con modelli AI molto noti e performanti, tra cui varianti di BERT specializzate sulla scienza (SciBERT, BioBERT), modelli ottimizzati per la similarità di frasi (E5, MPNet), il popolare Llama-3.2 e persino un metodo classico come TF-IDF.
I Risultati? Decisamente Promettenti!
E i risultati? Beh, sono stati davvero incoraggianti! In media, su tutti i domini, il nostro approccio MoEall ha ottenuto le performance migliori nel classificare correttamente le coppie di abstract co-citate, seguito da vicino dal modello SEall. Abbiamo misurato la performance usando una metrica chiamata F1max, che ci dice quanto è bravo il modello a trovare un equilibrio tra precisione e “completezza” nel riconoscere le coppie giuste. Il nostro MoEall ha raggiunto un ottimo 0.8875 di F1max medio!
Un altro dato interessante è il “rapporto di similarità”: quanto più alta è la similarità media delle coppie co-citate rispetto a quelle non co-citate. Anche qui, i modelli MoE hanno mostrato una maggiore capacità di separare i due gruppi, rendendo le rappresentazioni vettoriali più “distinte”.
Come ci aspettavamo, i modelli addestrati su un singolo dominio (come SEcancer) sono stati i campioni assoluti *all’interno* del loro specifico campo di specializzazione (tranne per le malattie autoimmuni, dove MoEall ha avuto un leggerissimo vantaggio). Questo dimostra l’efficacia della specializzazione. Tuttavia, è notevole che anche i modelli addestrati su un solo dominio abbiano superato molti modelli pre-allenati generici quando valutati su *tutti* i domini, suggerendo che l’addestramento specifico su un dominio scientifico non “dimentica” completamente come gestire gli altri.
È interessante notare che nel dominio CVD, dove anche i modelli pre-allenati di base mostravano già una buona capacità di separazione, i nostri modelli super-specializzati non hanno superato alcuni campioni della similarità come MPNet ed E5. Questo suggerisce che il nostro approccio dà il meglio di sé quando la sfida discriminativa è più alta. Ad esempio, nel dominio COPD, dove i modelli base faticavano tantissimo (performance quasi casuali!), i nostri modelli hanno fatto un balzo di qualità enorme.

Perché Tutto Questo è Importante? Il Futuro è Adesso
Ma al di là dei numeri, cosa significa tutto questo? Significa che stiamo facendo passi da gigante verso la creazione di sistemi AI capaci di “comprendere” la letteratura scientifica a un livello molto più profondo e granulare. Immaginate motori di ricerca per database scientifici (come PubMed o Semantic Scholar) che non solo trovano articoli basati su parole chiave, ma capiscono veramente le connessioni concettuali tra di essi, anche quelle più sottili e specifiche di un dominio. Pensate a sistemi RAG che possono attingere a conoscenze biomediche incredibilmente precise per rispondere a domande complesse.
Il nostro approccio MoE, con il suo instradamento intelligente che non sacrifica la velocità, apre la porta a modelli “universali” ma specializzati: un unico modello AI capace di gestire con maestria dati provenienti da diversi campi della biologia o della medicina, attivando l’esperto giusto al momento giusto. Efficienza e precisione che vanno a braccetto.
Oltre l’Orizzonte: Cosa Ci Aspetta?
Le possibilità future sono elettrizzanti. Potremmo usare questi embedding vettoriali super precisi per migliorare il riconoscimento di entità specifiche (Named Entity Recognition – NER) nei testi scientifici. Oppure potremmo usarli per alimentare sistemi di Retrieval Augmented Generation (RAG) ancora più potenti. Un’altra frontiera affascinante è l’interpretabilità: studiare come questi “esperti” AI imparano e strutturano la conoscenza specifica del loro dominio potrebbe svelarci qualcosa di nuovo su come funziona l’apprendimento profondo stesso. Potremmo persino esplorare la fusione di questi esperti per creare modelli ancora più potenti ed efficienti.
In Conclusione: Un Passo Avanti per l’AI nella Scienza
Insomma, combinando l’apprendimento contrastivo basato su co-citazioni con l’architettura flessibile e potente della Mixture of Experts, abbiamo dimostrato che è possibile creare rappresentazioni vettoriali di documenti scientifici significativamente più precise e utili. È un passo avanti importante per rendere l’intelligenza artificiale uno strumento ancora più efficace al servizio della scoperta scientifica. Il viaggio è appena iniziato, ma la direzione è chiara: verso un’AI che non solo processa informazioni, ma le comprende davvero, aiutandoci a svelare i segreti nascosti nelle immense banche dati biologiche.
Fonte: Springer