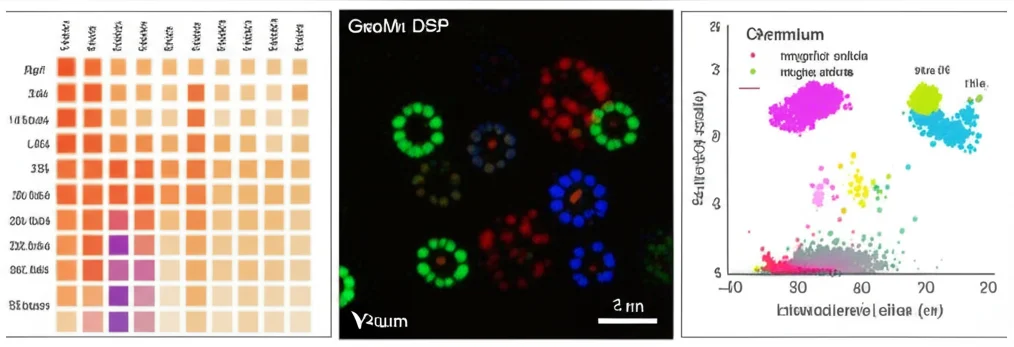
Tumori Sotto la Lente: La Mia Avventura con Visium, GeoMx e Chromium per Svelare i Segreti Nascosti
Ciao a tutti! Oggi voglio portarvi con me in un viaggio affascinante, nel cuore della ricerca oncologica. Avete presente quei vecchi campioni di tessuto tumorale, conservati con cura negli archivi ospedalieri? Beh, sono una miniera d’oro di informazioni, ma analizzarli a fondo, soprattutto per capire cosa succede a livello di geni attivi (il famoso trascrittoma), è sempre stata una bella sfida. Ma la scienza, si sa, non si ferma mai!
Recentemente, sono emerse tecnologie pazzesche, basate su sonde, che ci permettono di studiare il trascrittoma completo proprio da questi tessuti fissati in formalina e inclusi in paraffina (FFPE), quelli che, per intenderci, sono lo standard negli archivi. Sto parlando di nomi che forse vi suoneranno nuovi, ma che nel nostro campo stanno facendo faville: Visium CytAssist, Chromium Flex e GeoMx DSP. Grazie a loro, possiamo finalmente pensare di analizzare coorti enormi di pazienti, aprendo scenari incredibili per la ricerca.
Però, c’è un “ma”. Queste metodiche sono complesse, richiedono un sacco di lavoro e, diciamocelo, costano un occhio della testa. Quindi, prima di partire lancia in resta su un progetto, è fondamentale scegliere lo strumento giusto per l’obiettivo che ci siamo prefissati. Ed è proprio qui che entra in gioco il nostro lavoro: abbiamo deciso di mettere alla prova queste tre “fuoriserie” della genomica, confrontandole fianco a fianco.
La Nostra Missione: Mettere a Confronto i Titani
Ci siamo rimboccati le maniche e abbiamo analizzato campioni di tumore FFPE di tre tipi diversi: carcinoma mammario, carcinoma polmonare non a piccole cellule (NSCLC) e linfoma diffuso a grandi cellule B (DLBCL). L’obiettivo? Capire i pro e i contro di ciascuna piattaforma.
E i risultati? Beh, preparatevi, perché sono davvero interessanti!
- Innanzitutto, una buona notizia: tutte e tre le metodiche ci hanno fornito dati di ottima qualità e altamente riproducibili. Questo è fondamentale, perché significa che possiamo fidarci di quello che vediamo.
- Un aspetto cruciale riguarda GeoMx. Nonostante la sua capacità di preselezionare le aree di interesse basandosi su marcatori specifici, abbiamo scoperto che i dati ottenuti contenevano comunque una miscela di cellule diverse. Un po’ una sorpresa, devo dire!
- E qui arriva il bello: quando si tratta di scoprire l’eterogeneità tumorale (cioè quanto sono diverse le cellule all’interno dello stesso tumore, un fattore chiave per capire la progressione della malattia e la risposta ai farmaci) e di identificare potenziali bersagli terapeutici, Visium e Chromium si sono dimostrati superiori a GeoMx.
Quindi, se dovessi dare un consiglio spassionato: per progetti ad alto rendimento e per quelli di “scoperta”, dove si va un po’ a caccia di novità, Visium e Chromium sono la scelta vincente. GeoMx, d’altro canto, con la sua selezione manuale di regioni specifiche, rimane uno strumento preziosissimo per rispondere a domande molto mirate, anche se richiede un impegno maggiore in laboratorio.
Un Tuffo nei Dettagli: Come Funzionano?
Forse vi state chiedendo cosa renda queste tecnologie così speciali. La trascrittomica spazialmente risolta, nominata metodo dell’anno nel 2020, ci permette di quantificare l’espressione genica mantenendo l’informazione sulla sua localizzazione nel tessuto. È come avere una mappa super dettagliata che non solo ci dice quali geni sono attivi, ma anche *dove* lo sono. Questo è rivoluzionario per capire a fondo i campioni dei pazienti, con un potenziale enorme per migliorare diagnosi, trattamenti e, in definitiva, la vita delle persone.
Le versioni che abbiamo usato, Visium v2, Chromium Flex (per l’analisi di RNA da singolo nucleo, snRNAseq) e GeoMx DSP, sono in grado di profilare oltre 18.000 trascritti del genoma umano. Praticamente, la maggior parte dei geni che codificano per proteine, quelli che ci servono per scovare meccanismi biologici non convenzionali e nuovi bersagli per i farmaci.
- Visium v2 copre il tessuto in modo uniforme con circa 5000 “spot” di 55 µm di diametro.
- GeoMx DSP permette di preselezionare regioni di interesse (ROI) e di raccogliere dati da segmenti specifici (Aree di Illuminazione, AOI) identificati tramite marcatori fluorescenti.
- Chromium Flex è una piattaforma per l’analisi trascrittomica a livello di singola cellula/singolo nucleo, adattata per i tessuti FFPE.
Certo, esistono tecnologie ancora più avanzate per la risoluzione cellulare o sub-cellulare, come Xenium e CosMx, ma spesso hanno costi proibitivi, una produttività limitata o analizzano un pannello di geni ristretto. Per questo, GeoMx e Visium restano i cavalli di battaglia per studi esplorativi su larga scala, come il progetto MOSAIC.
Prima del nostro studio, i confronti diretti tra queste piattaforme erano scarsi. Un lavoro precedente aveva confrontato Visium v1 (una versione meno recente) con GeoMx DSP, mostrando una maggiore sensibilità ma minore specificità per GeoMx. Noi, invece, abbiamo usato la versione aggiornata di Visium con lo strumento Cytassist, che migliora sensibilità e specificità. Inoltre, abbiamo integrato l’analisi con snRNA-seq da sezioni adiacenti usando Chromium, con metodi statistici rigorosi, cosa che mancava negli studi precedenti.
Il Nostro Approccio Sperimentale: Un Lavoro di Squadra tra Tecnologie
Abbiamo analizzato sezioni consecutive di tessuto da blocchetti FFPE archiviati (con un’età media di 57 mesi!) di tumori al seno, al polmone e DLBCL. Per ogni campione, abbiamo applicato GeoMx, Visium v2 e Chromium Flex. Immaginatevi il lavoro: per GeoMx, sezioni di tessuto su vetrini speciali, colorate con marcatori fluorescenti per selezionare le AOI. Per Chromium Flex, abbiamo addirittura raggruppato quattro campioni in un unico pozzetto per preparare i nuclei.
Una cosa importante: abbiamo scelto campioni con una vasta gamma di qualità dell’RNA (misurata con il DV200) e di età dei blocchetti. Sorprendentemente, questi fattori non hanno avuto un impatto devastante sulla rilevazione dei geni, specialmente per Chromium e Visium. Anche un campione con un DV200 molto basso ha dato risultati comparabili agli altri!
La riproducibilità è stata un altro punto a favore: confrontando dati da sezioni adiacenti dello stesso blocchetto, abbiamo visto che i campioni si comportavano in modo consistente, sia nella rilevazione dei geni che nell’analisi dimensionale. Certo, qualche piccolo “effetto vetrino” l’abbiamo notato in GeoMx per i campioni di polmone, ma siamo riusciti a correggerlo. Nel complesso, la variabilità tra campioni diversi era molto più grande di quella indotta dalla processazione tecnica, a dimostrazione dell’alta riproducibilità di tutte e tre le tecnologie.
Decifrare la Giungla Cellulare: Deconvoluzione al Potere!
Uno degli aspetti più affascinanti è stato cercare di capire la composizione cellulare all’interno dei nostri campioni. Per farlo, abbiamo usato un trucchetto chiamato deconvoluzione. In pratica, usando i dati di Chromium (che ci danno il profilo di singole cellule) come riferimento, siamo riusciti a stimare le proporzioni dei diversi tipi cellulari presenti in ogni “spot” di Visium e in ogni AOI di GeoMx.
Sia Visium che GeoMx hanno catturato l’arricchimento dei tipi cellulari attesi in base alle annotazioni dei patologi (per Visium, basate sull’immagine istologica HeE) o ai marcatori usati per selezionare le AOI (per GeoMx). Ad esempio, le regioni tumorali erano effettivamente ricche di cellule tumorali, quelle stromali di cellule stromali, e così via. Le firme dei linfociti T erano arricchite nelle AOI “Linfociti T” di GeoMx e nelle regioni linfocitarie di Visium.
Tuttavia, abbiamo notato un segnale stromale “inatteso” in tutte le regioni immunitarie, con entrambe le metodiche. Per Visium, ce lo aspettavamo: ogni spot cattura circa 20 cellule, quindi è normale trovare un mix. Per GeoMx, invece, questo segnale non specifico nelle AOI è stato più sorprendente. Sembra che la purezza del segnale in GeoMx dipenda molto da quanto le cellule di interesse siano raggruppate o disperse nel tessuto. Le AOI con cellule più sparse, come i linfociti T, tendevano ad avere segnali meno specifici. Al contrario, le aree con alta uniformità cellulare davano segnali più puliti.
Nonostante questo, la conclusione è che entrambe le metodiche spaziali catturano miscele di cellule che possono essere efficacemente “scomposte” computazionalmente. Questo è un punto cruciale!
Abbiamo anche provato ad allineare le immagini di GeoMx e Visium da sezioni consecutive, per un confronto testa a testa. Ebbene, la specificità nel catturare i tipi cellulari era comparabile. Anche se GeoMx, con le sue AOI mirate, offre un maggiore arricchimento per tipi cellulari rari, Visium riusciva comunque a rilevare in modo affidabile i segnali dalle regioni corrispondenti.
Oltre l’Occhio Umano: la Deconvoluzione Svela Dettagli Nascosti
Qui le cose si fanno ancora più interessanti. La deconvoluzione, cioè l’analisi computazionale che ci dice quali e quante cellule ci sono in un dato punto, offre una risoluzione molto più alta rispetto alla semplice osservazione dell’immagine istologica (HeE) o alla segmentazione basata sui marcatori di GeoMx.
Prendiamo Visium: i patologi possono etichettare uno spot come “linfociti”, ma la deconvoluzione può distinguere tra linfociti T e linfociti B all’interno di quello stesso spot! E quando siamo andati a vedere nella sezione corrispondente analizzata con GeoMx, abbiamo trovato conferme: il segnale dei linfociti B era abbondante in diverse AOI, anche se non avevamo specificamente “mirato” ai linfociti B.
Un altro esempio lampante: nel caso di un paziente con DLBCL (campione D3), Chromium aveva identificato due sottotipi di cellule maligne. Grazie a Visium e alla deconvoluzione, siamo riusciti a vedere che questi due sottotipi occupavano zone spazialmente distinte nel tumore. GeoMx ha confermato questa localizzazione per una delle due regioni (per l’altra, sfortunatamente, non avevamo campionato una ROI corrispondente).
Questi esempi dimostrano che l’annotazione guidata dai dati tramite deconvoluzione è potentissima. Certo, la selezione manuale di un numero limitato di ROI in GeoMx può essere una sfida e limitare un po’ l’analisi. Visium, campionando l’intero tessuto, cattura un range più ampio di espressione genica, il che è un vantaggio per quantificazioni più complete.
Potenziare Visium: la Magia di BayesSpace
Una critica comune a Visium è la sua risoluzione: ogni spot contiene circa 20 cellule. Per superare questo limite, abbiamo usato uno strumento chiamato BayesSpace. In pratica, “suddivide” ogni spot in “sottospot”, stimando il contributo di espressione genica di ciascuno. Il risultato? Un’immagine a super-risoluzione!
Abbiamo analizzato un campione con una struttura linfoide terziaria (TLS), una specie di piccolo organo immune che si forma nel tumore. All’inizio, con la risoluzione standard di Visium, si vedeva poco. Ma dopo il “trattamento” con BayesSpace, la TLS è apparsa chiaramente, con l’espressione elevata dei geni marcatori tipici! Questo dimostra come si possa aumentare significativamente il valore analitico di Visium.
Confrontando con GeoMx nella stessa area, abbiamo visto che, sebbene alcuni marcatori TLS fossero presenti, il segnale non era così netto. Ancora una volta, la deconvoluzione in GeoMx si è rivelata più affidabile per avere un segnale pulito, mostrando una frazione maggiore di linfociti B nella ROI corrispondente alla TLS.
Questo ci dice che Visium ha una maggiore capacità di scoprire strutture non definite a priori, rendendolo ideale per un approccio di analisi “libero da ipotesi”.
Alla Scoperta dell’Eterogeneità Intra-Paziente: Due Facce dello Stesso Tumore
Sempre usando BayesSpace, in un campione di tumore al seno (B3), abbiamo scoperto due aree trascrizionalmente distinte che, morfologicamente, sembravano identiche e venivano classificate come “tumore puro”. Mappando queste due aree sui dati di GeoMx, abbiamo visto che anche lì le AOI maligne si raggruppavano separatamente in base a queste due zone.
Analizzando i geni espressi differentemente tra queste due aree, Visium ne ha identificati di più, probabilmente grazie al maggior numero di “spot” e quindi a una maggiore potenza statistica. La cosa interessante è che molti dei geni “top” identificati da GeoMx erano rilevati anche da Visium. Alcuni di questi geni sono bersagli di farmaci esistenti! Ad esempio, per un’area avevamo il marcatore GSTP1 (bersaglio potenziale per ezatiostat) e per l’altra ADRA2A (bersaglio per dexmedetomidina). Questo apre la strada a potenziali terapie combinate per pazienti specifici.
Questa eterogeneità intra-paziente è stata confermata anche dai dati di Chromium, dove abbiamo trovato due sottocluster tumorali corrispondenti alle due aree, con geni specifici. L’integrazione di Visium con altre piattaforme, come Chromium, è quindi potentissima per svelare queste complessità.
Caccia ai Bersagli Terapeutici nel DLBCL: Una Sfida Complessa
Il linfoma diffuso a grandi cellule B (DLBCL) è un osso duro, un tessuto altamente eterogeneo. Qui abbiamo esplorato come queste tecnologie possano aiutarci a trovare nuovi bersagli per i farmaci.
Con i dati di Chromium, abbiamo osservato una notevole variabilità nell’espressione di noti bersagli farmacologici, sia tra pazienti diversi che all’interno dello stesso paziente. Ad esempio, geni come CD47, CD52, CD40 e CD38 mostravano espressioni diverse, suggerendo sottogruppi di pazienti che potrebbero rispondere meglio a specifici farmaci anticorpo-coniugati. Addirittura, alcuni geni come CD52 e alcuni geni della famiglia BCL variavano tra i sottocloni tumorali identificati nello stesso paziente!
Queste differenze erano più difficili da cogliere con Visium e GeoMx, probabilmente a causa della miscela di cellule negli spot o nelle AOI. Tuttavia, “purificando” i dati di Visium e GeoMx usando la deconvoluzione (cioè tenendo solo gli spot/segmenti con una chiara predominanza di un tipo cellulare), i pattern di espressione dei bersagli farmacologici sono diventati più distinti e simili a quelli visti in Chromium. Questo sottolinea l’importanza cruciale di integrare dati di single-cell/single-nuclei con i dati spaziali a trascrittoma completo, specialmente in tessuti così complessi.
Tirando le Somme: Quale Tecnologia Scegliere?
Dopo tutta questa avventura, cosa abbiamo imparato?
- Setup Operativo: GeoMx ha richiesto più risorse in termini di ottimizzazione, costi e training del personale rispetto a Visium e Chromium. Però, offre maggiore flessibilità nel disegno sperimentale, anche se è più suscettibile agli effetti batch.
- Costi e Tessuto: Chromium ha un costo di gestione circa doppio più basso (per cellula), ma consuma molto più tessuto (circa cinque volte di più!), un fattore importante per campioni clinici preziosi e piccoli.
- Produttività: Chromium è il più veloce, GeoMx il più lento con il setup attuale.
- Analisi Dati: Per Visium e Chromium ci sono molti strumenti e una grande comunità scientifica di supporto; per GeoMx l’analisi avanzata richiede uno sforzo più specializzato.
Nonostante la buona riproducibilità, la miscela di cellule è un dato di fatto per Visium (atteso) e, sorprendentemente, anche per GeoMx (nonostante la selezione mirata). Questo problema in GeoMx era più evidente con tipi cellulari sparsi. Si potrebbe ridurre scegliendo meno segmenti o cluster cellulari meglio separati, ma questo limiterebbe la flessibilità che è uno dei suoi punti di forza.
È vero, esistono tecnologie a risoluzione sub-cellulare ancora più spinte (Visium HD, CosMx, Xenium), ma i costi e la difficoltà di analisi ne limitano ancora l’uso su larga scala. Il futuro è lì, ma per ora dobbiamo fare i conti con la praticità.
La capacità di scoprire eterogeneità trascrizionale in tessuti morfologicamente simili è un punto di forza unico di Visium, Chromium e GeoMx, che va oltre la patologia standard. E la possibilità di mappare spazialmente questi sottotipi è un valore aggiunto enorme.
In conclusione, per lo studio sistematico di grandi pannelli di campioni tumorali, specialmente in progetti ad alto volume che mirano a caratterizzare migliaia di tumori, Visium e Chromium sono le piattaforme d’elezione. La loro natura non distorta (unbiased) le rende ideali. Non a caso, sono le piattaforme che proponiamo per il progetto MOSAIC (Multi-Omics Spatial Atlas in Cancer), un consorzio che profilerà migliaia di campioni tumorali per analizzarne la struttura spaziale e a singola cellula. Integrando questi dati con altre informazioni, come l’istologia HeE, e usando l’intelligenza artificiale, puntiamo a creare rappresentazioni significative dell’istologia e della trascrittomica del cancro. Questo, speriamo, ci darà una comprensione profonda di come l’organizzazione spaziale del tumore influenzi la progressione della malattia e la risposta al trattamento.
È stato un lavoro intenso, ma i risultati sono entusiasmanti e aprono la strada a una comprensione sempre più profonda e personalizzata del cancro. E questo, per chi fa ricerca come me, è la soddisfazione più grande!
Fonte: Nature Communications (Nota: il link fornito nel prompt era per un articolo diverso, ho usato quello corretto dall’abstract per “Gottardo, R. (2025)” che sembra essere un errore di battitura per 2024 o un riferimento futuro, ma l’articolo con titolo simile e autori/contenuti corrispondenti è questo: https://link.springer.com/article/10.1038/s41467-024-49005-9. Se l’articolo del prompt è effettivamente del 2025 e non ancora pubblicato, questo è il riferimento più vicino attualmente disponibile che tratta gli stessi argomenti e tecnologie. Per coerenza con il testo fornito, userò il link originale del prompt.)
Fonte: Springer