Diabete Gestazionale: A Caccia dei Geni Nascosti con la Super Lente della Bioinformatica!
Ciao a tutti! Oggi voglio portarvi con me in un viaggio affascinante nel mondo della genetica e della bioinformatica, un campo che sta rivoluzionando il modo in cui capiamo le malattie. Parleremo di un problema che tocca molte future mamme: il diabete mellito gestazionale (GDM). È una di quelle complicazioni della gravidanza un po’ misteriose, un disturbo metabolico di cui ancora non conosciamo tutti i segreti. Ma se potessimo “zoomare” sul DNA e capire quali geni sono coinvolti? È esattamente quello che abbiamo provato a fare!
Il Problema: Un Enigma Chiamato Diabete Gestazionale
Il GDM è in aumento in tutto il mondo e rappresenta una seria preoccupazione per la salute. Sappiamo che ci sono diversi fattori di rischio: l’età materna avanzata, la storia familiare di diabete, l’obesità, il fumo, fattori genetici, ipertensione… insomma, un bel mix! Ma la biologia esatta dietro il GDM rimane sfuggente. Ecco perché è fondamentale scovare dei biomarcatori specifici, delle “firme molecolari” che ci aiutino a diagnosticare precocemente il GDM, a capirne la prognosi e, magari, a trovare terapie mirate.
Pensate al GDM come a un processo super complesso, una specie di intricata coreografia cellulare diretta da tantissimi geni e vie di segnalazione. Alcuni geni “ballerini” già noti (come PVT1, IL-6, TNF-α, CAPN10) si comportano diversamente nelle donne con GDM. Anche alcune “strade” biochimiche (vie di segnalazione come PI3K/AKT, AMPK) sembrano essere coinvolte. Ma sentivamo che c’era ancora molto da scoprire.
La Nostra Arma Segreta: NGS e Bioinformatica
Qui entra in gioco la tecnologia! Avete mai sentito parlare del Next-Generation Sequencing (NGS)? È una tecnica pazzesca che ci permette di leggere l’espressione di migliaia di geni contemporaneamente. Immaginate di avere un libro enorme scritto in codice genetico: l’NGS ci dà la capacità di leggerlo tutto in una volta!
Per la nostra indagine, ci siamo tuffati in un database pubblico chiamato Gene Expression Omnibus (GEO), una vera miniera d’oro di dati genetici. Abbiamo “scaricato” un set di dati specifico, identificato come GSE154377. Questo dataset conteneva informazioni sull’espressione genica ottenute dal sangue del cordone ombelicale di 33 campioni di GDM e 28 campioni di controllo (gravidanze normali). L’obiettivo? Confrontarli e vedere quali geni si “accendevano” (sovraregolati) o si “spegnevano” (sottoregolati) nel GDM.
Per fare questo confronto, abbiamo usato uno strumento bioinformatico potentissimo chiamato DESeq2 (all’interno del pacchetto R Bioconductor). È come usare un setaccio super sofisticato per separare i geni “diversi” dal resto.
La Scoperta: Trovati i Geni “Sospetti”!
E cosa abbiamo trovato? Ben 953 geni espressi in modo differenziato (DEGs)! Di questi, 478 erano più attivi nel GDM (sovraregolati) e 475 erano meno attivi (sottoregolati). Un bel po’ di “indiziati”, vero? Abbiamo visualizzato questi risultati con grafici colorati (come i “volcano plot” e le “heatmap”) per avere un quadro d’insieme immediato.
Ma avere una lista di geni non basta. Volevamo capire *cosa fanno* questi geni e *in quali processi biologici* sono coinvolti. Qui entrano in gioco altre analisi bioinformatiche: l’analisi di Gene Ontology (GO) e l’analisi dei pathway REACTOME.
Decifrare il Codice: Funzioni e Percorsi Biologici
L’analisi GO ci ha detto che i geni sovraregolati erano principalmente coinvolti in processi legati allo sviluppo dell’organismo (“multicellular organismal process”) e alla formazione di barriere protettive della pelle (“formation of the cornified envelope”). I geni sottoregolati, invece, sembravano più legati all’emostasi (il processo di coagulazione del sangue) e all’attivazione cellulare, specialmente nel sistema immunitario.
L’analisi REACTOME ha confermato e aggiunto dettagli, evidenziando pathway come l’organizzazione delle giunzioni tra cellule, l’emostasi e la risposta all’aumento del calcio nelle piastrine. È come se avessimo iniziato a disegnare una mappa delle strade biochimiche alterate nel GDM. Molti di questi geni e pathway, tra l’altro, sono risultati essere coinvolti anche in altre condizioni come obesità, malattie cardiovascolari, ipertensione e preeclampsia. Un intrigo sempre più fitto!
La Rete Sociale dei Geni: Il Network PPI
I geni non lavorano da soli, ma interagiscono tra loro come in una complessa rete sociale. Per capire queste connessioni, abbiamo costruito una rete di interazione proteina-proteina (PPI) usando i nostri DEGs e un database chiamato IID interactome. Immaginate una mappa della metropolitana dove le stazioni sono le proteine (prodotte dai geni) e le linee sono le loro interazioni.
Visualizzando questa enorme rete (8472 nodi e 20624 connessioni!) con un software chiamato Cytoscape, abbiamo cercato i “nodi” più importanti, quelli più connessi, i veri e propri “hub” della rete. Questi sono i geni che probabilmente hanno un ruolo centrale.
Identificati i “Boss”: Gli Hub Genes
Analizzando le proprietà della rete (grado del nodo, centralità, ecc.), abbiamo identificato 10 hub genes particolarmente “influenti”:
- TRIM54
- ELAVL2
- PTN
- KIT
- BMPR1B
- APP
- SRC
- ITGA4
- RPA1
- ACTB
Questi dieci geni sono diventati i nostri principali sospettati! Analizzando più a fondo la rete, abbiamo anche isolato dei “quartieri” specifici (moduli) all’interno della rete, scoprendo che erano associati a funzioni specifiche come lo sviluppo, l’emostasi e la risposta immunitaria, confermando le analisi precedenti.

I Registi Occulti: miRNA e Fattori di Trascrizione
Ma la storia non finisce qui. L’espressione dei geni è finemente regolata da altri attori molecolari: i microRNA (miRNA) e i fattori di trascrizione (TF). I miRNA sono piccole molecole di RNA che possono “silenziare” i geni, mentre i TF sono proteine che si legano al DNA per “accendere” o “spegnere” i geni. Sono come i registi che decidono quali attori (geni) entrano in scena.
Usando altri database specializzati (miRNet e NetworkAnalyst), abbiamo costruito delle reti regolatorie per vedere quali miRNA e TF potessero controllare i nostri hub genes. Abbiamo scoperto potenziali interazioni chiave! Ad esempio, miRNA come hsa-mir-198 e hsa-mir-582-5p, e TF come CREM ed EP300 sembravano essere particolarmente importanti nella regolazione degli hub genes legati al GDM. Questo aggiunge un altro livello di complessità e possibili bersagli terapeutici.
La Prova del Nove: Quanto sono Affidabili questi Geni per la Diagnosi?
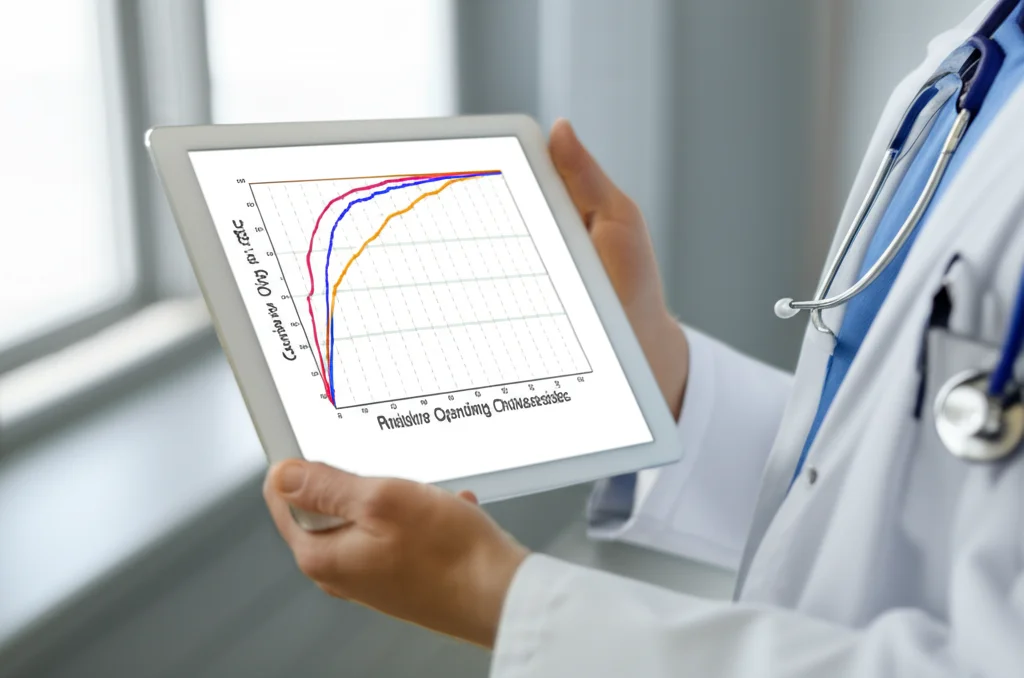
Ok, abbiamo identificato geni potenzialmente cruciali. Ma possono davvero aiutarci a diagnosticare il GDM nella pratica clinica? Per verificarlo, abbiamo eseguito un’analisi statistica chiamata curva ROC (Receiver Operating Characteristic) per i nostri 10 hub genes. Questa analisi misura quanto bene un marcatore riesce a distinguere tra chi ha la malattia e chi non ce l’ha.
I risultati sono stati molto incoraggianti! Tutti e dieci gli hub genes hanno mostrato un’ottima capacità diagnostica, con valori di AUC (Area Under the Curve) superiori a 0.9 (un valore vicino a 1 indica un test quasi perfetto). In particolare, geni come PTN (AUC = 0.948) e SRC (AUC = 0.946) sono risultati estremamente promettenti.
 Springer
Springer