ADGCN: Viaggio nel Cervello con Reti Neurali a Grafo Migliorate per Svelarne i Segreti!
Amici appassionati di scienza e tecnologia, tenetevi forte! Oggi vi porto con me in un viaggio affascinante nel mondo delle neuroscienze e dell’intelligenza artificiale. Parleremo di come stiamo cercando di “mappare” il nostro cervello in modi sempre più precisi e, credetemi, è una sfida che fa tremare i polsi!
Ma perché è così importante “parcellizzare” la corteccia cerebrale?
Immaginate la corteccia cerebrale, quella parte esterna e super ripiegata del nostro cervello, come una metropoli incredibilmente complessa. È il centro di comando per le nostre capacità cognitive superiori, le emozioni, la percezione… insomma, quasi tutto ciò che ci rende umani! Capire come è organizzata, dividendola in regioni funzionalmente distinte (un po’ come i quartieri di una città), è fondamentale. Questa “parcellizzazione” ci aiuta a:
- Svelare i principi organizzativi del cervello.
- Comprendere i meccanismi funzionali.
- Identificare le basi neurali dei processi cognitivi ed emotivi.
- Studiare come la corteccia cambia durante lo sviluppo, l’invecchiamento o a causa di malattie.
- Migliorare la diagnosi di patologie neurologiche e valutare l’efficacia dei trattamenti.
Il problema? La superficie corticale è un labirinto di pieghe e solchi, con enormi variazioni da persona a persona. Analizzarla è come cercare di leggere una mappa accartocciata e scritta in una lingua antica. Mica facile!
I metodi tradizionali: un po’ lenti e macchinosi
Finora, per affrontare questa sfida, ci siamo affidati a metodi che spesso semplificano eccessivamente la geometria del cervello. Pensate alla “espansione sferica”: si prende la corteccia e la si “gonfia” come un palloncino fino a farla diventare una sfera. Questo processo, seppur popolare, richiede ore e ore, e non sfrutta appieno tutte le informazioni strutturali intrinseche. Immaginate di dover appiattire una catena montuosa per studiarla: perdereste un sacco di dettagli, no?
Metodi come FreeSurfer, un classico nel campo, impiegano tempi biblici: 3-4 ore per segmentare un emisfero, più altre ore per la mappatura e la registrazione sferica. Anche alternative più recenti e veloci, come FastSurfer, pur essendo ottime, dipendono molto dalla qualità dei dati iniziali e da passaggi di pre-elaborazione complessi.
Poi ci sono gli approcci basati su atlanti multipli. L’idea è: più mappe di riferimento hai, meglio è. Ma questo significa allineare il cervello del soggetto con ogni singolo atlante, un’operazione computazionalmente intensiva. E se la precisione non è la priorità assoluta, si finisce per usare metodi più semplici basati su atlanti probabilistici, che però introducono distorsioni.
Le tecniche di machine learning tradizionali, come le foreste casuali, sono state usate, ma la vera svolta sembra arrivare dal deep learning.
L’era del Deep Learning e le Sfide con le Reti Convoluzionali (CNN)
Il deep learning, e in particolare le Reti Neurali Convoluzionali (CNN), ha rivoluzionato la segmentazione delle immagini mediche. Le CNN sono fantastiche, ma hanno un “piccolo” problema: sono nate per lavorare su dati strutturati in griglie regolari, come le immagini 2D o 3D (spazio Euclideo). La superficie corticale, invece, è una struttura complessa, ripiegata, che vive in uno spazio non Euclideo. Applicare direttamente le CNN è come cercare di usare un martello per avvitare una vite.
Per far funzionare le CNN sulla corteccia, spesso si ricorre a trasformazioni complesse, come proiettare la superficie su una sfera e poi applicare versioni “sferiche” delle CNN, come la U-Net sferica. Questi metodi sono ingegnosi e hanno portato a buoni risultati, ma c’è sempre quel passaggio di mappatura sferica che richiede tempo e può introdurre distorsioni, specialmente se la superficie originale ha difetti topologici (buchi, triangoli invertiti, ecc.). Inoltre, spesso serve un post-processing per rifinire i risultati.
E qui, amici, entra in scena la nostra proposta: l’ADGCN (Attention-guided Deep Graph Convolutional network). Un nome un po’ altisonante, lo so, ma l’idea di base è potente!
ADGCN: La Nostra Arma Segreta per la Parcellizzazione Corticale
Invece di trasformare la corteccia, perché non lavorarci direttamente nella sua forma originale? È qui che entrano in gioco le Reti Neurali a Grafo (GNN). Le GNN sono perfette per analizzare dati strutturati come grafi, e la superficie corticale (rappresentata come una maglia di vertici e spigoli) è un grafo naturale!
Perché le GNN sono meglio delle CNN per questo compito?
- Struttura non Euclidea: Le GNN gestiscono nativamente la complessità della corteccia, con i suoi numeri variabili di vertici e connessioni.
- Conservazione della Topologia: Operando direttamente sulla maglia corticale, le GNN preservano le informazioni topologiche, evitando le distorsioni delle proiezioni.
- Adattabilità: Si adattano flessibilmente alle variazioni morfologiche tra individui senza richiedere allineamenti rigidi.
Il nostro ADGCN è un tipo speciale di GNN, progettato per una parcellizzazione end-to-end direttamente sulla superficie corticale primitiva. Ecco i suoi “superpoteri”:
- Architettura a “U” Profonda (Deep Graph Convolutional Layer): Ispirata alla celebre U-Net, questa struttura simmetrica permette di trasmettere efficacemente le informazioni dettagliate del grafo cerebrale originale. L’encoder-decoder con connessioni “scorciatoia” (skip connections) cattura sia i dettagli locali che il contesto globale. Questo è cruciale per maneggiare le strutture geometriche complesse e le caratteristiche fini della corteccia.
- Modulo Squeeze-and-Excitation (SE): Questo è il nostro “meccanismo di attenzione”. Permette alla rete di dare più peso alle caratteristiche importanti e di sopprimere quelle meno rilevanti. Immaginatelo come un direttore d’orchestra che dice a certi strumenti di suonare più forte e ad altri più piano, per ottenere l’armonia perfetta. Questo migliora significativamente le prestazioni con un piccolo costo computazionale.
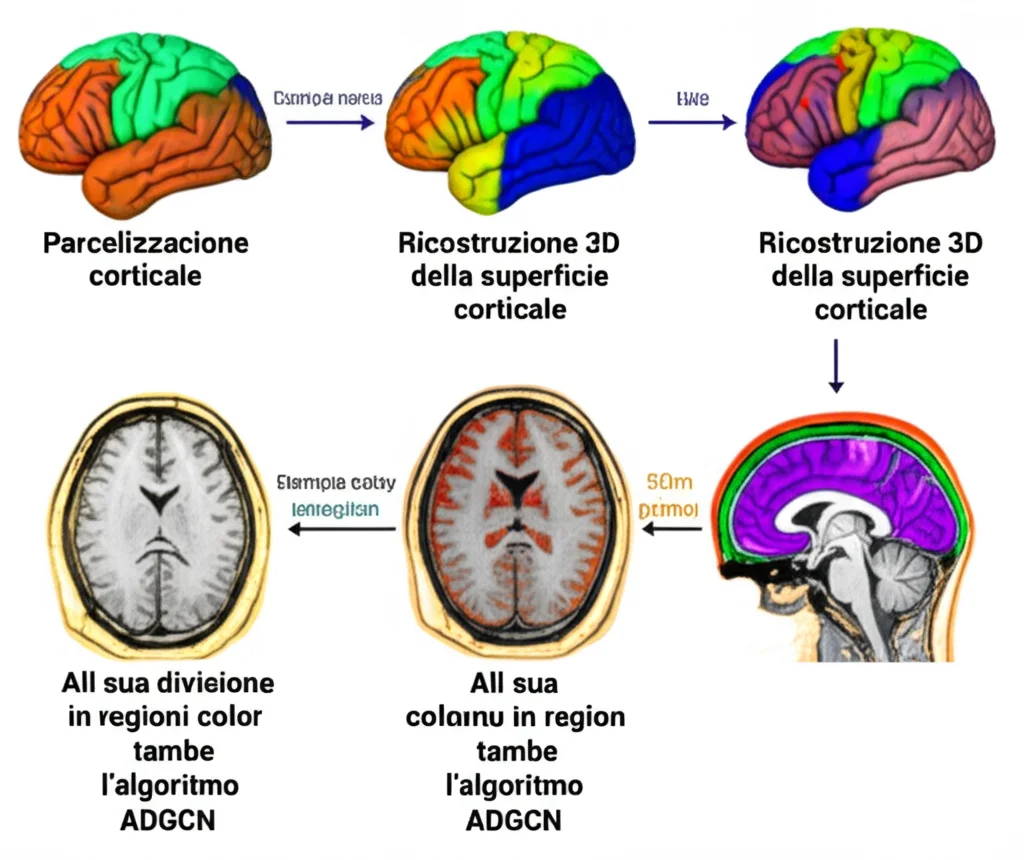
In pratica, prendiamo le immagini di risonanza magnetica strutturale (sMRI), usiamo il software FreeSurfer per pre-elaborarle e ottenere la superficie della materia bianca (WM) e le sue caratteristiche morfologiche. Da qui, costruiamo un “grafo cerebrale” (i vertici della superficie sono i nodi, le connessioni tra loro sono gli spigoli). Questo grafo, insieme alle caratteristiche, viene dato in pasto alla nostra rete ADGCN, che assegna un’etichetta a ogni nodo, ottenendo così la parcellizzazione.
Mettiamolo alla Prova: Il Dataset Mindboggle-101
Per testare l’ADGCN, abbiamo usato il dataset pubblico Mindboggle-101. Contiene 101 cervelli (noi ne abbiamo usati 100 perché uno non aveva le etichette manuali complete) etichettati manualmente secondo il protocollo DKT (31 regioni corticali per emisfero). Queste etichette manuali sono il nostro “gold standard”.
Abbiamo pre-elaborato i dati, estraendo sei caratteristiche morfologiche per ogni vertice della superficie WM: area superficiale, curvatura media, misura Jacobiana, profondità dei solchi, spessore corticale e distorsione della misurazione. Per garantire una risoluzione adeguata e un numero uniforme di vertici, tutte le superfici corticali sono state ricampionate a una configurazione standard (fsaverage5, con 10.242 vertici).
Le metriche di valutazione? Il coefficiente di Dice (misura la sovrapposizione tra predizioni ed etichette reali) e l’accuratezza (percentuale di vertici predetti correttamente). Più alti sono, meglio è!
I Risultati: Cosa Ci Dice l’ADGCN?
Abbiamo condotto esperimenti per vedere l’impatto del modulo SE e del numero di strati nascosti nella rete. Ebbene, aumentare il numero di strati (fino a 7, con una struttura a U simmetrica per le dimensioni delle feature) ha migliorato notevolmente le prestazioni. Ad esempio, con 7 strati nascosti, l’ADGCN ha ottenuto un coefficiente di Dice dell’86.58% e un’accuratezza dell’86.57%, un bel salto rispetto a un modello con un solo strato nascosto! Questo suggerisce che, per grafi cerebrali sparsi come i nostri, una rete più profonda impara meglio le caratteristiche.
L’introduzione del modulo SE (con un rapporto di riduzione r=4, che si è rivelato ottimale) ha ulteriormente affinato i risultati. Combinando 7 strati nascosti e il modulo SE, il nostro ADGCN finale ha raggiunto un coefficiente di Dice dell’88.53% e un’accuratezza del 90.27%. Non male, eh?
Confrontando l’ADGCN con altri metodi, abbiamo notato cose interessanti:
- Metodi su dominio sferico: FreeSurfer ottiene un Dice dell’83.69%. SPHARM-Net arriva all’88.64%. SegRecon all’88.69%. L’U-Net sferica con attention (Attention-gated spherical U-net) raggiunge l’89.90%. Un metodo di Zhao et al. (2023) che sfrutta la consistenza longitudinale arriva addirittura al 92.70% di Dice. Questi metodi sono forti, ma, come dicevamo, la mappatura sferica è un processo complesso, lungo e sensibile a difetti topologici, il che li rende meno adatti a cervelli “danneggiati”.
- Metodi su dominio originale (come il nostro): Una GCN base ottiene l’86.48% di Dice. ASEGAT, un altro approccio basato su GNN con meccanismi di attenzione e vincoli anatomici, raggiunge l’89.00% di Dice e il 90.65% di accuratezza. ASEGAT è molto performante, ma il nostro ADGCN, pur essendo leggermente sotto in termini di Dice rispetto ad ASEGAT, utilizza le informazioni della struttura del grafo in modo più diretto ed efficiente, rendendo l’addestramento più stabile.
In generale, i metodi sul dominio sferico tendono ad avere performance leggermente superiori, forse perché la mappatura su una sfera aiuta a stabilire meglio le corrispondenze tra atlante e individuo. Tuttavia, il nostro ADGCN, lavorando direttamente sul dominio originale, evita i problemi della mappatura sferica, offrendo alta efficienza, semplicità operativa e una forte interpretabilità.
Limiti e Prospettive Future: Non Ci Fermiamo Qui!
Siamo entusiasti dei risultati, ma siamo anche i primi a riconoscere che c’è ancora strada da fare. Ecco alcune aree su cui ci concentreremo:
- Sfruttare meglio le informazioni globali: Attualmente, il ricampionamento a uno spazio standard potrebbe far perdere qualche informazione strutturale. Stiamo pensando a metodi di registrazione non rigida basati su deep learning per minimizzare questa perdita.
- Robustezza alla qualità della ricostruzione: Piccoli errori nella maglia corticale possono dare problemi. Svilupperemo algoritmi di ottimizzazione iterativa che preservino la topologia per correggere questi errori.
- Integrazione di più feature: Per ora usiamo solo caratteristiche morfologiche superficiali. Vogliamo integrare informazioni sulla microstruttura e sulla connettività funzionale, magari combinando dati volumetrici (come la densità della materia grigia) con quelli superficiali.
- Generalizzazione e applicazioni cliniche: Valideremo il nostro metodo su dataset più ampi e diversificati, come l’ABIDE (Autism Brain Imaging Data Exchange), che include soggetti con disturbo dello spettro autistico e controlli sani. Questo ci aiuterà a capire il potenziale diagnostico del nostro ADGCN.

In Conclusione: Un Passo Avanti per Capire il Cervello
Il nostro ADGCN rappresenta, a mio modesto parere, un passo avanti significativo nella parcellizzazione della superficie corticale. Combinando la potenza delle GCN con un’architettura a U profonda e meccanismi di attenzione, siamo riusciti a ottenere segmentazioni accurate e veloci direttamente sulla complessa geometria originale del cervello.
Questo approccio non solo facilita lo studio dei cambiamenti corticali legati allo sviluppo, all’invecchiamento e alle malattie, ma ha il potenziale per migliorare l’accuratezza diagnostica delle patologie neurologiche e l’oggettività nella valutazione dell’efficacia terapeutica. Il viaggio per decodificare il cervello è ancora lungo, ma strumenti come l’ADGCN ci forniscono mappe sempre più dettagliate per navigare in questo straordinario universo.
Spero che questo piccolo assaggio del nostro lavoro vi abbia incuriosito. La ricerca non si ferma mai, e chissà quali altre scoperte ci riserva il futuro!
Fonte: Springer